
 |  |
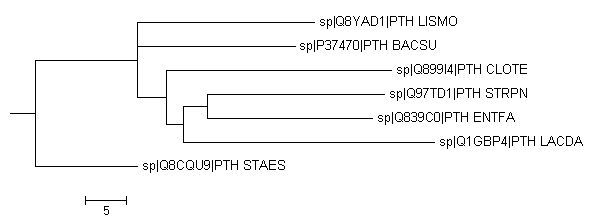
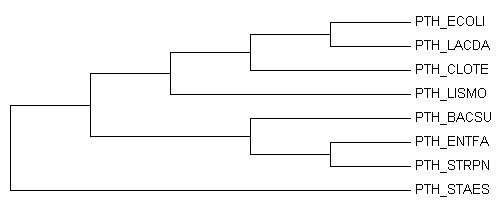
| Рисунок 1. Оригинальное дерево | Рисунок 2. Укорененное в среднюю точку дерево |

 |
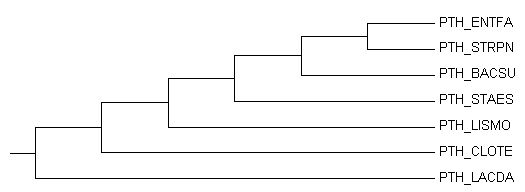 |
| Рисунок 4. Неукорененное дерево с внешней группой. | Рисунок 5. Укорененое дерево с внешней группой (клада Firmicutes) |
 |
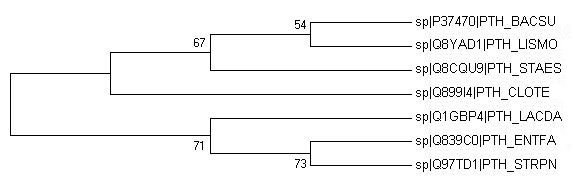 |
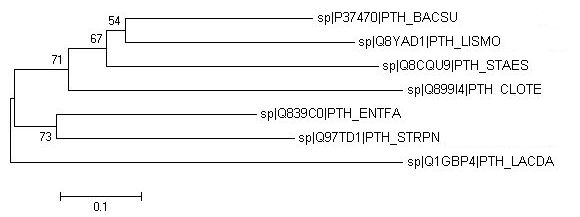
| Рисунок 6. Оригинальное дерево | Рисунок 7. Дерево бутстрэпа |
| Главная страница | Первый семестр | Второй семестр | Третий семестр | Обо мне | Ссылки |