| AC | ID | Функция | Число последовательностей | Число архитектур |
| PF03949 | Malic_M | Связывание НАД | 6923 | 36 |
| Архитектура | Число доменов | Кратко о втором домене | Число последовательностей |
| Malic_M + malic | 2 | PF00390: N-концевой домен малатдекарбоксилаз | 4298 |
| Malic_M + malic(x2) | 3 | PF00390: N-концевой домен малатдекарбоксилаз | 602 |
| Архитектура | Число последовательностей | Proteobacteria | Firmicutes |
| Malic_M + malic | 26 | 15 | 11 |
| Malic_M + malic(x2) | 30 | 15 | 15 |
| Архитектура | Proteobacteria | Firmicutes |
| Malic_M + malic | P2 | F2 |
| Malic_M + malic(x2) | P3 | F3 |
 |
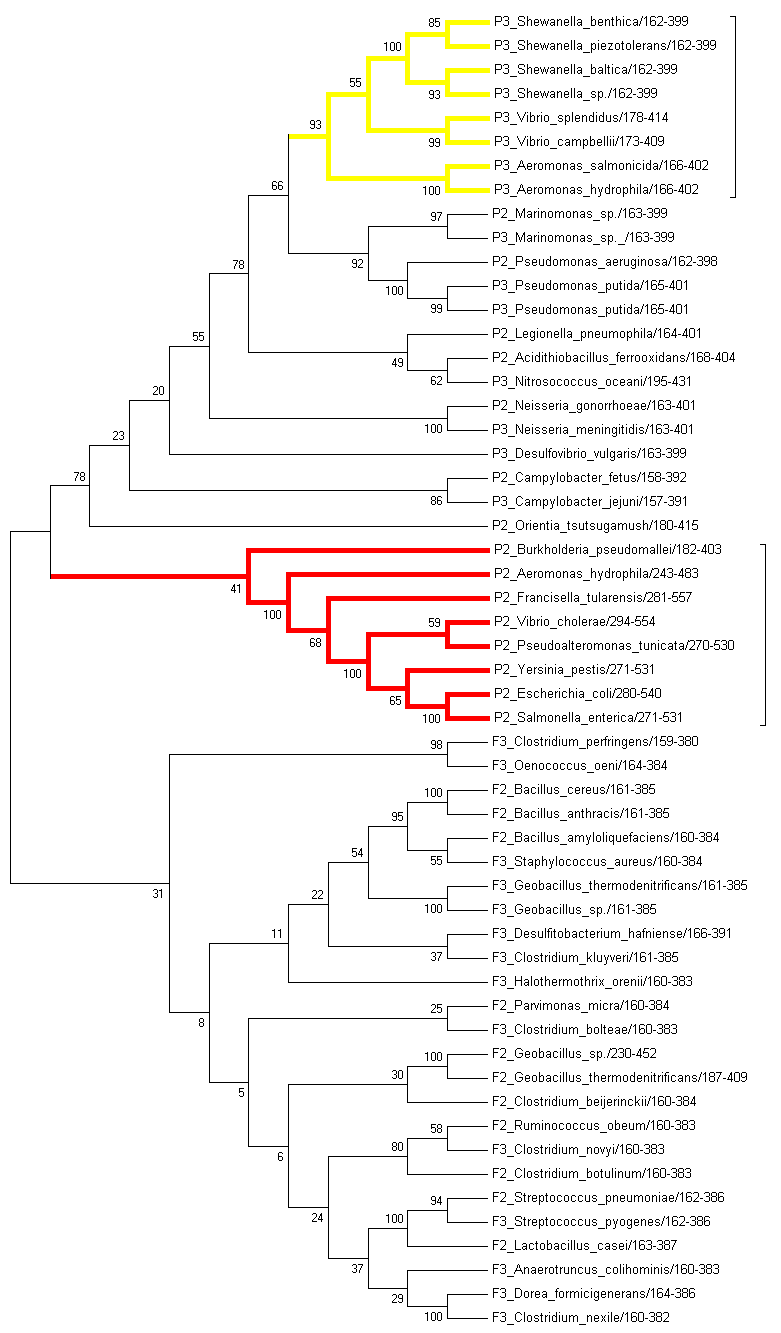
| Рисунок 2. Филогенетическое дерево домена Malic_M, построенное методом maximal likelyhood с использованием бутстрэпа. |
 |
| Рисунок 3. Схематичное изображение эволюции рассматриваемых архитектур в выбранных таксонах. 2 - двухдоменная архитектура, 3 - трехдоменная. |
| Главная страница | Первый семестр | Второй семестр | Третий семестр | Обо мне | Ссылки |