Реконструкция деревьев по нуклеотидным последовательностям. Деревья, содержащие паралоги
Построение дерева по нуклеотидным последовательностям
В предыдущем разделе филогенетическое дерево выбранных вдов строилось по выравниванию белковых последовательностей. Также можно это делать по нуклеотдным последовательностям.
16S рибосомная РНК - достаточно консервативная последовательность, которая часто используется для посторения филогенетических деревьев. Построим филогенетическое дерево выбранных
видов бактерий по последовательностям их 16S рРНК. Их последовательности возьмем из полногеномных записей банка EMBL, полученных командой entret. Для этого используем команду seqret -sask.
| Мнемоника вида |
AC записи в EMBL |
Начало последовательности |
Конец последовательности |
Цепь |
| LACDA |
CR954253 |
45160 |
46720 |
Прямая |
| CLOTE |
AE015927 |
41801 |
43309 |
Обратная |
| BACSU |
D26185 |
73411 |
74963 |
Прямая |
| STAES |
AE015929 |
1722288 |
1723841 |
Обратная |
| LISMO |
AL591974 |
37466 |
39020 |
Прямая |
| ENTFA |
AE016830 |
248466 |
249987 |
Прямая |
| STRPN |
Нет |
1 |
1412 |
Обратная |
У бактерии Streptococcus pneumoniae в записи банка EMBL не оказалось 16S рРНК, поэтому пришлось воспользоваться поиском blastn. Была взята последовательность Enterococcus faecalis и найден ее гомолог у Streptococcus pneumoniae.
Последовательности были выровняны программой MUSCLE. Ссылка на вырванивание.
По выравниванию с помощью программы MEGA было построено филогенетическое дерево. Деревья, построенные тремя способами (maximum likelyhood, neighbour-joining, minimal evolution), оказались одинаковыми.
 | Рисунок 1. Дерево, построенное по послеовательностям 16S рРНК выбранных бакерий. |
Полученное дерево не очень похоже на правильное (в общем я иного и не ожидал).
Построение и анализ дерева, содержащего паралоги
С пмощью программы blastp среди выбранных видов были найдены гомологи бекла CLPX_BACSU. По их выравниванию было построено дерево.
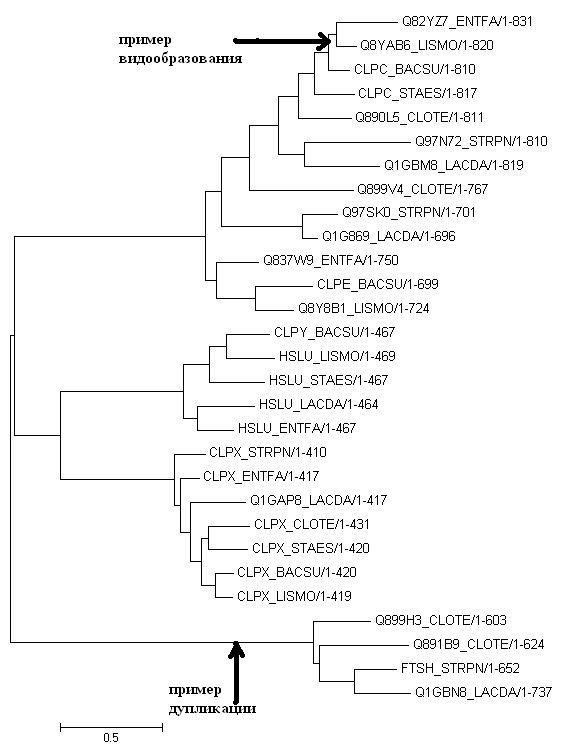 Рисунок 2. Дерево гомологов CLPX_BACSU, показаны пример разделения белков в ходе видообразованияс (появление ортологов) и дупликации белка внутри одного организма (появление паралогов).
Рисунок 2. Дерево гомологов CLPX_BACSU, показаны пример разделения белков в ходе видообразованияс (появление ортологов) и дупликации белка внутри одного организма (появление паралогов).
Пример пары паралогов:
Q899H3_CLOTE и Q891B9_CLOTE
Примеры пар ортологов:
CLPX_BACSU и CLPX_LISMO
HSLU_LACDA и HSLU_ENTFA
© Марк Меерсон, 2014
Последнее обовление: 08.03.2014


 Рисунок 2. Дерево гомологов CLPX_BACSU, показаны пример разделения белков в ходе видообразованияс (появление ортологов) и дупликации белка внутри одного организма (появление паралогов).
Рисунок 2. Дерево гомологов CLPX_BACSU, показаны пример разделения белков в ходе видообразованияс (появление ортологов) и дупликации белка внутри одного организма (появление паралогов).