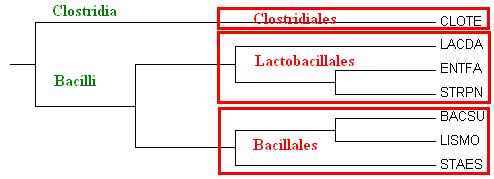
| Название | Мнемоника | Тип | Класс | Отряд | Семейство |
| Clostridium tetani | CLOTE | Firmicutes | Clostridia | Clostridiales | Clostridiaceae |
| Bacillus subtilis | BACSU | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Lactobacillus delbrueckii | LACDA | Firmicutes | Bacilli | Lactobacillales | Lactobacillaceae |
| Enterococcus faecalis | ENTFA | Firmicutes | Bacilli | Lactobacillales | Enterococcaceae |
| Staphylococcus epidermidis | STAES | Firmicutes | Bacilli | Bacillales | Staphylococcaceae |
| Streptococcus pneumoniae | STRPN | Firmicutes | Bacilli | Lactobacillales | Streptococcaceae |
| Listeria monocytogenes | LISMO | Firmicutes | Bacilli | Bacillales | Listeriaceae |

 1
1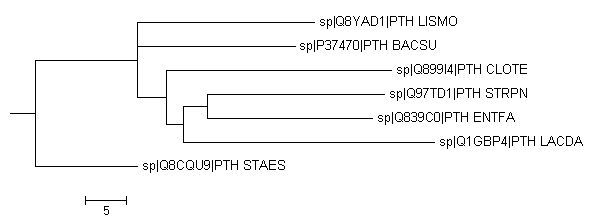 3
3 2
2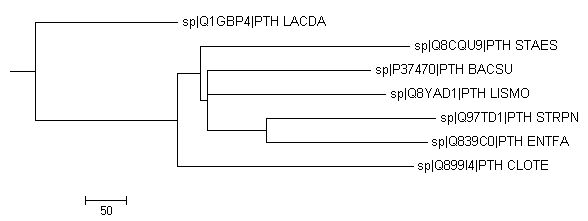 4
4
