Совмещение структур
Поиск и анализ структурных гомологов 3DL0
С помощью PDBeFold были найдены структурные гомологи аденилаткиназы B. subtilis (структура 3DL0).
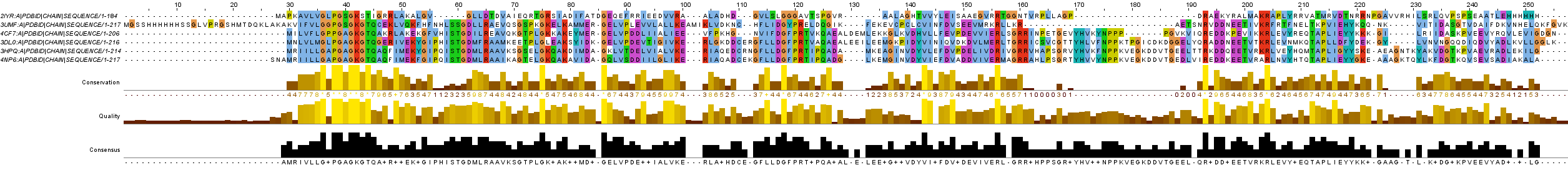
Были отобраны следующие гомологи: 4CF7, 3HPQ, 3UMF, 4NP6, 2IYR. Выранвивание этих структур с 3DL0 покзано на рисунке 1.
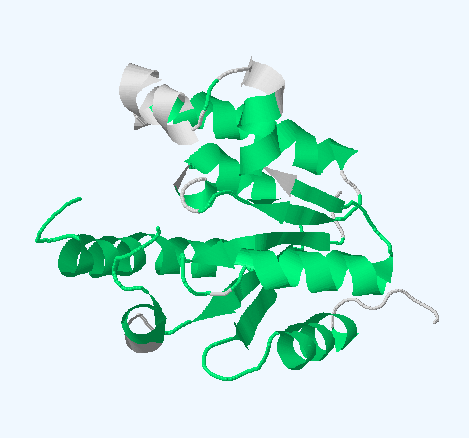
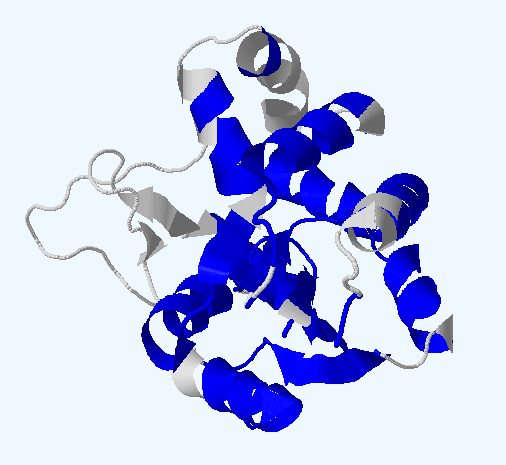
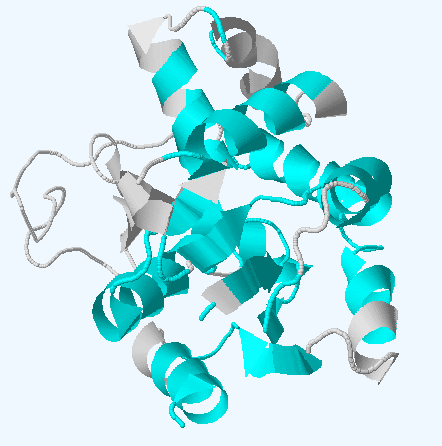
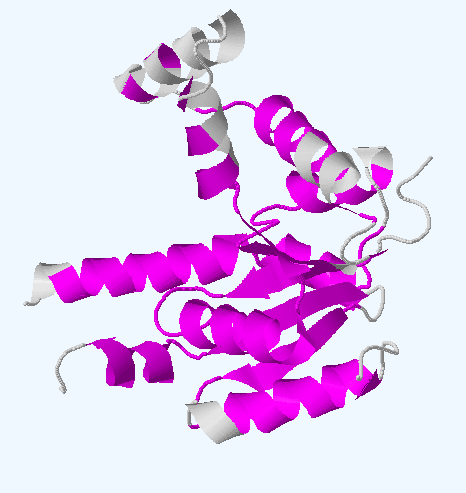
Рисунок 1. Разное представление множественного структурного выравнивания структурных гомологов 3DL0 (сверху) и структуры гомологов по отдельности (снизу).
На рисунке 1 цветами показаны области, которые выровнялись во всех структурах. Видно, что, например, цинк-связывающий домен выровнялся не у всех
(он выделен красным на правом выравнивании).
В то же время собственно каталитический выровнялся по всем структурам.
Сервис PDBeFold также выдаёт выравнивание последовательностей, соотвествующее выравниванию структур (рис. 2).
Можно сравнить это выравнивание, с выравниванием, построенным исключительно по последовательностям, например, программой MUSCLE.
Рисунок 2. Множественное выравнивание, соотвествующее выравниванию структур, полученное PDBeFold (сверху), и выравнивание, построенное программой MUSCLE (снизу).
Выравнивания визуализированы через Jalview, раскраска ClustalX.
Если присмотреться, выравнивания на самом деле достаточно похожи, хотя сразу видно, что выравнивание, построенное MUSCLE, гораздо лучше (особенно, если учесть, что мелкие буквы в верхнем выравнивании означают невыровненные остатки).
Однако в обоих выравниваниях, например, хорошо виден большой пропуск в 2 последовательностях, соотвествующий отсутсвию цинк-связывающего домена (если посмотреть на структуры на рисунке 2, видно, что его нет именно у этих 2 белков - 2IYR и 3UMF).
И вообще, начиная примерно со 140-ого остатка выранвнивания очень похожи. Кроме того у них почти одинаков первый крупный блок.
Глобальное отличие выравниваний - наличие большого невыровненного участка после этого блока в PDBeFold, в то время как MUSCLE этот участок выровнял. Причина здесь может быть в разности выравнивания последовательности 3UMF: из рисунка видно, что она выровнена принципиально по-разному.
Поиск структурных гомологов домена из 1B0M
1B0M - структура аконитазы свиньи. Если вести поиск в PDBeFold по домену A:203-315, результата программа не выдаёт. Однако если поменять праметр % выровненных остатков в запросе (query) на 100%, то программа находит целевую структуру.
Совмещение по заданному выравниванию
Для работы была выбрана структура 1OGA. Нужно было совместить цепь альфа (D 118-202) и цепь бета (E 119-245).
Для начала эти домены были вырезаны из исходной структуры и записаны в отдельные PDB-файлы: альфа-цепь и бета-цепь.
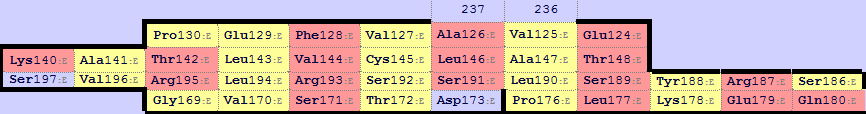
Затем с помощью SheeP были построены карты бета-листов для этих доменов. Нарисунке 3 показаны листы уже переведнные в правильную ориентацию.
Рисунок 3. Карты бета-листов, выданные SheeP: верхний - для альфа-цепи, 2 нижних - для бета-цепи.
В цепи бета, вообще говоря, 2 листа, и оба из них можно поместить в ориентацию, совпадающую с листом из цепи альфа.
Я решил выравнивать тот лист, который больше.
Скрипт для выравнивания
Выровненными a priori считались остатки цистеина внутри листа и соседние с ним остатки из того же гребня, а также 2 соседних гребня (см скрипт).
Таким образом задается поверхность листа.
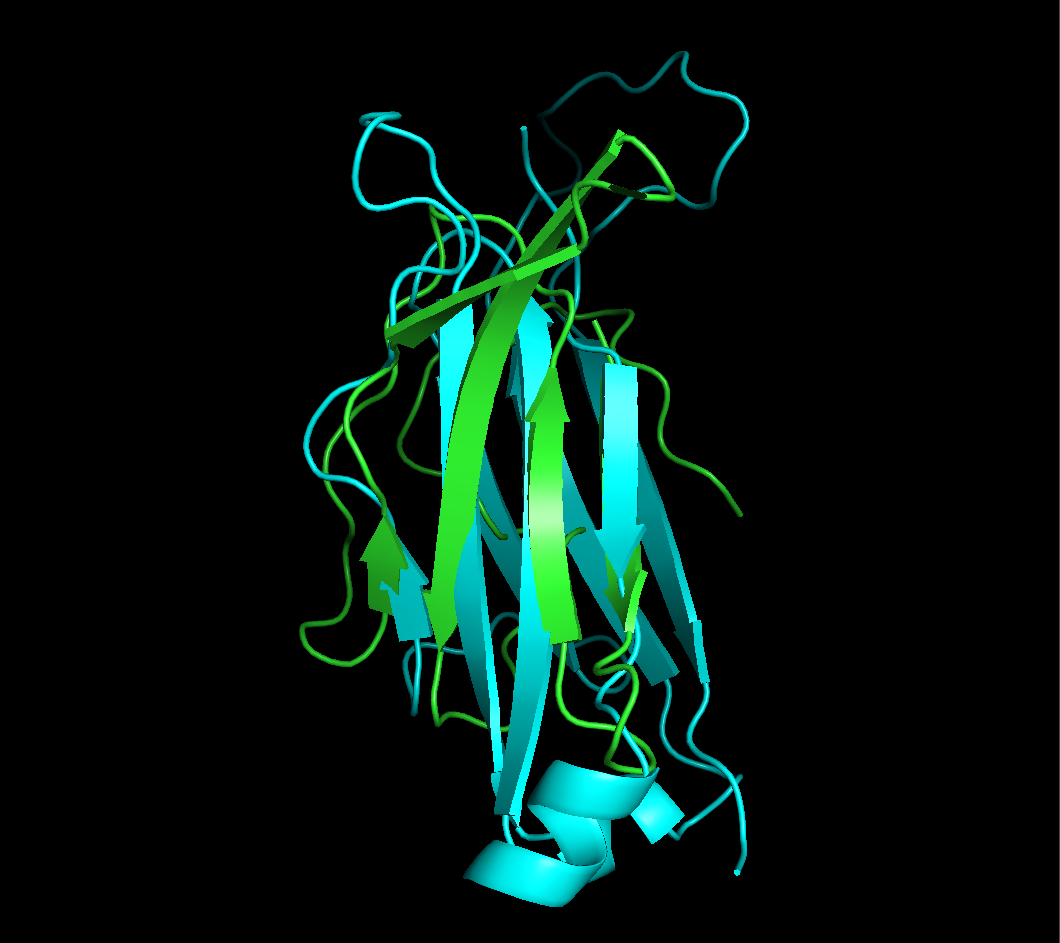
Рисунок 4. Выровненные альфа (зеленая) и бета (голубая) цепи из структуры 1OGA. Красным показаны участки, на основе которых проводилось совмещение.
Видно, что выравнивание не блестящее, один бета тяж из альфа цепи вообще ни с чем не выровнялся.
На всякий случай, сессия PyMOL для выравнивания.
Можно сказать, что у выровненных участков структур достаточно сходные топологии. Видно что выровнены (более или менее) оказались 4 тяжа.
Однако 1 тяж и цепи альфа не имеет соотвествия в цепи бета. Кроме того в цепи бета между двумя тяжами есть альфа-спираль, которой нет в цепи альфа.
Так что топологии совпадают неполностью.
© Марк Меерсон, 2015
Последнее обновление 21.12.2015