Определение вторичной структуры
Stride
Для сравнения аннотациИ вторичной структуры, полученной программой Stride, с данными из PDB использовалась аденилаткиназа B. subtilis (PDBid = 3DL0).
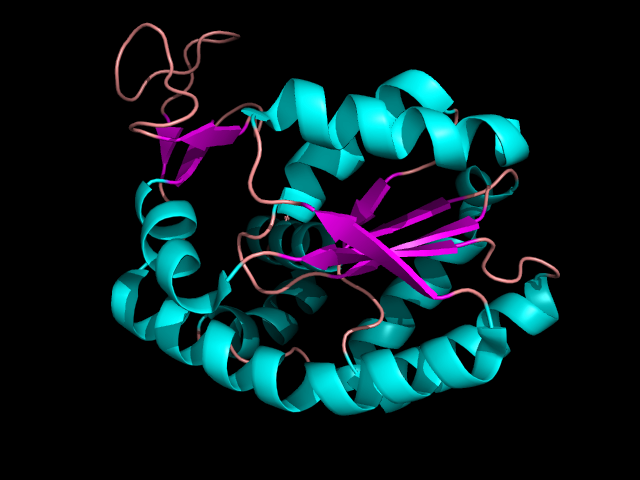
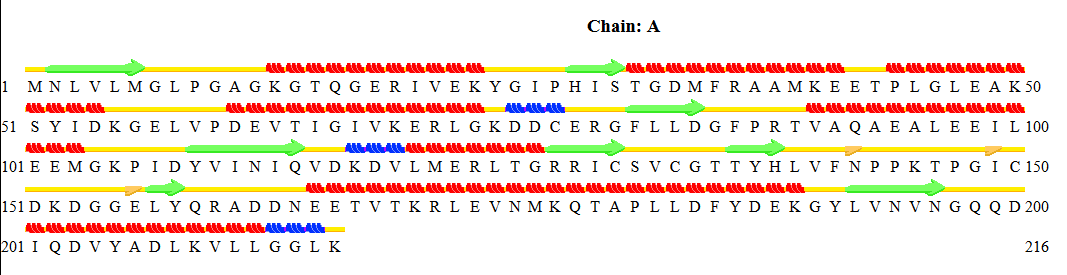
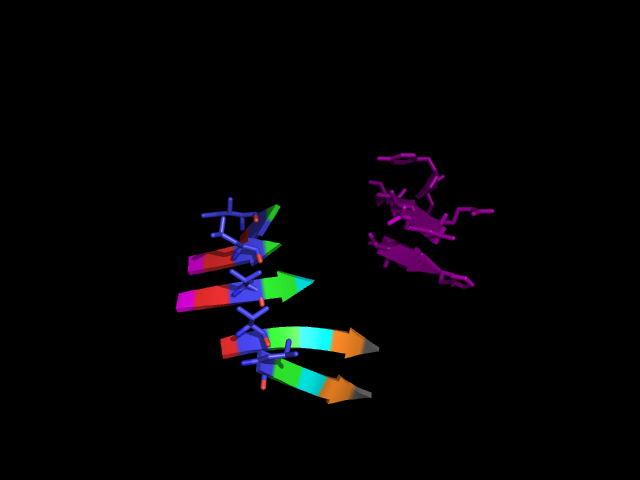
На рисунке 1 показано графическое изображение выдачи программы Stride для цепи А из структуры 3DL0 (в работе использовалась только цепь А, так как цепь B ей идентична).
На том же рисунке показано визуалированная в PyMOL цепь А из 3DL0.
Рисунок 1. Структура аденилаткиназы (слева) и разметка вторичной структуры на той же цепи, полученная через Stride.
Аннтоцию Stride (файл) можно сравнивать с данными о вторичной структуре, записанными в PDB-файле (файл).
Бета-листы Stride нашел все те же, что и в PDB файле, см рисунок 2. Альфа-спирали, показанные Stride несколько отличаются от тех, что записаны в PDB:
- В PDB их 10, а Stride выдал 8
- Те, что выданы Stride, почти все неполностью совпадают со спиралями из PDB
Рисунок 2. Сравнение эелемнтов вторичной структуры PDB (слева) и Stride (справа): альфа-спиралей (сверху) и бета-листов (снизу).
Хорошим примером несоотвествия альфа-спирали из PDB альфа-спирали Stride является спираль 116-118.
В PDB она записана, как альфа-спираль, а Stride определил её, как спираль типа 3-10 (кстати их он обнаружил несколько, см рисунок 1 (выделение синим)).
Стоит сказать, что на структуре эта спираль действительно выглядит довольно напряженной, она выделена на рисунке 3.
Рисунок 3. Альфа-спирали PDB, показана спираль 116-118, проаннотированная Stride как спираль 3-10.
Кроме того, Stride даёт информацию о других элементах вторичной структуры, а именно поворотах. В частности, Stride обнаружил в аденилаткиназе
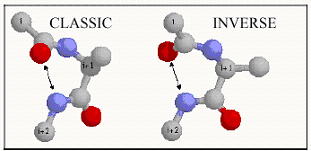
2 инвертированных гамма-поворота (GammaInv). Для этого типа поворота характерно сильное изменение торсионных углов, в следствии чего остаток, образующий поворот становится похож на остаток другой хиральности.
Рисунок 4. Сравнение обычного поворота (слева) и инвертированного (справа).
SheeP
Программа SheeP (с настройками по дефолту) нашла в структуре 3DL0 6 бета-листов (по 3 на цепь). Странно даже не то, в PDB структуре листов явно 2 на цепь, а
то, что листы в цепи А и В в предсказании SheeP отличаются, хотя цепи идентичны. На рисунке 5 представлена карта самого большого бета-листа
из цепи А, полученная SheeP и этот же бета-лист, выделенный из структуры PDB.
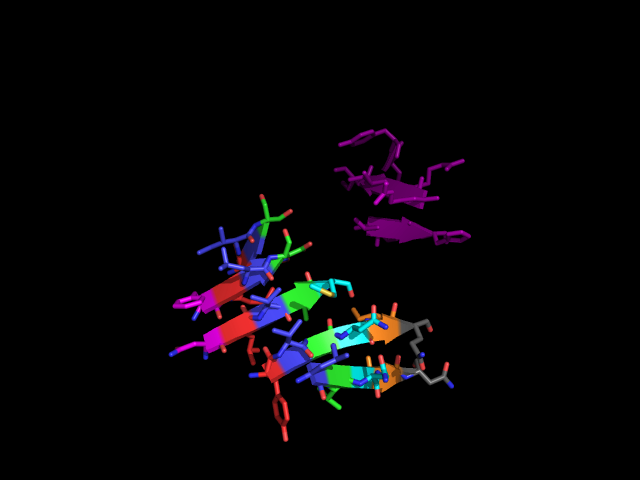
Рисунок 5. Самый крупный бета-лист из цепи А, найденный SheeP. Слева - карта листа (столбцы - гребни, строки - тяжи), справа - разметка, выданная SheeP с помощью Jmol.
Этот лист состоит из 5 тяжей и формирует 7 гребней. На рисунке 6 показаны гребни, выделенные разными цветами в PyMOL, видно, что они соотвествуют гребням, размеченным SheeP (рис. 5).
Рисунок 6. Самый крупный бета-лист из цепи А, найденный SheeP. Разными цветами выделены гребни. Слева показаны все гребни, справа выделен один, соотвествующий тертьей слева колонке на карте листа из рисунка 5.
Понять какие гребни (красные или желтые) образены к гидрофобному ядру сложно, потому что распределение гидрофобных остатков в них примерно одинаковое.
Хотя в выделенном на рисунке 6 гребне пристутствуют исключительно гидрофобные остатки (остальные гребни смешанные), а он красный, так что, пожалуй, всё-таки красные гребни обращены к ядру.
Кроме того с помощью SheeP была сгенерирована карта бета-листов. На ней показаны пептидные (---) и водородные (\ или /) связи в листе.
© Марк Меерсон, 2015
Последнее обновление 19.12.2015