
1.0
2.0
3.0
4.0
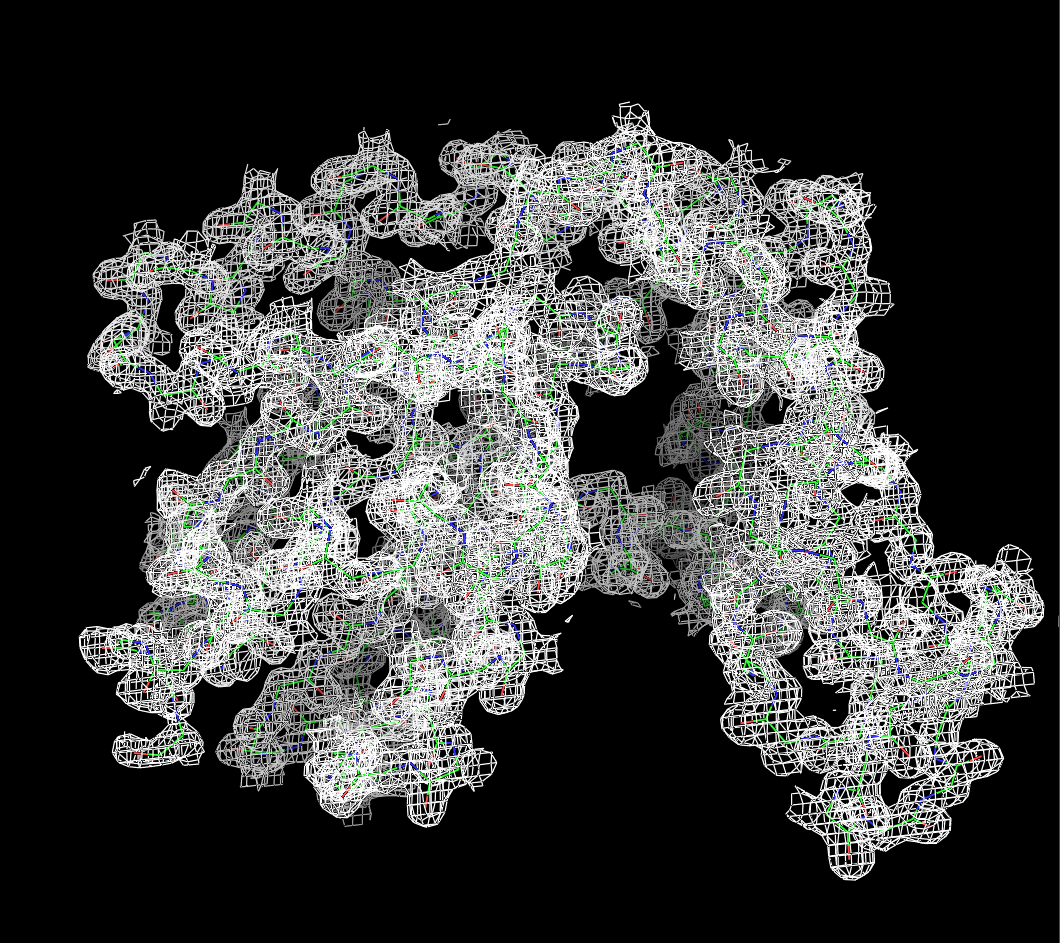
Для работы использовалась 3D структура аденилаткиназы из Bacillus subtilis, белка, который уже изучался в первых семестрах. BDBid стркутруры - 3DL0. PDB-файл содержит 2 цепи длиной 216 аминокислот (гомодимер).
TRANSFERASE 26-JUN-08 3DL0
CRYSTAL STRUCTURE OF ADENYLATE KINASE VARIANT AKLSE3
ORGANISM_SCIENTIFIC: BACILLUS SUBTILIS;
ORGANISM_TAXID: 1423;
EXPRESSION_SYSTEM: ESCHERICHIA COLI;
EXPRESSION_SYSTEM_TAXID: 562;
EXPRESSION_SYSTEM_STRAIN: BL21(DE3);
EXPRESSION_SYSTEM_VECTOR_TYPE: PLASMID;
EXPRESSION_SYSTEM_PLASMID: PET11
PHOSPHOTRANSFERASE, ZINC COORDINATION, ATP-BINDING, KINASE,
BINDING, NUCLEOTIDE BIOSYNTHESIS, NUCLEOTIDE-BINDING, TRANS
R.M.BANNEN,C.M.BIANCHETTI,C.A.BINGMAN,J.G.MCCOY
15-OCT-14 3DL0 1 JRNL
25-JUN-14 3DL0 1 JRNL
24-OCT-12 3DL0 1 VERSN
09-JUN-09 3DL0 0
S.MOON,R.M.BANNEN,T.J.RUTKOSKI,G.N.PHILLIPS,E.BAE
EFFECTIVENESS AND LIMITATIONS OF LOCAL STRUCTURAL E
OPTIMIZATION IN THE THERMAL STABILIZATION OF MESOPH
THERMOPHILIC ADENYLATE KINASES.
PROTEINS V. 82 2631 2014
ISSN 0887-3585
24931334
10.1002/PROT.24627
RESOLUTION. 1.58 ANGSTROMS.
Разрешение данной структуры 1,58 ангстрем, это очень хорошее разрешение, чего и следовадо ждать, для столь хорошо изученного белка. fetch 3dl0
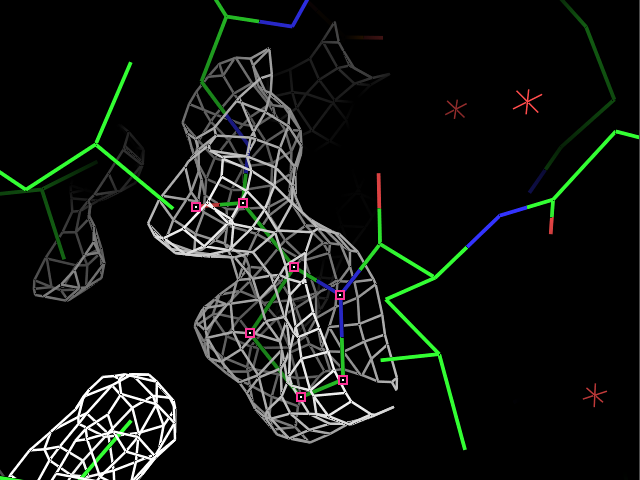
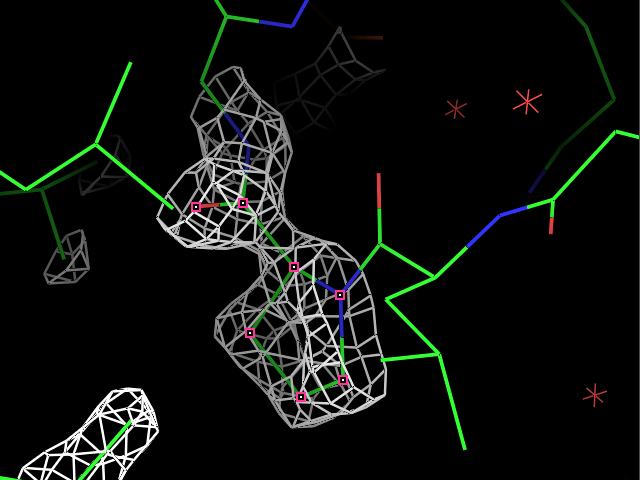
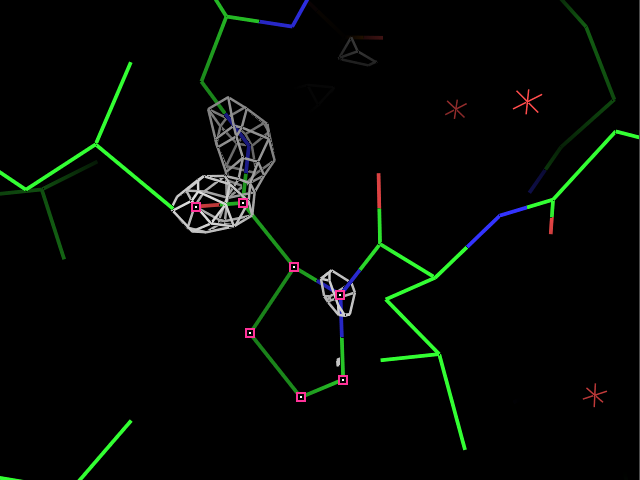
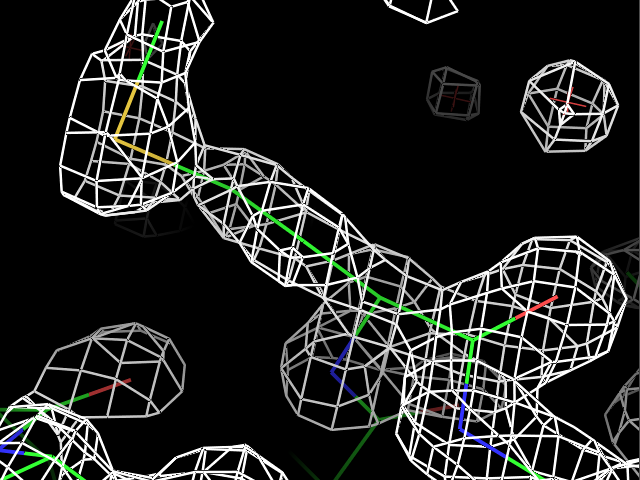
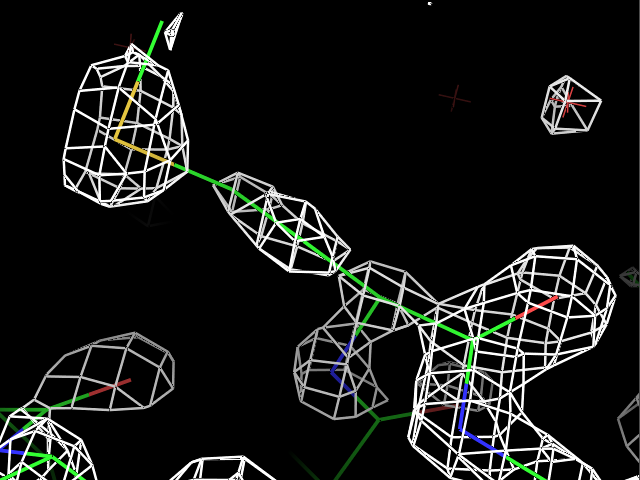
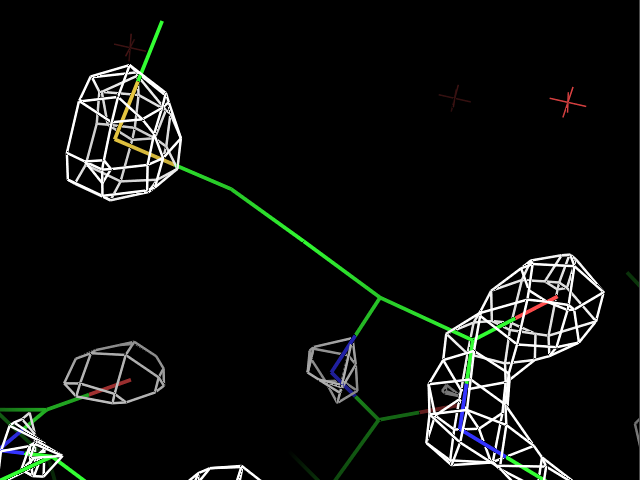
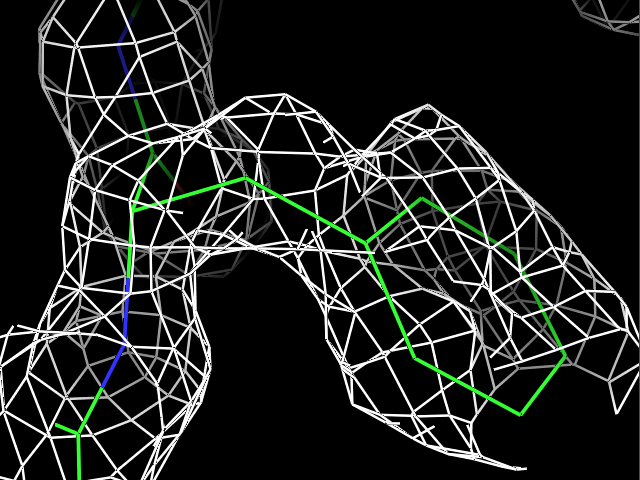
load $PYMOL_PATH/test/dat/3dl0.omap, new_map1Сначала ЭП была визуализирована для трех разных остатков при разных уровнях подрезки ЭП. Пример команды:
isomesh map_pro, new_map1, 2.0, resi 9Результаты для остатков Pro9, Met34 и Phe81 с уровнями подрезки 1, 2, 3, 4 показаны в таблице.
| Pro9 |  |
 |
 |
 |
| Met34 |  |
 |
 |
 |
| Phe81 |  |
 |
 |
 |
1.0 |
2.0 |
3.0 |
4.0 |
 |
 |
| Главная страница | Первый семестр | Второй семестр | Третий семестр | Обо мне | Ссылки |