Блок 3. Задание 2.
1.Задача - получить выравнивание вирусных белков, называемых "дельта-антигенами"
Для начала приведу невыровненные, и уже выровненные последовательности:
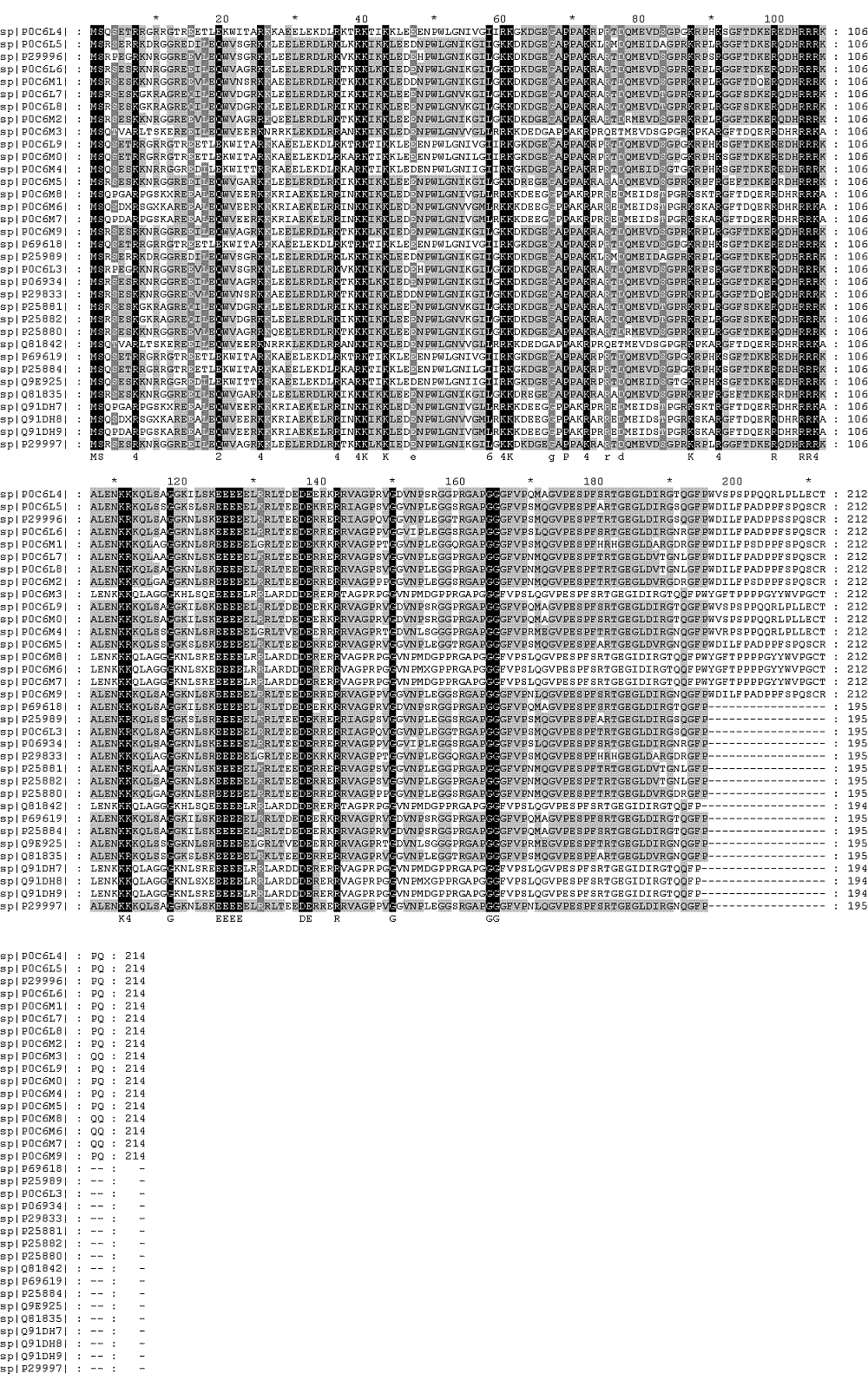
Невыровненные последовательности

Выровненные последовательности
Легко заметить, что у групп этих белков произошла вставка (или редукция) одного а.о. (соответственно 3-х нуклеодидов) в двух местах: в столбце 10 и в столбце 60. При этом можно выделить 3 группы белков: у которых в столбце 10 - аргинин, а в столбце 60 - гэп, группа у которой в столбце 10 - гэп, а в столбце 60 - глицин, и группа у которой в обоих столбцах гэпы. Интересно обратить внимание на то, что в столбцах 197-216 у некоторых белков есть участки последовательности, а у некоторых - нет, притом во всех трех группах есть как те белки, у которых есть эти участки, так и те, у которых этих участков нет. При этом можно заметить, что внутри групп эти участки очень схожи, а между группами эти участки имеют лишь некоторую (но заметную невооруженным глазом) долю сходства. Кстати, можно обратить внимание на то, чтоучастки в середине цепи, не совпадающие у всех дельта-антигенов часто совпадают внутри групп Таким образом, можно предположить (мне лично это кажется более вероятным), что появление или редукция а.о. в столбцах 10 и 60 повлекли за собой изменение концевых участков 197-216 различные в разных видах, и затем, уже конвергентно происходила редукция этих концевых участков во всех трех группах белков. Впрочем это лишь предположение, которое можно вынести только из просмотра выравнивания не смотря на роли и функции участков цепи.
2.Выравнивание набора гомологов своего белка
Для начала про выборку:
Т.к. гомологов (притом верных) как ни крути выходило очень много, я, дабы долго не мучаться с этим, выбрал из всех гомологов начиная с e-value около 10-5 и меньше.
Количество получилось несколько больше. чем ожидалось, но на то это и была произвольная выборка.
В итоге получилось такое выравнивание:
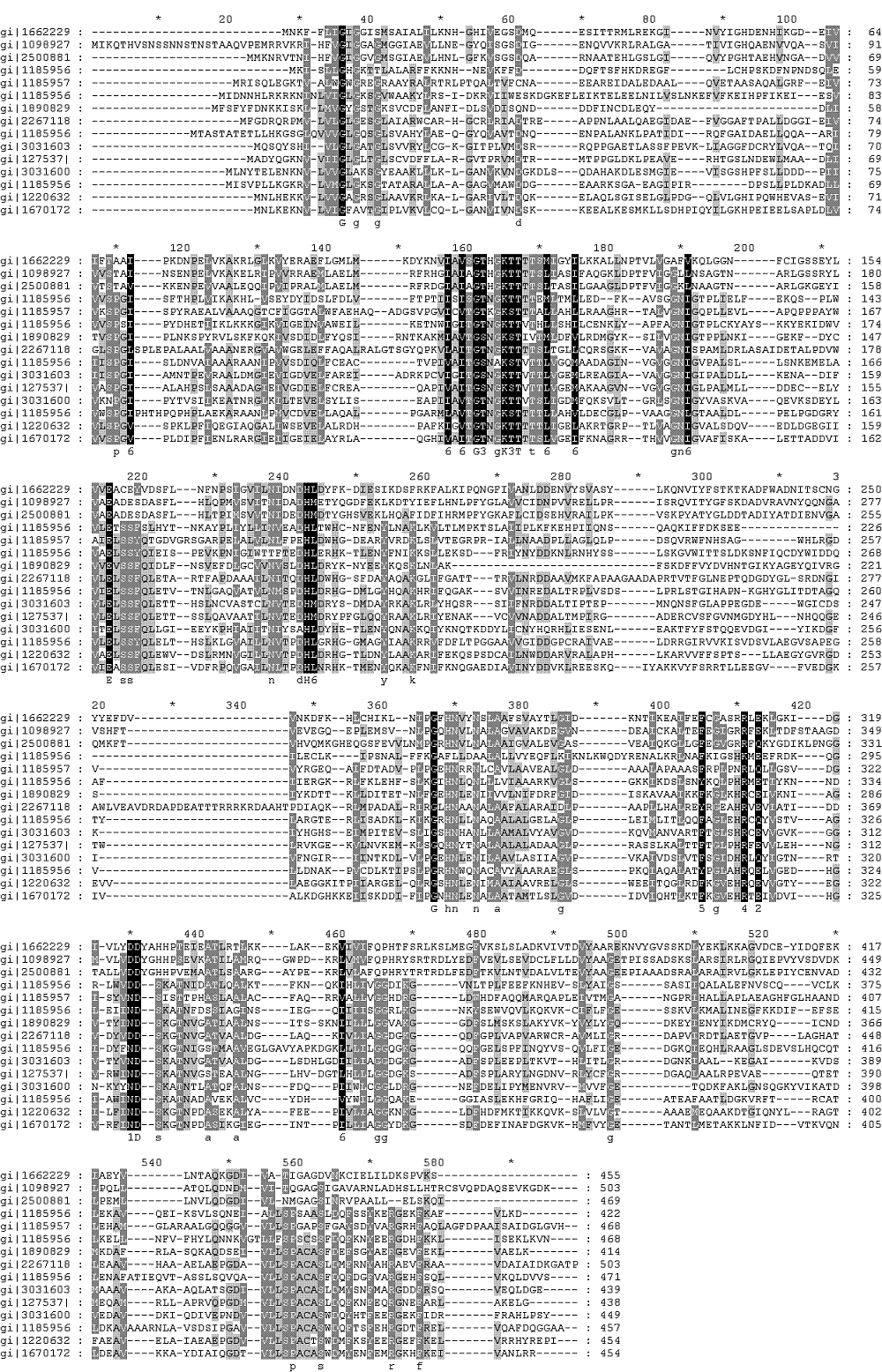
Кстати, что огорчило, так это то, что в выравнивании использовались наименее распространенниые идентификаторы (вероятно потому, что они стояли первыми, плюс полностью "убило" то, что программа переставила очередность последовательностей поэтому привожу список белков попавших в выборку, в той последовательности, что они приведены в выравнивании:
>gi|166222969 |sp|A4XGE5.1 |MURC_CALS8 >gi|109892700 |sp|Q47VR0.1 |MURC_COLP3 >gi|25008815 |sp|Q9JSZ8.2 |MURC_NEIMA >gi|118595659 |sp|Q1CU35.1 |MURD_HELPH >gi|118595703 |sp|Q3BR29.1 |MURD_XANC5 >gi|118595673 |sp|Q319C4.1 |MURD_PROM9 >gi|189082909 |sp|B0TZ72.1 |MURD_FRAP2 >gi|226711888 |sp|B1JV76.1 |MURD_BURCC >gi|30316031 |sp|Q8E9P6.1 |MURD_SHEON >gi|118595677 |sp|Q1Q883.1 |MURD_PSYCK >gi|30316001 |sp|Q8CSX6.1 |MURD_STAES >gi|127537 |sp|P14900.3 |MURD_ECOLI >gi|118595663 |sp|Q2W0H5.1 |MURD_MAGMM >gi|122063244 |sp|Q24TE4.1 |MURD_DESHY >gi|167017286 |sp|A8MH34.1 |MURD_ALKOOТеперь обратим внимание собственно на выравнивание:
Вообще можно обратить внимание на то, что выравнивание визуально выглядит как будто состоящее из частей, при этом чаще у одной цепи (притом все время разной) в "промежутках" есть цепь. а у всех остальных в этом месте находится гэп.
Сразу бросается в глаза очевидно консервативный блок в 156-172 столбцах (107-123 а.о. белка MurD_ecoli), с точностью до концевых а.о.
Также можно заметить еще несколько консервативных участков:
33-42 столбцы (10-19 а.о. белка MurD_ecoli)
105-112 столбцы (68-75 а.о. белка MurD_ecoli)
189-192 столбцы (138-140 а.о. белка MurD_ecoli)
213-221 столбцы (156-164 а.о. белка MurD_ecoli)
236-245 столбцы (177-186 а.о. белка MurD_ecoli)
участок 365-380 (264-279 а.о. белка MurD_ecoli) я бы не рискнул назвать целиком консервативным, можно лишь сказать, что в нем есть консервативные а.о.
аналогично про участок 405-415 (297-307 а.о. белка MurD_ecoli)
далее идет консервативный участок 429-431 (316-318 а.о. белка MurD_ecoli)
460-463 столбцы (340-343 а.о. белка MurD_ecoli)
Далее после 555 столбца (408 а.о. белка MurD_ecoli) идет участок, про который можно сказать. что он обладает некоторой долей консерватизма.
Заметить какие либо очевидные неточности мне не удалось, наоборот, ввиду общего расположения гэпов (продолжительные, общие, часто устроены так, что между гэпами лежит консервативный участок), я считаю, что такое выравнивание довольно верно.
3.Другие программы множественного выравнивания
Для начала рассмотрим выравнивание созданное mafft:
Я не буду приводить картинку целиком, приведу только начало:

При сравнении этого выравнивания с выравниванием muscle можно заметить, что консервативные участки в большинстве своем остаются те же самые, но гэпы меняются довольно существенно.
И в этом выравнивании можно точно указать неточность (не зря же я привел начало выравнивания) - гомология метионинона в столбце не оправдана, т.к. любой белок не подвергшийся сплайсингу начинается с метионина.
При работе программы edialign приятно было то, что идентификаторы белков остались буквенные.
Приведу опять только начало:
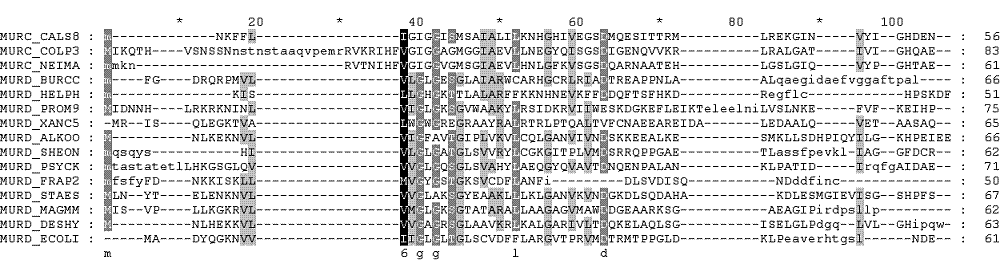
При сравнении выравнивании можно заметить те же тенденции: сходство консервативных участков (что еще больше подтверждает их консерватизм) и расхожение гэпов (притом между всеми тремя выравниваниями).
Мне непонятна осталась логика выставления метионинов и гэпов в первом столбце, именно такое выравнивание начального участка мне кажется еще менее вероятным, чем в варианте mafft.
4. Знакомство с некоторыми программами обработки множественных выравниваний
Не понял, в чем конкретно состояло задание, но было интересно.
Программа plotcon позволяет графически изобразить степень консерватизма в зависимости от столбца выравнивания.
Программа consambig позволяет получить "резюме" множественного выравнивания - каждому столбцу соответствует столбец выравнивания, и в нем показывается, насколько схожи а.о. в этом столбце выравнивания.
Программа distmat позволяет получить матрицу, в которой будет показано предпологаемое эволюционное родство между каждой парой белков.
Под конец приведу ссылки на файлы, использованные в данном задании:
Невыровненные дельта-антигены в fasta формате
Выровненные дельта-антигены в fasta формате
Выровненные дельта-антигены в msf формате
Невыровненные гомологи белка MurD_ecoli (выборка) в fasta формате
Выравнивание выборки гомологов белка MurD_ecoli в fasta формате
Выравнивание выборки гомологов белка MurD_ecoli в msf формате
Выравнивание программой mafft выборки гомологов белка MurD_ecoli в fasta формате
Выравнивание программой mafft выборки гомологов белка MurD_ecoli в msf формате
Выравнивание программой edialign выборки гомологов белка MurD_ecoli в fasta формате
Выравнивание программой edialign выборки гомологов белка MurD_ecoli в edialign формате
График консервативных участков выполненный программой plotcon в формате ps
"Резюме" выравнивания выборки гомологов белка MurD_ecoli в fasta формате выполненное программой consambig
Матрица эволюционного родства выравнивания выборки гомологов белка MurD_ecoli в distmat формате выполненное программой distmat
Александр Мартынов, 2008 ©