Выравнивание как отражение эволюции. JalView
1. Возможности JalView
В рамках освоения основных возможностей JalView, я построила выравнивание 6 последовательностей гомологичных белков из семейства HSP70 - белков-шаперонов DNAK.
Шапероны - это особый класс белков, основной функцией которых является “контроль” правильного сворачивания третичной и четвертичной структуры белка, а также сборка различных белковых комплексов. Такая функция должна быть важна для организмов всех систематических групп, а значит и белок должен быть достаточно консервативным. Это можно проверить с помощью выравнивания.
Я выбрала по два белка в каждом из трёх доменов: археи, эубактерии, эукариоты:
Q12WE6 - белок теплового стресса у Methanococcoides burtonii
Q01100 - выполняет аналогичную функцию у Haloarcula marismortui
P19993 – шаперон Mycobacterium leprae
A9ILH7 – шаперон Bartonella tribocorum
Q6FJI3 – белок теплового шока у Candida glabrata
P27322 – подобная функция у Solanum lycopersicum
Затем добавила в JalView нужные мне для выравнивая последовательности: File -> Add sequence -> text file , после чего выполнила выравнивание этих последовательностей: Web service -> Alignment -> TCoffee with Defaults (показался наиболее удобным).
После этих операций нужно было посмотреть на строго консервативные участки исследуемых белков. Для этого я «раскрасила» выравнивание по цветовой схеме ClustalX с условием Identity Threshold = 100%, в результате чего окрашенными оказались только полностью совпадающие участки.
 Добавлена разметка по типам консервативности: позиции, консервативные на 80% или более (C), абсолютно функционально консервативные (F), позиции с гэпами (G).
Добавлена разметка по типам консервативности: позиции, консервативные на 80% или более (C), абсолютно функционально консервативные (F), позиции с гэпами (G).
Основная информация о выравнивании представлена в таблице 1. Она была получена с помощью различных агрумаентов команды "infoalign" пакета EMBOSS.
Комментрарии: длина выравнивания составляет 748.
Таблица 1. Информация о выравнивании
| Name |
SeqLength |
Identical positions |
Identical positions (%) |
Similar positions |
Similar positions (%) |
Gaps |
Gaps(%) |
70% identical positions |
70% identical positions (%) |
different positions |
different positions (%) |
| DNAK_METBU_1 |
620 |
88 |
14,2% |
190 |
30,6% |
18 |
2,9% |
312 |
50,3% |
308 |
49,7% |
| DNAK_HALMA_1 |
635 |
88 |
13,9% |
190 |
29,9% |
15 |
2,4% |
306 |
48,2% |
329 |
51,8% |
| DNAK_MYCLE_1 |
620 |
88 |
14,2% |
190 |
30,6% |
17 |
2,7% |
301 |
48,5% |
319 |
51,5% |
| DNAK_BART1_1 |
630 |
88 |
13,9% |
190 |
30,2% |
18 |
2,9% |
319 |
50,6% |
311 |
49,4% |
| HSP7F_CANGA_1 |
694 |
88 |
12,7% |
190 |
27,4% |
12 |
1,7% |
139 |
20,0% |
555 |
80,0% |
| HSP72_SOLLC_1 |
644 |
88 |
13,7% |
190 |
29,5% |
16 |
2,5% |
264 |
41,0% |
380 |
59,0% |
2. Совершенно небывалая эволюция!
Это задание я решила выполнить на примере белка пилина Neisseria gonorrhoeae (pilE1). Для "создания мутагенных условий" я пользовалась командой
msbar пакета EMBOSS. При внесении мутаций я руководствовалась тем, что не должно быть блоковых(крупных) мутаций, 7 сайтов мутаций на поколение.
В резульате я получила 7 новых последовательностей, которые вместе с изначальной поместила в файл FASTA-формата (назвала последовательности p1, p2... p8).
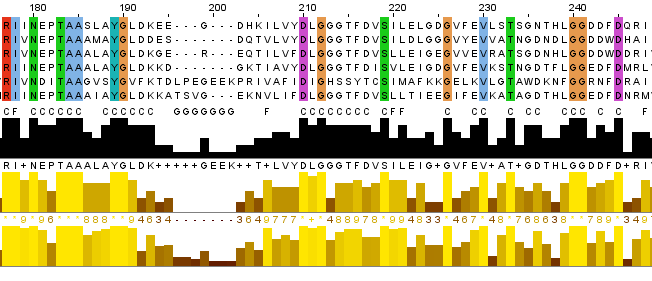
Первое выравнивание (автоматическое) представлено на рис.2, а "ручное" - на рис.3.
Такие мутации были получены при выполнении этого скрипта

Рис. 2 Автоматическое выравнивание
Исходя из выравнивания мы можем судить о том, какие мутации произошли:
1. При переходе от исходной последовательности к p1: Вставка лейцина(L) в 15 позиции, вставка валина(V) в 96й позиции и в 104й позиции, вставка треонина(Т) в 161й позиции, вставка аспартата(D) в 182й позиции,замена лизина(K) на лейцин(L) -184п, замена гистидина(H) на лизин(K) - 185, замена лейцина(L) на гистидин(Н)- 186, вставка лейцина(L) в 187
2. При переходе p1 -> p2: вставка K в 6й, вставка L в 16й, вставка L в 119й,вставка S в 171й, вставка G в 176й, втсавка R в 194й
3. При переходе p2 -> p3: вставка S в 18й, I - в 21й, T - в 22й, H - в 41й, S - в 83й, замена С на L в 153й, вставка N в 197й
4. При переходе p3 -> p4: делеция T в 22й, вставка D в 51й, L - в 68й, К - в 98й, S - в 121й, замена с S на I в 122й, вставка L в 152й, N - в 197й
5. При переходе p4 -> p5: делеция I в 21й, вставка V в 67й, вставка Q в 81й, вставка R в 142й, вставка G в 143й, замена E на N в 144й, делеция L в 152й, вставка L в 152й, делеция А в 169й
6. При переходе p5 -> p6: вставка А в 62й, вставка V в 63й, вставка E в 111й, вставка N в 152й, делеция T в 163й, замена R на S в 192й, инсерция R в 193й,
7. При переходе p6 -> p7: замена E на G в 14й, вставка А в 35й, замена R на A в 138й, вставка RR в 140й, 141й, вставка G в 154й, вставка L в 156й, вставка D в 169й.
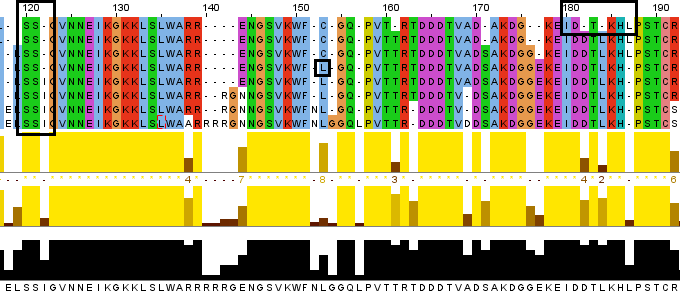
Рис. 3 Ручное выравнивание
Проанализировав полученное выравнивание, я внесла в него следующие изменения:
1. Лизин из последовательности р4 с 152й преместила на 153ю позицию.
2. Последовательность р1 сдвинула на одну позицию вправо в выделенной области, таким образом все три аминокислотрных остатка попали в консервативные столбцы. Т.е с позиций 184-185-186 на позиции 185-186-187.
3. Серины из 122х позиций последовательностей р1, р2, р3 и р4 я переместила в колонку 121, где располагаются серины р5, р6 и р7. Таким образом, теперь колонка 121 полностью состоин из серинов, а колонка 122 - содержит гэпы (р1 ,р2, р3, р4)
и изолейцины (р5, р6, р7).
Ссылка проект JalView
Источники:
1.Uniprot.org
2.en.wikipedia.org/wiki/Chaperone
3.kodomo.fbb.msu.ru/wiki/Main/alignment
|