Поскольку как правило, только на уровне вида ортологичность участков геномов можно определить по сходству нуклеотидных последовательностей, были выбраны бактерии одного вида, геном которых представлен одной собраной полностью хромосомой. Теоретически, геномные перестройки должны быть характерной чертой патогенов, которые находятся в постоянной гонке вооружений со своим хозяином. По этой причине, я стала искать их у рода Yersinia (гаммапротеобактерии). В итоге выбрала три штамма одного вида Yersinia ruckeri. Оказалось, что эти бактерии вызывают "Enteric redmouth disease" у морских рыб (название болезни я предпочла оставить без перевода).
1. Yersinia ruckeri (NZ_CP011078.1)
2. Yersinia ruckeri (NZ_CP023184.1)
3. Yersinia ruckeri (NZ_CP017236.1)
Будем называть их 1 бактерия, 2я бактерия и 3я баетрия, соответственно.
2.Вычисление сходства (identity %) на гомологичных участках геномов и покрытие геномов гомологичными участками
Была использована онлайн-версрия программы blast2seq. Query cover, полученные для трёх выравниваний равны:
1 + 2: NZ_CP023184.1 vs NZ_CP011078.1 - 96%, E-value 0.0
1 + 3: NZ_CP011078.1 vs NZ_CP017236.1 - 94%, E-value 0.0
2 + 3: NZ_CP023184.1 vs NZ_CP017236.1 - 96%, E-value 0.0
Кроме того, результат - карты сходства последвательностей (рис. 1-3).
3. Исследование крупных геномных перестроек
На рисунках 1, 2 и 3 представлены карты локального сходства геномов NZ_CP023184.1 vs NZ_CP011078.1, NZ_CP011078.1 vs NZ_CP017236.1 и NZ_CP023184.1 vs NZ_CP017236.1, соответственно. В общем, я заметила интересный момент: 2я и 3я карты - это по сути одно и тоже, с небольшим сдвигом, что указывает на то, что NZ_CP011078.1(первая бактерия) и NZ_CP023184.1(вторая бактерия) - очень похожи, что подтверждает карта их локального сходства (1). В то же время, 3я бактреия от них отличается. По инсттитуту, где были собраны геномы я выяснила, что 1я и 2я были получены в разных институтах Канады, а 3я - в Австралии. Вряд ли можно судить о геномных перестройках у австралийских ерсиний относительно канадских, но всё равно интересно :). Подробнее о картах:
Из рисунка 1 видно, что геномы очень похожи, не наблюдается никаких геномных перестроек, тем более крупных. Есть совсем небольшие делеции.
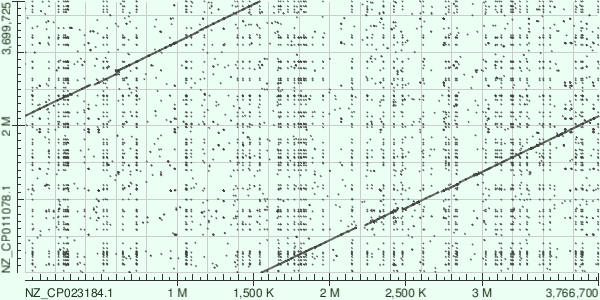
Рис 1. NZ_CP023184.1 vs NZ_CP011078.1.
Далее, можно описывать один из рисунков 2 и 3, тк они очень похожи. Рассмотрим рисунок 3. На картах наблюдается приличное число небольших делеций на протяжении обеих последовательностей. Из крупных перестроек видна очень большая инверсия (с 0,5 Мb и до 3,7 Mb), кроме того, в самом начале имеется дупликация.
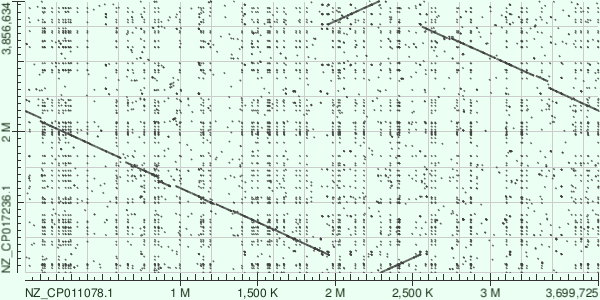
Рис 2. NZ_CP011078.1 vs NZ_CP017236.1
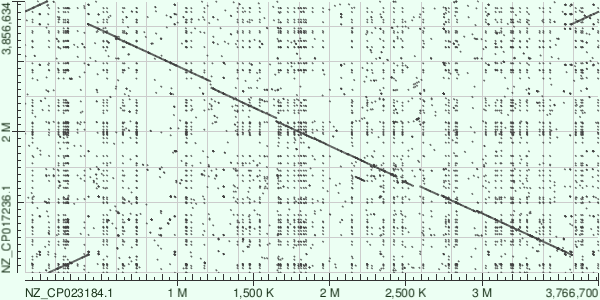
Рис 3. NZ_CP023184.1 vs NZ_CP017236.1