A- и В- формы ДНК. Структура РНК
1. Построение различных форм ДНК с помощью программы fiber пакета 3DNA.
Для построения a, b и z форм ДНК
были использованы команды -a, -b и -z (соответственно) пргограммы fiber. Команы выполнялись из командной строки, на выходе получилось 3 файла в формате .pdb.
Последовательность ДНК во всех формах представляет собой 5 повторений короткой последовательности GATC.
На рис.2 представлены отображения a, b и z - форм ДНК в программе JMol. Ссылки на соответствующие структуры:
gatc-a.pdb
gatc-b.pdb
gatc-z.pdb
2. Сравнение структур 3-х форм ДНК с помощью средств JMol.
Исследование положения цитозина в полученной Б-форме ДНК с помощью программы JMol показало,
что в сторону большой бороздки обращены атомы C32.C4, C32.C5, C32.C6, C32.n4, а в сторону малой C32.O2, C32.c2, C32.n3.
На рисунке 1 показана ДНК в б-форме (для наглядности выделены нуклеотиды с цитозином) и изображение цитозина в MarvinSketch.
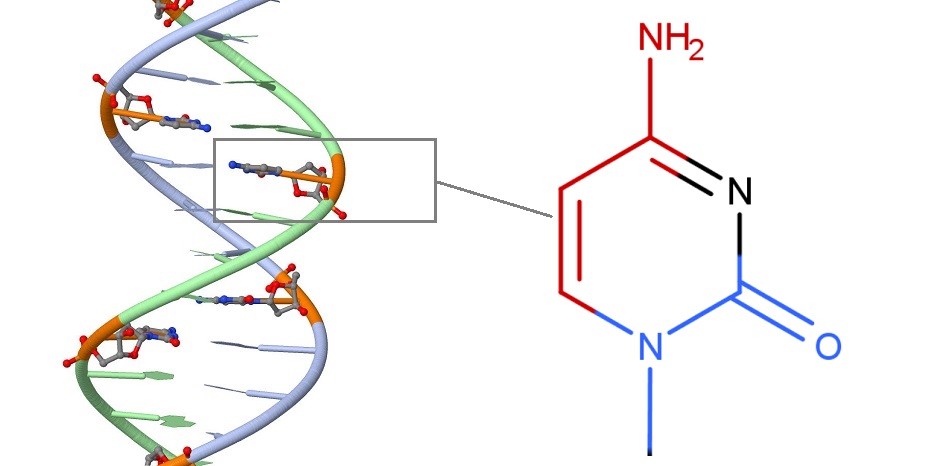
Рис 1. Красным цветом выделены атомы, смотрящие в сторону большой бороздки, синим - в сторону малой.
Сравнение основных спиральных параметров разных форм ДНК было выполнено в программе JMol и может быть представлено в виде таблицы 1.
| |
A-форма |
B-форма |
Z-форма |
| Тип спирали (правая или левая) |
Правая |
Правая |
Левая |
| Шаг спирали (A) |
28,03 |
33,75 |
43,5 |
| Число оснований на виток |
11 |
10 |
12 |
| Ширина большой бороздки |
16.81[30:B.P - 13:A.P] |
17.21[6:A.P - 32:B.P] |
15.17[32:B.P - 9:A.P] |
| Ширина малой бороздки |
7.98[4:A.P - 31:B.P] |
11.69[30:B.P - 15:A.P] |
9.87[15:A.P - 30:B.P] |
Табл.1
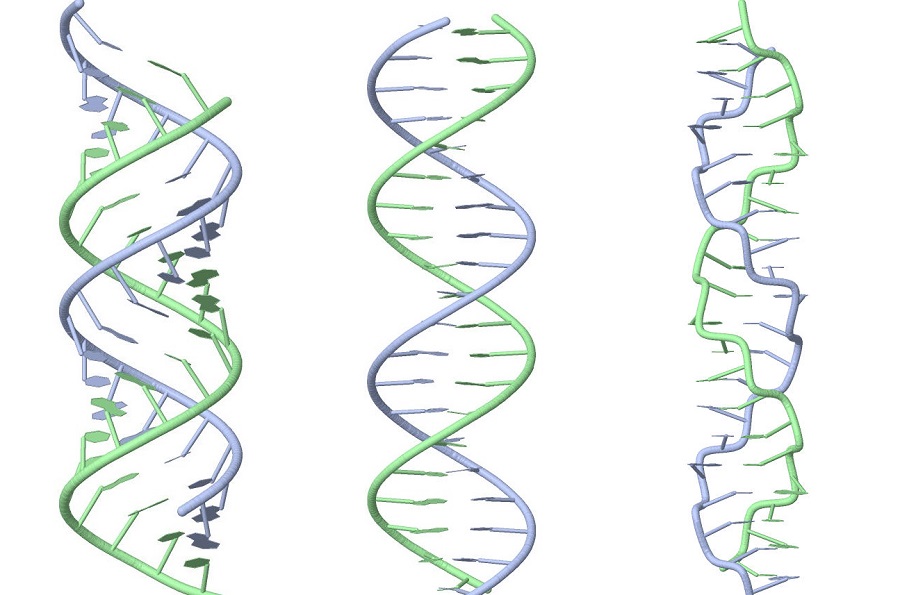
Рис. 2 a-, b- и z- формы ДНК
3. Определение параметров структур нуклеиновых кислот
Параметры структур нуклеиновых кислот определялись с помощью программ пакета 3DNA.
Для определения значения торсионных углов в заданной структуре тРНК (pdb id: 2dxi), были использованы программы find_pair и analyze.
Исследуемый РНК-белковый комплекс представлен в двух отображениях, в работе я рассмотриваю только одно из них. Из карты торсионных углов видно, что
стуктура РНК больше всего похожа на А-форму ДНК.
Средние значения торсионных углов показаны в таблице 2. Также в этой таблице приверены значения для торсионных углов самого деформированного нулеотида.
Стоит отметить, что колебания углов для различных форм ДНК невелики, хоть и наблюдаюся.
| | alpha | beta | gamma | delta | epsilon | zeta | chi |
| tRNA | -42,3073 | 71,97143 | 48,84643 | 86,25357 | -132,296 | -52,4625 | -133,959 |
| a-form | -51,7 | 174,8 | 41,7 | 79,08 | -147,7947368 | -75,09473684 | -157,2 |
| b-form | -29,9 | 136,34 | 31,15 | 143,3 | 140,8 | -160,5 | 98 |
| z-form | -43,8 | 21,1 | -61,45 | -42,7 | -100,05 | 8,6 | -95,6 |
| Guanosin | -175 | 174,5 | 56 | 88,2 | -143,1 | -72 | 179,8 |
Табл.2
Таким образом, деформированный нуклеотид - 22й гуанозин.
Определение структуры водородных связей заключалось в определении номеров нуклеотидов, образующих стебли(stems) во вторичной структуре заданной тРНК,
поиске неканонических пар оснований в структуре тРНК и выявлении наличия дополнительных водородных связей в тРНК, стабилизирующих ее третичную структуру.
Результаты работы можно посмотреть в таблице 3.
| Стебли шпилек | Неканонические пары нуклеотидов | Дополнительные связи |
C501[G]G-----C[C]572
…
C507[A]A-----U[U]566 | C502[G]G-*---U[U]571C
C554[U]U-**--A[A]558C
C555[U]U-**+-G[G]518C
C538_:[A]A-**--C[C]532C
C544[A]A-**--G[G]526C
C513[U]U-*---G[G]522C
C515[G]G-**+-C[C]548C
|
C554[U]U-**--A[A]558C
C555[U]U-**+-G[G]518C
C514[A]A-**--U[U]508C
C515[G]G-**+-C[C]548C
|
C549[G]G-----C[C]565
…
C553[G]G-----C[C]561C |
C538[A]A-**--C[C]532
...
C544[A]A-**--G[G]526C |
Табл. 3
Казалось бы: почему U-A и G-C - неканонические пары? Всё дело в том, что азотистые основания образуют неправильные (неканонические) связи друг с другом, а значит это не Уотсон-Криковское взаимодействие.
Стекинг-взаимодействие на рис. 4.
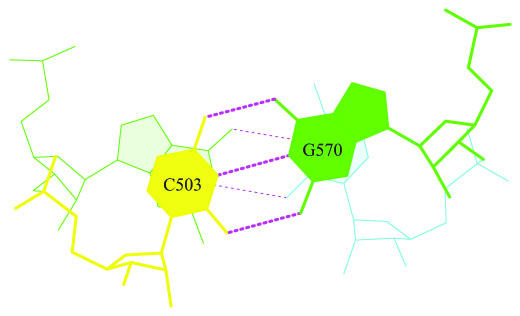
Рис.4
|