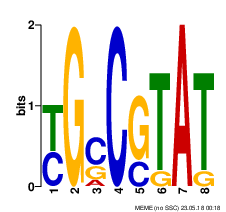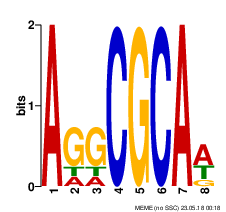Из списка "Popular organisms" я выбрала штамм с 8 записями Swiss-Prot, аннотированных как участвующие в биосинтезе пуринов для данного штамма. Выбранный штамм: Yersinia pestis bv. Antiqua (strain Angola) , Uniprot-мнемоника штамма: YERPG, список выбранных белков представлен в таблице 1.
Табл. 1 Список выбранных белков
| Entry | Entry name | Protein names | Gene names | Gene coordinates |
| A9R5W9 | PURT_YERPG | Formate-dependent phosphoribosylglycinamide formyltransferase | purT | 1687096..1688277 |
| A9QYM6 | PURA_YERPG | Adenylosuccinate synthetase, AMPSase, AdSS | purA | complement(1339605..1340471) |
| A9R256 | FOLD_YERPG | Bifunctional protein FolD | folD | complement(701035..702333) |
| A9R7Z2 | GUAA_YERPG | GMP synthase [glutamine-hydrolyzing] | guaA | complement(422193..423770) |
| A9R8E1 | PUR9_YERPG | Bifunctional purine biosynthesis protein PurH | purH | complement(485854..487443) |
| A9QZB3 | PURR_YERPG | HTH-type transcriptional repressor PurR | purR | not found :( |
| A9QZW5 | PUR5_YERPG | Phosphoribosylformylglycinamidine cyclo-ligase | purM | complement(3307987..3309030) |
| A9R2H1 | PUR7_YERPG | Phosphoribosylaminoimidazole-succinocarboxamide synthase | purC | 3322470..3323183 |
Координаты исследуемых генов были найдены в полном геноме бактерии (скачан из ENA/EMBL). Для каждого из генов выбранных белков были записаны координаты Upstream-региона из 100 нуклеотидов (то есть 100 нуклеотидов с 5'-стороны от статового кодона гена). Координаты Upstream-регионов приведены в табл.2. Ссылка на fasta-файл с вырезанными Upstream-регионами: Upstream.fasta
Табл. 2 Список выбранных белков c координатами Upstream-регионов из 100 нуклеотидов
| Entry | Entry name | Upstream |
| A9R5W9 | PURT_YERPG | 1686996 - 1687096 |
| A9QYM6 | PURA_YERPG | 1340471 - 1340571 |
| A9R256 | FOLD_YERPG | 702333 - 702433 |
| A9R7Z2 | GUAA_YERPG | 423770 - 423870 |
| A9R8E1 | PUR9_YERPG | 487443 - 487543 |
| A9QZB3 | PURR_YERPG | :( |
| A9QZW5 | PUR5_YERPG | 3309030 - 3309130 |
| A9R2H1 | PUR7_YERPG | 3322370 - 3322470 |
Для того, чтобы вырезать необходимые участки из файла с геномом, выполнялись следующие команды (приведены примеры для прямой и обратной цепей):
>descseq CP000901.txt -sbegin1 1686996 -send1 1687096 -osformat2 fasta -out purT -desc "up purT"
>descseq CP000901.txt -sbegin1 1340471 -send1 1340571 -sreverse1 -osformat2 fasta -out purA -desc "up purA"
Для поиска мотивов (3 мотива) в последовательностях, из командной строки была запущена программа MEME.
>ememe upstream.fasta -nmotifs 3 -revcomp
Ссылка на полную выдачу MEME: meme.html
Разберём, что говорят эти результаты. Является данная последовательность мотивом или случайной находкой - позволяет определить его информационное содержание (Information Content). Оно приводится для каждого мотива. По грубым оценкам, слово длины n = I/2 встречается в геноме раз в 4^n пар нуклеотидов. Кроме того, для каждгог мотива приводится E-value. Оно показывает, насколько велика вероятность находки с таким же или большим весом. Визуалзируется мотив (наиболее правдоподобная его последовательность) с помощью LOGO. LOGO - это диаграмма, в которой высота столбца равна I рассматриваемой позиции (показывает вклад в суммарное I всего мотива), а высота букв равна произведению I столбца и вероятности встретить данную букву на данной позиции. Ниже приведены LOGO трёх найденных программой мотивов.