Для данной последовательности белка неоьходимо было составить семейство гомологов, пользуясь PSI-BLAST. Я выбрала идентификатор Q7VDL2. Это septum site-determining protein MinC - белок, который ингибирует клеточное деление бактерий путём блокировки формирования Z-кольца полярной перегородки. Принадлежит морской цианобактерии Prochlorococcus marinus. С помощью белкового BLAST в NCBI (PSI-BLAST и поиск по банку Swiss-Prot), осуществлялся поиск выбранного белка. Результаты каждой иттерации вносились в таблицу ниже.
Табл.1 Результат поиска в BLASTP
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 145 | Q7N522.1 | 0,003 | B2VJ44.1 | 0,008 |
| 2 | 188 | B6JKX0.1 | 7e-08 | A7H8E6.1 | 0.062 |
| 3 | 188 | Q9ZM51.1 | 2e-12 | A7H8E6.1 | 0.013 |
| 4 | 189 | A8MHK8.1 | 0.001 | A7H8E6.1 | 0.012 |
Выяснилось, что несмотря на то, во второй и третьей иттерациях число находок было одинаковым, четвёртая иттерация позволила сделать еще одну находку, после чего результат стабилизироввался. В каждой последующей иттерации программа выдавала уведомление: "No new sequences were found above the 0.005 threshold". Все находки blast оносятся к белку MinC, их довольно много, и они относятся к различным таксонам бактерий, что говорит о том, что изучаемый белок достаточно консервативен (по крайней мере не специфичен).
2. Prosite
Цель задания: уточнить паттерн одного из семейств белков так, чтобы он описывал не все белки данного семейства, а только белки протеобактерий. Для этого использовалось выравнивание енолаз протеобактерий из 2 практикума: ENO_align.fasta.
В первую очередь банке Prosite (prosite.expasy.org) был найден паттерн (нашёлся всего один), описывающий выбранное семейство белков. Название семейства: Enolase (EC 4.2.1.11) Паттерн:[LIVTMS]-[LIVP]-[LIV]-[KQ]-x-[ND]-Q-[INV]-[GA]-[ST]-[LIVM]-[STL]-[DERKAQG]-[STA]. Далее, с помощью выравнивания, был сделан более строгий паттерн: N-S-I-L-[VI]-K-[VFI]-N-Q-I-G-[ST]-L-T-E-T, на рисунке ниже жёлтой рамкой выделен окончательный паттерн.
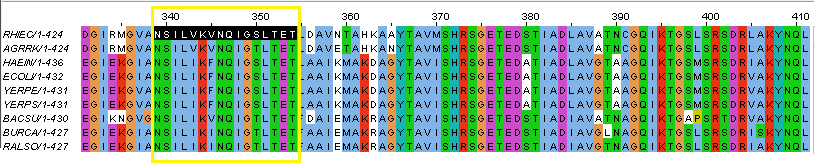
Рис.1 Строгий паттерн енолазы на основе выравнивания.
Далее были найдены все соответствия полученному паттерну в банке Swiss-Prot. Всего на выходе получилось 384 находки, которые были сохранены в файл в формате matchlist: hits.
Для сравнения списка находок с "правильным" списком, то есть со списком всех представителей данного семейства белков из Proteobacteria, имеющихся в Swiss-Prot, я получила "правильный" список с помощью сайта Uniprot: Entry Name [ID] - ENO_*, Taxonomy [OC] - Proteobacteria. Выдача uniprot (396 находок): right_hits . С помощью Excel я сравнила списки белков, найденных построенным паттерном, и белков протеобактерий из семейства. Результаты сравнения приведены в таблице ниже.
Табл. 1: TP: True positives - число истинных находок (то есть размер пересечения списков), FP: False positives - число ложных находок (число тех белков, которые нашлись паттерном, но не входят в правильный список), FN: False negatives - число ненайденных
| TP | FP | FN |
| 218 | 166 | 164 |