1. Указать перестройки доменной архитектуры на ветвях филогенетического дерева домена
2.Привязать изменения доменной архитектуры к эволюции видов (таксономии) с помощью филогении видов, из которых получены белки.
Задание 1. Построить выравнивание представителей домена Pfam белков с разной доменной архитектурой
1. В первую очередь я выбрала подходящий домен в Pfam: pfam.xfam.org/family/PF06456
AC: PF06456
ID: Arfaptin
Функция домена: арфаптин - это домен, который взаимодействует с ARF1 - небольшой ГТФазой, вовлеченной в процесс отпочковывания везикул от комплекса Гольджи. При связывании с ГТФазой, арфаптин формирует удлиненный серповидный димер.
Доменные архитектуры с выбранным доменом (всего их 28): список архитектур
Выравнивание доменов этого семейства было скачено с pfam, визуализировано с помощью программы JalView. Все выравнивание(arfaptin.mfa) раскрашено ClustalX, порог консервативности - 10. Cсылка на проект: arfaptin.jvp
2. Выбор доменных архитектур
В первую очередь получена таблица с информацией об архитектуре всех последовательностей, содержащих выбранный домен. Файл swisspfam, содержащий эту информацию для всех последовательностей Uniprot, скачан на kodomo (/srv/databases/pfam/swisspfam.gz), из него с помощью скрипта swisspfam-to-xls.py были отобраны последовательности с указанным доменом, составлена таблица для Excel:
>python swisspfam-to-xls.py -i /srv/databases/pfam/swisspfam.gz -z -p PF06456 -o arch.xls
Были выбраны две доменные архитектуры, они изображены на рисунке ниже, в таблице 1 представлена основная информация о выбранных доменных архитектурах.

Рис.1 Выбранные доменные архитектуры
Табл.1 Информация о выбранных архитектурах
| Архитектура | Число последовательностей | Характеристика других доменов |
| 1 architecture | 227 | PDZ-домен. Частовстречающийся структурный домен,
используется в сигнализации бактерий, дрожжей, растений, вирусов и животных. Играет ключевую роль
в заякоривании рецепторных белков в мембране, в формировании и функционировании сигнальных комплексов.
AC: PF00595. 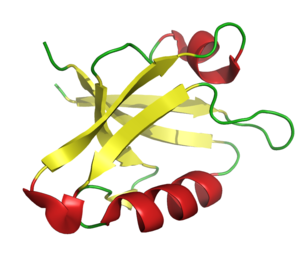 |
| 2 architecture | 155 | Islet cell autoantigen ICA69, C-terminal domain. Белки (69 kD), которые считаются аутоантигенами в островковых клетках при диабете 1го типа. Точная функция неизвестна. AC: PF04629 |
Из сводной таблицы были выделены AC последовательностей, содержащих данные доменные архитектуры. Данные последовательности были затем скачаны По AC с сайта Uniprot. После этого с помощью скрипта uniprot-to-taxonomy.py, для последовательностей была определена таксономия:
>python uniprot-to-taxonomy.py -i sequences_Uniprot.txt -o taxonomy.xls
3. Выбор таксона и подтаксонов
На основе сводной таблицы я составила таблицу, содержащую AC последовательностей, имеющих выбранные для работы доменные архитектуры, нее была перенесена информация по таксономии. Теперь среди представителей архитектур 1 и 2 были выбраны выбраны последовательности, относящиеся к таксону Metazoa и подтаксонам Chordata и Ecdysozoa.
Результат работы представлен в таблице:arc.xlsx .
4. Из исходного выранвивания, полученного на I этапе, были оставлены только выбранные последовательности (список их AC находится в файле selected.txt ). Командная строка:
>python filter-alignment.py -i arfaptin.mfa -m selected.txt -o selected.mfa -a "_"
IV этап Полученное выравнивание открыто и отредактировано в Jalview (удалены пустые позиции, убраны плохо выровненные концевые участки, удалены явно выбивающиеся последоватлеьности). Выравнивание было разбито на группы по доменным архитектурам, раскрашено по консервативности внутри групп (Clustal с 15% идентичности). Ссылка на проект JalView: selected.jvp .
Задание 2: построение филогенетического дерева домена
На основе отредактированного выравнивания в программе MEGA было построено филогенетическое дерево. Метод - Neighbor Joining. Проект MEGA: tree.mtsx . Скобочная форма дерева: tree.nwk . На рисунке 2 представлено филогенетическое дерева изучаемого домена.
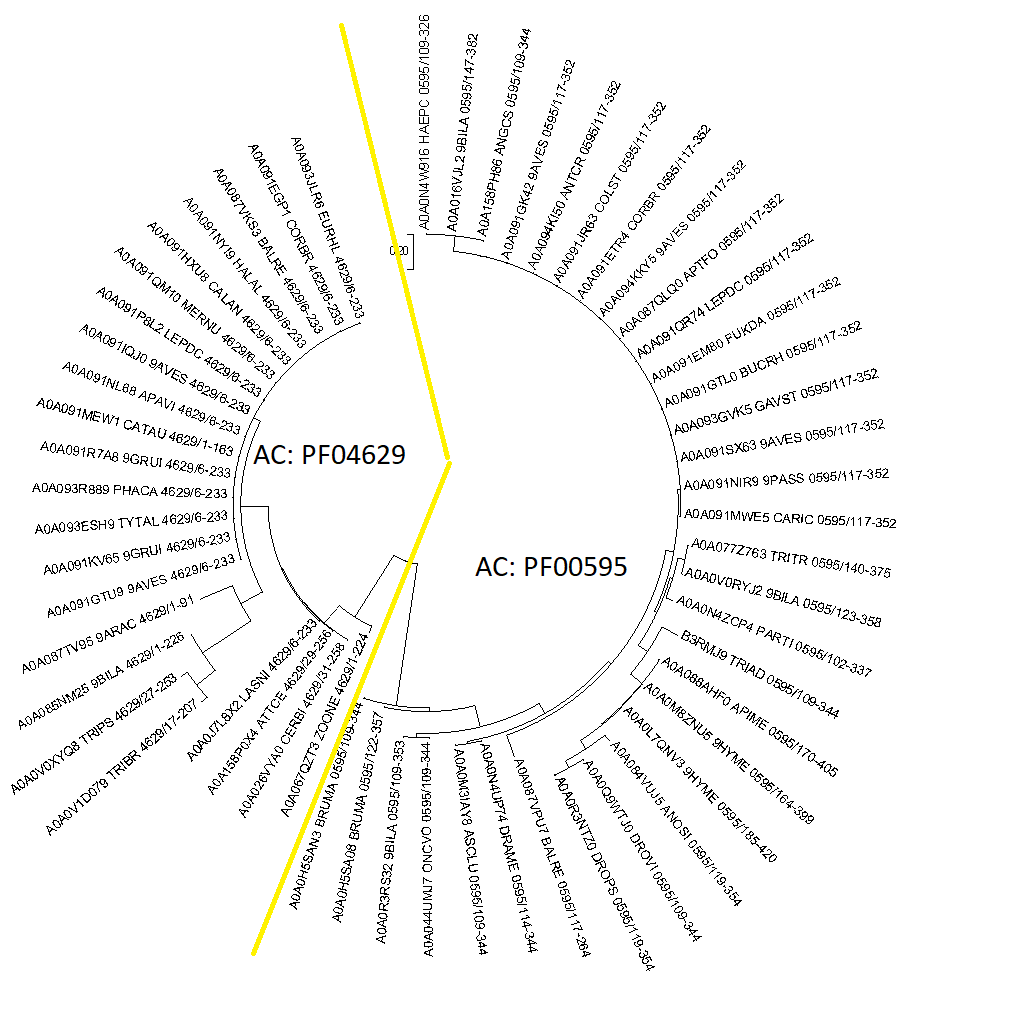 Рис. 2 Филогенетическое дерево
Рис. 2 Филогенетическое дерево