Прочтение последовательностей по Сэнгеру
1. Анализ качества чтений и Очистка чтений были выполнены в практикуме 11, доступном по ссылке
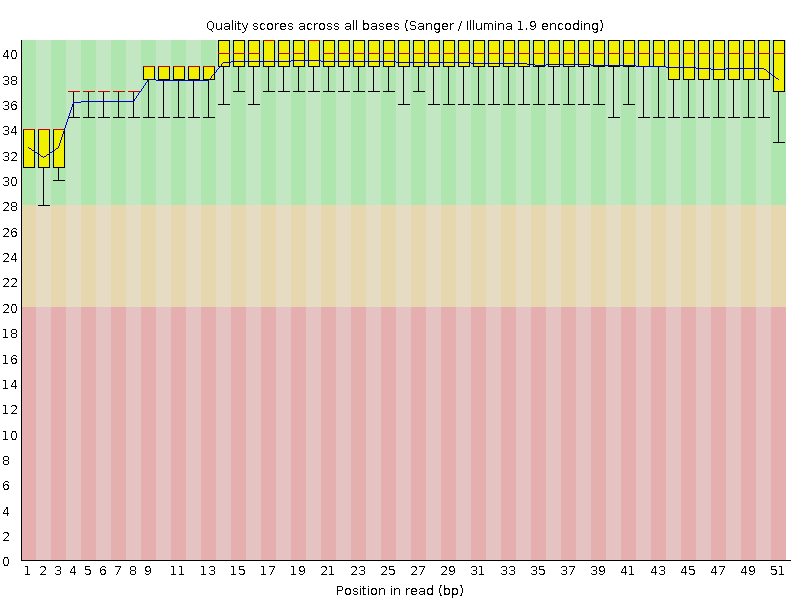
Картинки из FastQC "Per base quality" до чистки Рисунок 1. Per base sequence quality
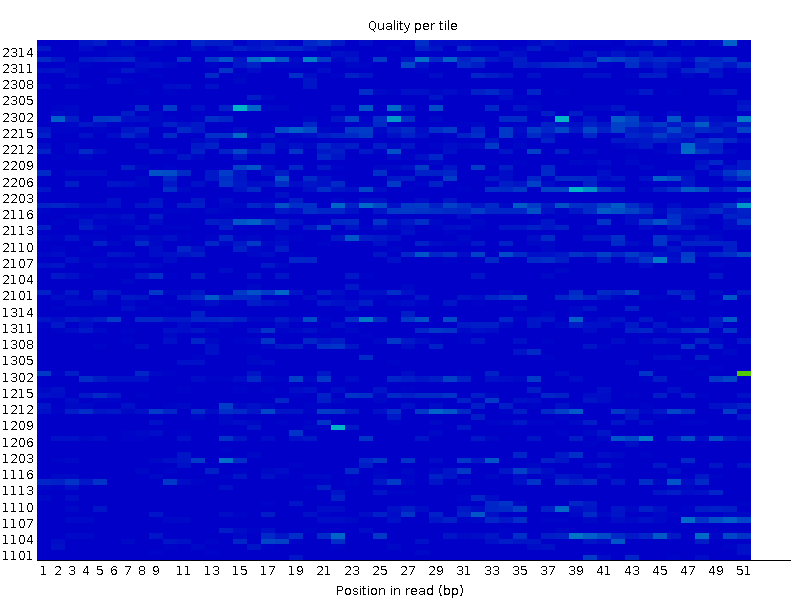
Картинки из FastQC "Per base quality" до чистки Рисунок 2. Per tile sequence quality
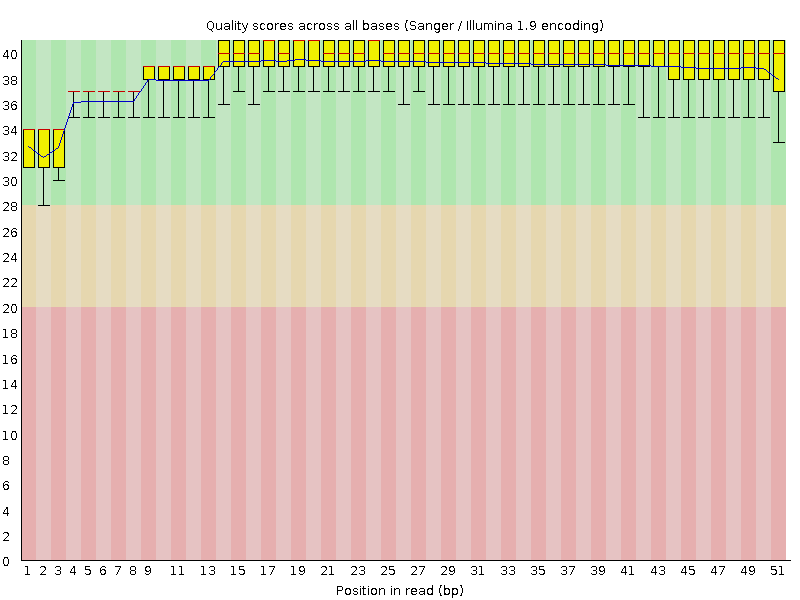
Картинки из FastQC "Per base quality" после чистки Рисунок 3. Per base sequence quality
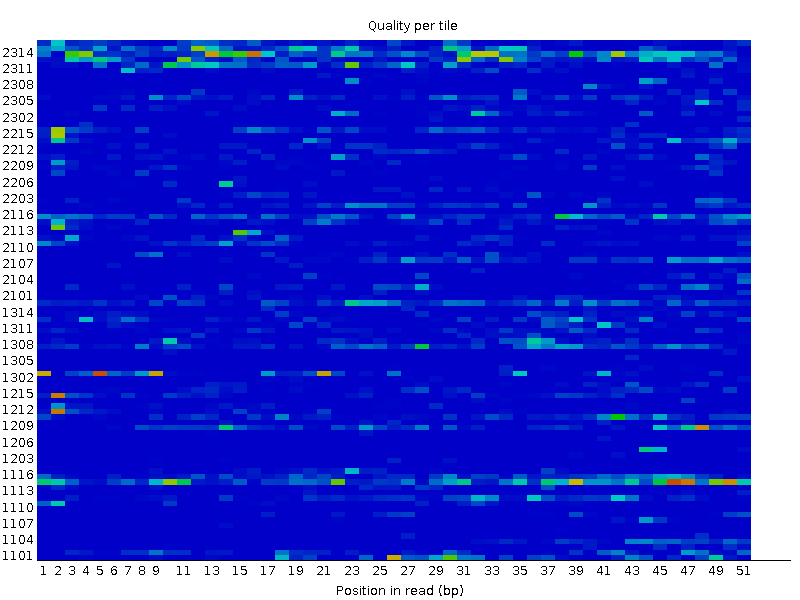
Картинки из FastQC "Per base quality" после чистки Рисунок 4. Per tile sequence quality
Trimmomatic: Input Reads: 7883 Surviving: 7842 (99,48%) Dropped: 41 (0,52%)
Диаграмма с BASE была не информативна, так как все риды были довольно хорошего качества. Ограничение длины рида 50 помогает улучшить показатель per TILE sequence quality. Желто-зелено-красные цвета показывают нуклеотиды плохого качества, ниже среднего. На фотографиях видно, как меняется качество после работы Trimmomatic.
2. Картирование чтений тоже было выполнено в практикуме 11
Таблица команд
| Команда (со всеми параметрами) | Описание (что делает) |
| fastqc chr15.2.fastq | контроль качества чтений в файле "chr15.2.fastq" с помощью программы FastQC |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr15.fastq chr15.2_out.fastq TRAILING:20 MINLEN:50 | программа Trimmomatic отрезает с конца каждого чтения в файле "chr15.2.fastq" нуклеотиды с качеством ниже 20, оставляет из файла "chr15.2_out.fastq" только чтения длиной не меньше 50 нуклеотидов и сохраняет их в файле "chr15.2_out.fastq" |
Input Reads: 5068 Surviving: 4946 (97,59%) Dropped: 122 (2,41%)
| Команда (со всеми параметрами) | Описание (что делает) |
| fastqc chr15.2_out.fastq | контроль качества чтений в файле "chr15_out.fastq" с помощью программы FastQC |
| hisat2-build chr15.fasta chr15_i | индексирование референсной последовательность в файле "chr15.fasta" с помощью программы Hisat2 |
| hisat2 -x chr15_i -U hr15_out.fastq --no-softclip -S chr15_ali.sam | построение выравнивание прочтений и референса с помощью программы Hisat2 |
Мы не использовали параметр, запрещающий картировать без разрывов, т.к. в данном случае работа производится над РНК. По сравнению с ДНК в ней могли произойти модификации связанные с перегруппировкой (сплайсинг).
3. Анализ выравнивания был выполнен в практикуме 11
4946 reads; of these:
4946 (100.00%) were unpaired; of these:
11 (0.22%) aligned 0 times
4935 (99.78%) aligned exactly 1 time
0 (0.00%) aligned >1 times
99.78% overall alignment rate
4. Подсчет чтений
Команда : htseq-count -f 'bam' -s 'no' -i 'gene_id' -m 'union' chr15_bin.sorted.bam /P/y14/term3/block4/SNP/rnaseq_reads/gencode.v19.chr_patch_hapl_scaff.annotation.gtf
Опции:
-f - Формат файла с вырвниванием: bam, sam
-s - Направление цепи: yes(прямая), no(обратная), reverse(прямая, развёрнутая, без направления)
-i - Атрибут GFF (используется как feature-ID)
-m - Режим для обработки ридов, которые легли более, чем на одну features, или легли с перекрытиями: union, intersection-strict, intersection-nonempty
5. Анализ результатов
__no_feature 2067
__ambiguous 0
__too_low_aQual 0
__not_aligned 11
__alignment_not_unique 0
Чтения легли на следующие гены:
ENSG00000140382.10 - 551
HMG20A high mobility group 20A [ Homo sapiens (human) ]
ENSG00000166035.6 - 458
LIPC lipase C, hepatic type [ Homo sapiens (human) ]
LIPC кодирует печеночную триглицеридную липазу, которая экспрессируется в печени. LIPC выполняет двойную функцию триглицеридгидролазы и лиганда / связывающего фактора для рецептор-опосредованного поглощения липопротеинов.
ENSG00000157766.11 - 276
ACAN aggrecan [ Homo sapiens (human) ]
Кодируемый белок является неотъемлемой частью внеклеточного матрикса в хрящевой ткани и выдерживает сжатие в хряще. Мутации в этом гене могут быть вовлечены в дисплазию скелета и дегенерацию позвоночника. В этом гене были обнаружены несколько альтернативно сплайсированных вариантов транскрипта, которые кодируют разные изоформы белка.
© Grigorjeva Masha