Сборка генома de novo
Код доступа SRR4240361 проекта по секвенированию бактерии Buchnera aphidicola
1. Подготовка чтений программой trimmomatic.
Прежде всего удаляем возможные остатки адаптеров. Для этого используем программу trimmomatic.
1) Создадим свой файл, где объединим все адаптеры вместе (adapters.fasta)
2) Удалим возможные остатки адаптеров
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240361.fastq noadapters.fastq ILLUMINACLIP:adapters.fasta:2:7:7
Input Reads: 7272621 Surviving: 7272621 (100,00%) Dropped: 0 (0,00%)
3) Удалим плохие буквы с концов чтений, оставив только чтения длиной не менее 30 и с качеством выше 20
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 noadapters.fastq noadapters2.fastq TRAILING:20 MINLEN:30
Input Reads: 7272621 Surviving: 6919623 (95,15%) Dropped: 352998 (4,85%)
2. Подготовка k-меров программой velveth
Для подготовки k-меров длины 29 была ипользована программа velveth. Команда:
velveth dir1 29 -short -fastq noadapters2.fastq
-fastq означает, что файл имеет расширение .fastq
29 - длина k-меров (hash_length)
dir1 - директория, содержащая файлы: Log, Roadmaps и Sequences
-short = shortpainted, чтения короткие и не парные
3. Сборка генома программой velvetg
Затем запустили программу velvetg (сборка на основе k-меров)
velvetg dir1
N50 of 72780
(Final graph has 1235 nodes and N50 of 72780, max 113937, total 690944, using 0/6919623 reads)
Информация о самых длинных контигах
| ID |
length |
cov |
| 3 |
113937 |
32.444684 |
| 5 |
97880 |
36.315008 |
| 12 |
85024 |
34.686418 |
| 1 |
72780 |
35.527508 |
4. Анализ
Сравним программой megablast каждый из трёх самых длинных контигов с хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253). Алгоритм запущен с параметрами по умолчанию.
К сожалению, контиг 3 оказался слишком объемным, программа вопротивилась и отключалась (я пробовала 15 раз). Поэтому в таблице наверху дабавлен четвертый по длине контиг - замена слишком длинному в задании 4.
| ID |
length |
E-value |
% Identity |
Gaps |
Chains |
Chr start |
Chr end |
Read start |
Read end |
| 5 |
97880 |
0.0 |
75% |
1966 |
Plus/Plus |
83021 |
173180 |
1989 |
93340 |
| 12 |
85024 |
0.0 |
74% |
1495
|
Plus/Minus |
384182 |
454069 |
7198 |
77702 |
| 1 |
72780 |
0.0 |
77% |
2022
|
Plus/Minus |
462496 |
531590 |
5 |
70106 |
Комментарии
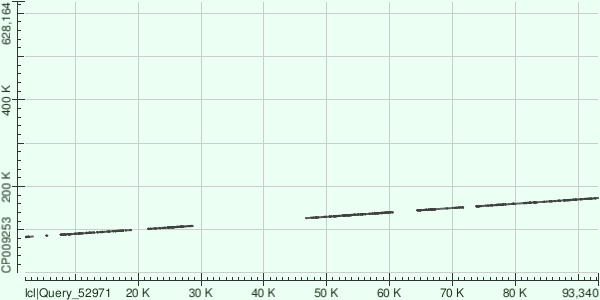
Всего 12 участков выравнивания. На карте пять сравнительно небольших разрывов и 1 огромный.
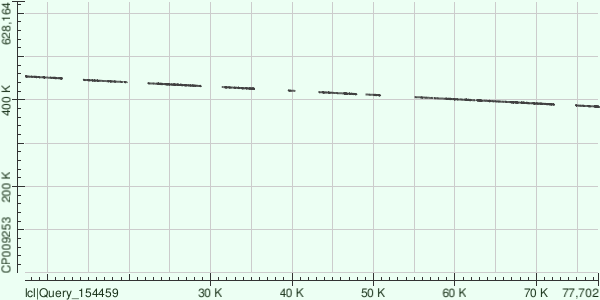
Всего 16 участков выравнивания. На карте - 5 средних разрывов, 1 мелкий и 2 крупных
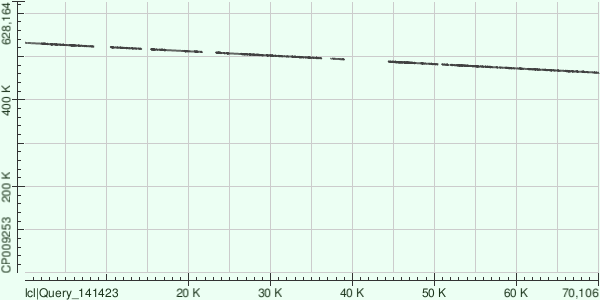
Всего 13 участков выравнивания. На карте - 5 мелких разрывов и 1 крупный
© Grigorjeva Masha


