Чтение последовательностей по Сэнгеру
1. Получить последовательность ДНК на основании данных, полученных из капиллярного секвенатора. Составить отчёт о проблемах при чтении хроматограмм
Данные хроматограммы из капиллярного секвенатора по Сэнгеру сохранены в формате .ab1 в виде двух файлов с прямой (F в названии файла - "forward chain") и обратной (R - "reverse chain") цепями. Для анализа и редактирования была использованна программа 4Peaks (после общего просмотра последовательностей были удалены нечитабельные участки хроматограммы, но в 4Peaks для этого специально существует утилита "Quality overview", которая помогает определить качество секвенирования на любом участке, в ней можно как выделить нужные учаски последовательности, так и обрезать их, как было указанно в задании)
Проблемный нуклеотид — тот, по которому вы приняли решение, отличное от предложенного программой, или вы согласились с программой, но необходимо было проанализировать хроматограммы и принять решение. Проблемные нуклеотиды в последовательности выделяйте строчными буквами.
Полиморфизм — это нуклеотид, про который вы решили, что в секвенируемой ДНК встречаются два (или более) варианта. Полиморфизмы обозначайте кодами вырожденных нуклеотидов (ambiguity codes).
Обе хроматограммы достаточно читаемы, вторая практически идеальна - уровень шума низкий, пики хорошо различимы. Редко встречаются места с шумами уровня пиков. Сигналы от целевой ДНК идут более или менее равномерно, с одинаковыми расстояниями между пиками , поэтому они хорошо различимы в спорных местах хроматограммы. Присутствует неравномерность силы сигнала и шума вдоль последовательности.
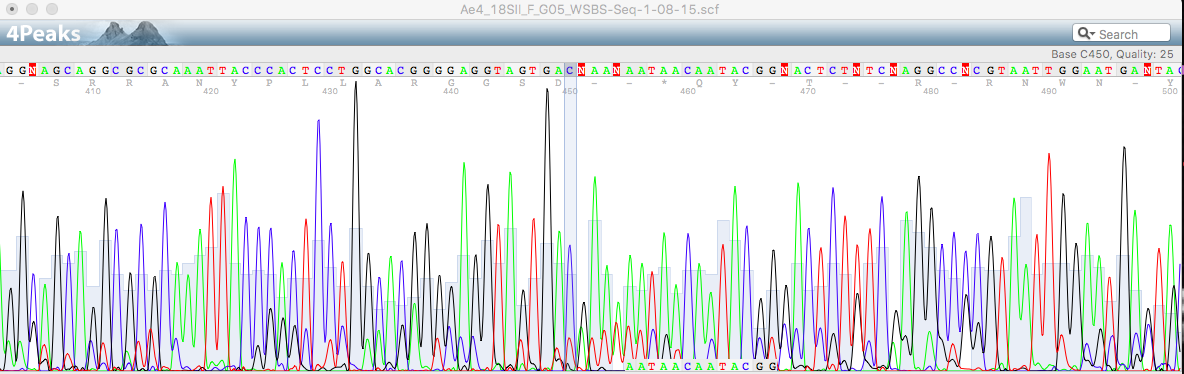
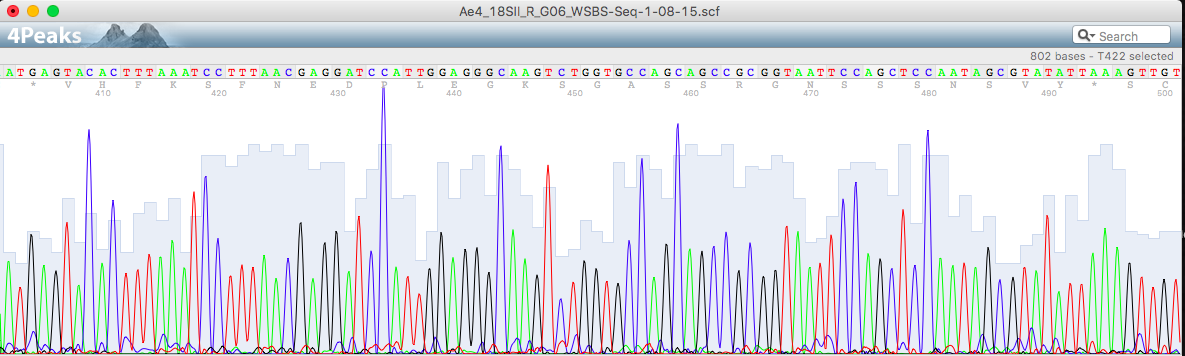
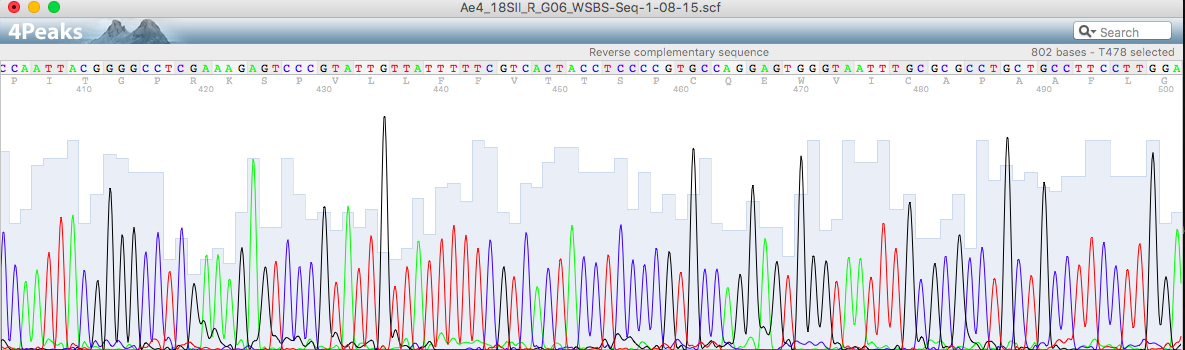
Прямая цепь
5' нечитаемый участок: 1-18
3' нечитаемый участок: 751-931
Обратная цепь
5' нечитаемый участок: 1-114
3' нечитаемый участок: 915-948
типичные сложные места:
1) шум выше среднего уровня шума и почти как сигнал
 _
_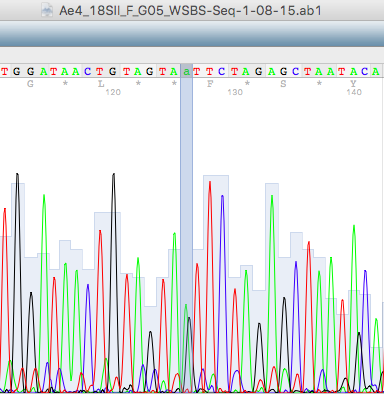
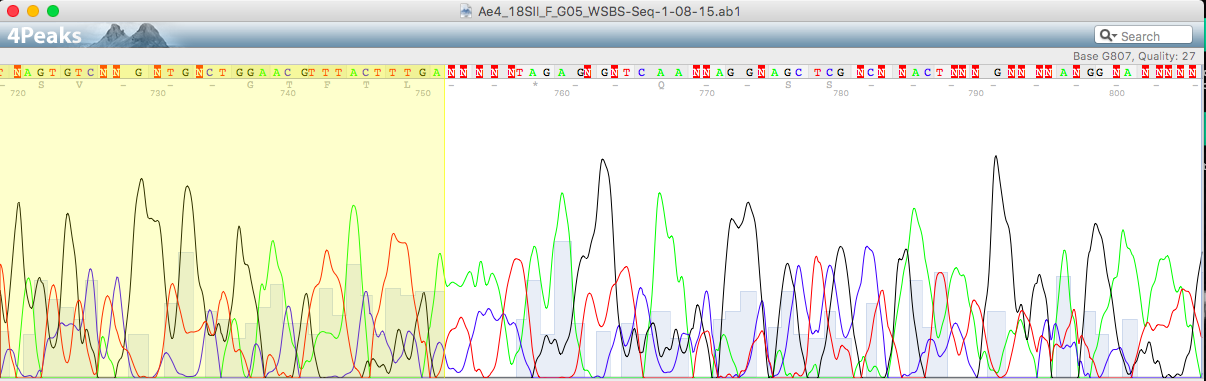
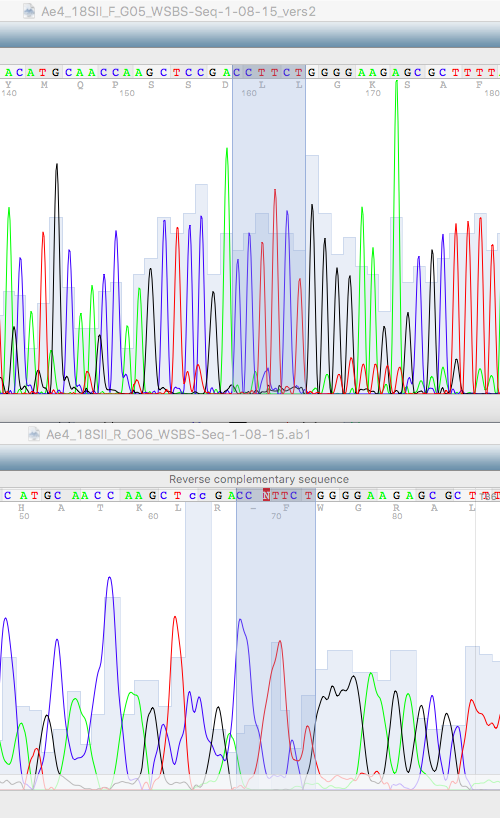
На картинке внизу пример того, как шум и сигнал накладываются друг на друга почти полностью (front), несмотря на это было принято решение поставить нуклеотид a (а не g), посколько в результатах второй последовательности (reverse) четко видно, что в этом месте стоит зеленый пик.
Так же в правой части картинки видно сложное место с наложением и небольши смещением пиков (в верхней хроматограмме черный пик перекрывает зеленый, при этом их центры смещены друг от друга). Поскольку черный пик был смещен относительно остальных и отсутствовал на нижней хроматограмме (формально там стоял высокий и четкий зеленый, даже выше, чем последующий, подтвержденный хроматограммой наверху), я решила его не учитывать, заменив N-букву на a.
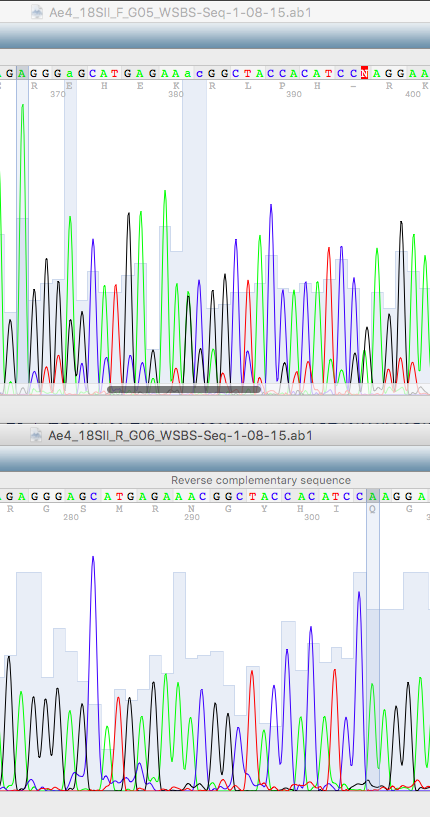
2) пик на нетипичном расстоянии от соседей:
вклинился лишний или, наоборот, соседние пики нетипично удалены
3) вместо двух или более пиков — один широкий.
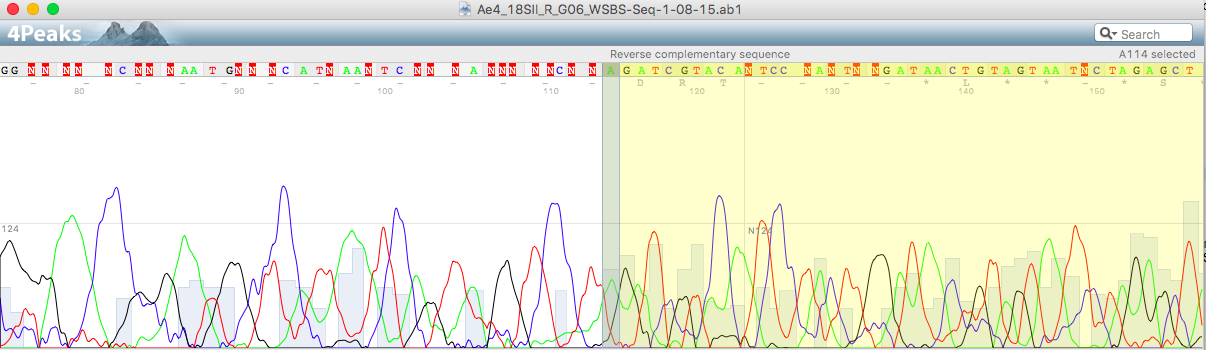
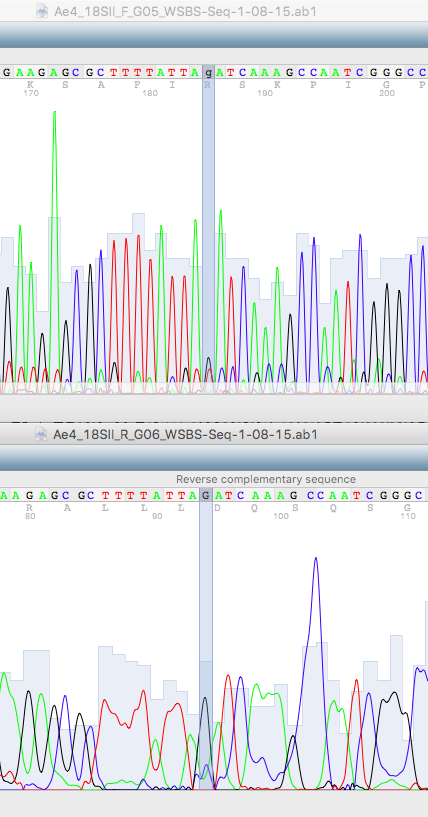
На первой картинке показано наличие лишней буквы в коде из-за лишнего синего пика (буква N сразу после выделенной области). Было принято решение удалить этот нуклеотид, потому что синий пик, вызывающий появление этой буквы, сравнительно невелик и может расцениваться как шум, а на второй хроматограмме и вовсе отсутствует. Плюс этот пример подходит для пункта 3, ведь на нем виден один большой красный пик, расценивающийся, как два красных пика, исходя из второй хроматограммы, где эти два пика явно разделены.
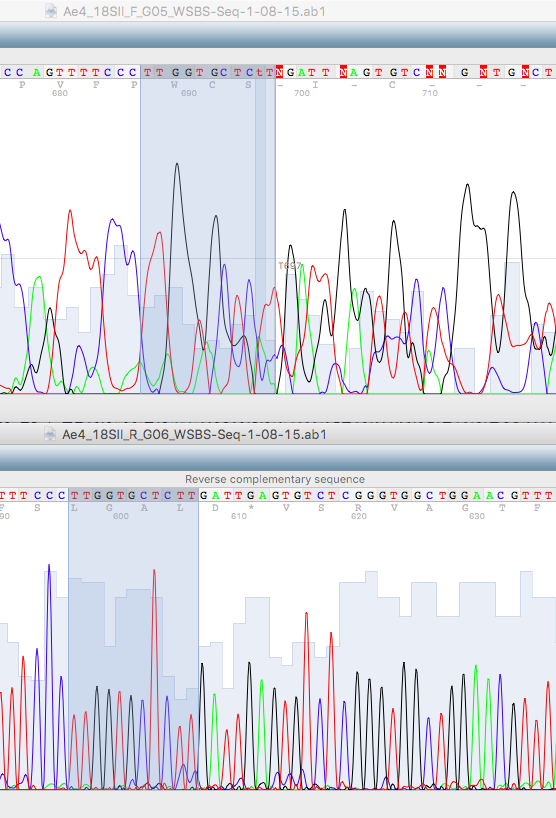
На второй картинке смещение пиков гуанина относительно друг друга (левый пик приближен к правому, правый на месте относительно остальных) вызывает проблемы в считывании синего пика, который хорошо виден и на первой, и на хроматограммах, пусть и перекрывается черным пиком. Было принято решение наличие этого пика игнорировать, ведь во второй хроматограмме его нет (только вполне конкретный одинарный черный пик).
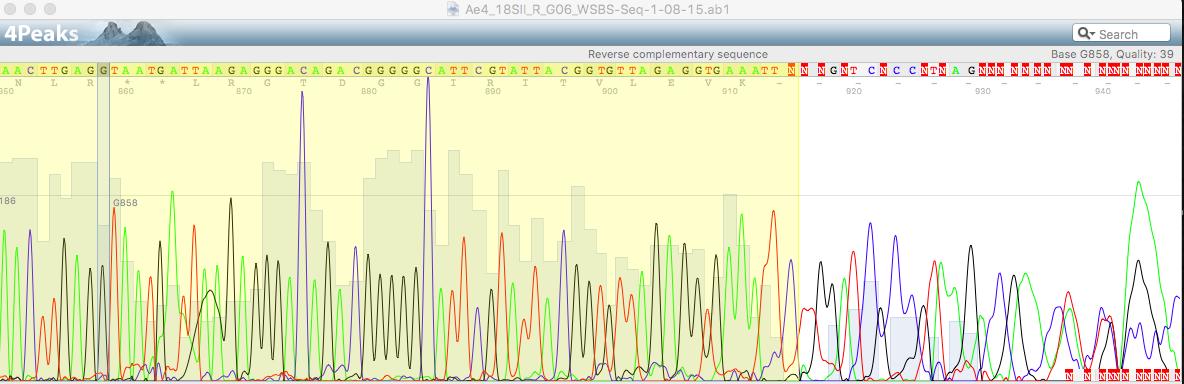
На третьей картинке очередное возникновение лишней буквы из-за смещенности/ширины пиков, но уже во второй последовательности. Первая же хроматограмма в данном случае достаточно четкая, пики высокие, узки, именно поэтому букву я считаю лишней, вызваной смещенностью широких синего и красного пиков второй хроматограммы друг относительно друга
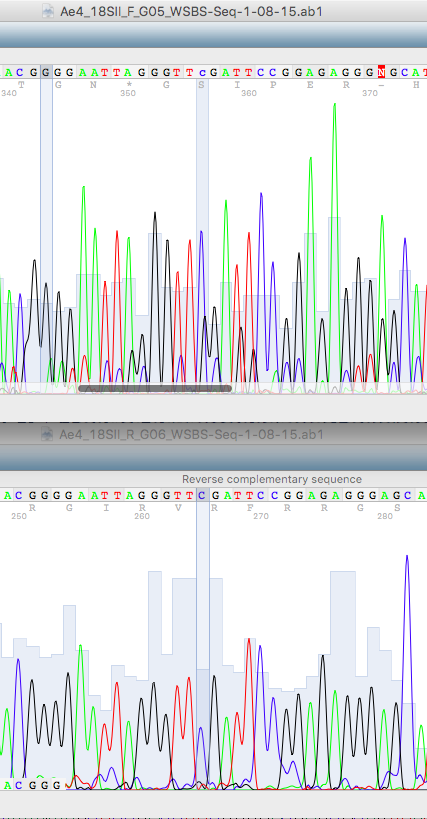
На этой картинке очень явно проиллюстрирован пункт три, но погрешностей он не вызывает. Однако N-буква возникла в том месте, где у первой хроматограммы черный пик был низким и почти сливался со своим красным шумом, а на второй черный пик - высокий, занимает по ширине два небольших синих шума, из чего я делаю вывод, что в этом месте должна находиться буква g
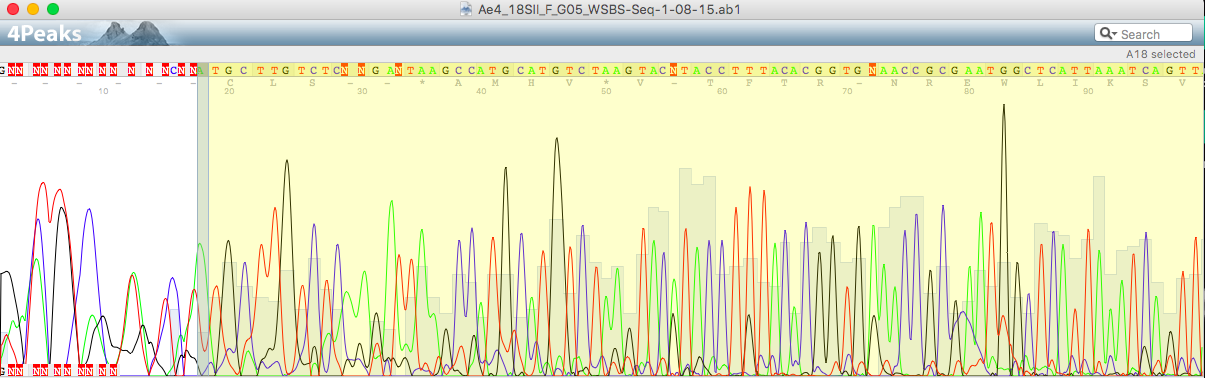
2. Привести пример нечитаемого фрагмента хроматограммы
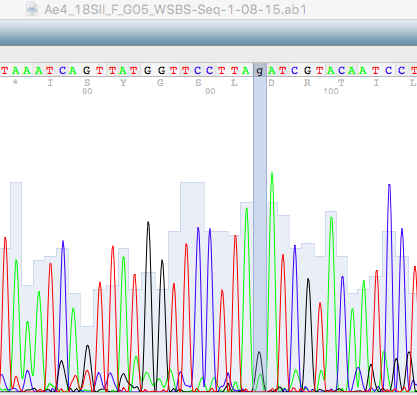
В данном упражнении нужно было описать нечитаемый участок хроматограммы. В качестве примера нечитаемой хроматограммы взят
исходный файл Ae4_18SII_R_G06_WSBS-Seq-1-08-15.ab1
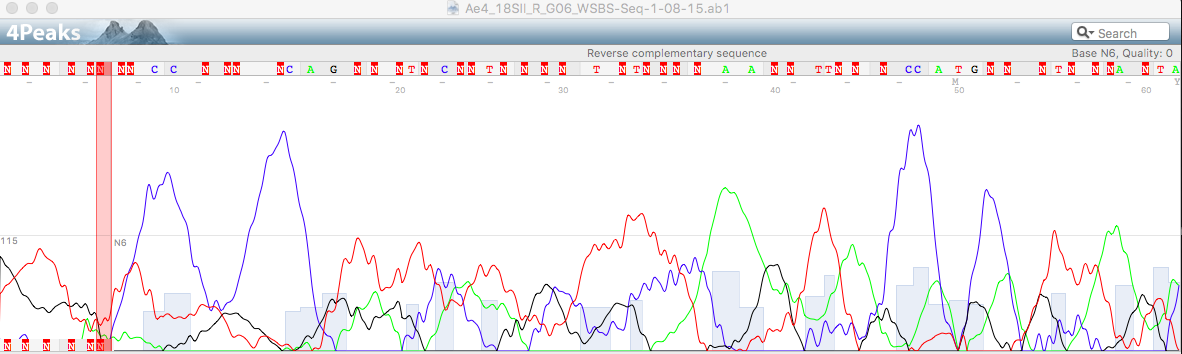
Последовательность выглядит так, как будто она загрязнена другой ДНК, так как пики наслаиваются друг на друга. Более того, пики размытые, широкие и непериодичные. Непонятно, как отличить сигнал от шума или размытой краски. Сигналы перекрываются и находятся на одном уровне с шумами, что не позволяет распознать нуклеотиды.







 _
_





