Построение и анализ дерева, содержащего паралоги
Протеомы выбранных бактерий (список представлен в таблице ниже) перенесены в один файл - all_bact.fasta с помощью команды:
cat file1.fasta >> file2.fasta
Отобранные бактерии
| Название | Мнемоника |
| Proteus mirabilis | PROMH |
| Saccharophagus degradans | SACD2 |
| Acidiphilium cryptum | ACICJ |
| Paracoccus denitrificans | PARDP |
| Roseobacter denitrificans | ROSDO |
| Bartonella henselae | BARHE |
| Agrobacterium fabrum | AGRFC |
| Rhizobium meliloti | RHIME |
Файл с протеомами преобразован в базу данных:
makeblastdb -in all_bact.fasta -dbtype prot
Файл с протеомами преобразован в базу данных:
blastp -db all_bact.fasta -query P0A6H1.fasta -out term4pr3.txt -evalue 0.001 -outfmt 7
Из всех найденных белков выбраны, имеющий процент идентичности больше 35, их список приведен в файле: term4pr3.txt
Был создан фаста-файл, содержащий последовательности всех этих белков: allhom.fasta
Последовательности этих белков были выровнены с помощью программы:
muscle -in allhom.fasta -out allhommus1.fasta
Далее я использовала программу MEGA для построения и анализа дерева, содержащего паралоги
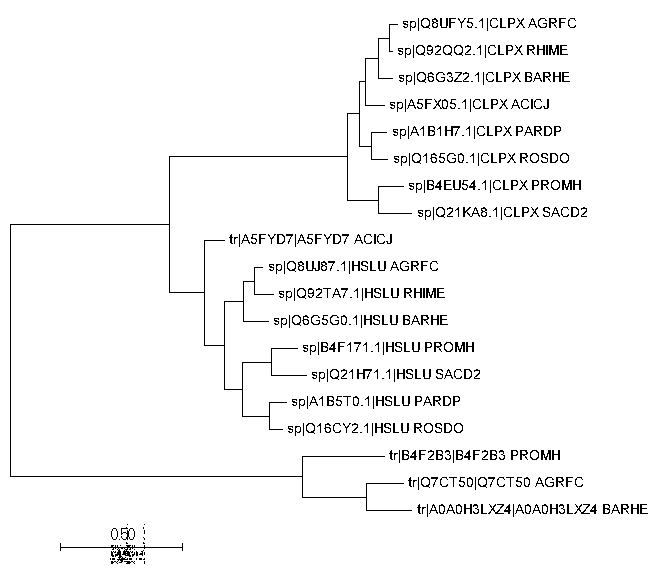
Примеры ортологов (произошло разделение путей эволюции белков в результате видообразования):
1. Белки с мнемоникой CLPX_*
2. Белки с мнемоникой HSLU_*
Примеры паралогов (произошла дупликация гена):
1. Белки CLPX_PROMH, HSLU_PROMH, B4F2B3_PROMH
2. Белки CLPX_AGRFC, HSLU_AGRFC, Q7CT50_AGRFC
© Grigorjeva Masha