Практикум 6. Взаимодействия. Субстратная специфичность
Задание 1. PoseView
PDB_ID: 5RE7 (структура протеазы Sars-Cov-2 с лигандом)
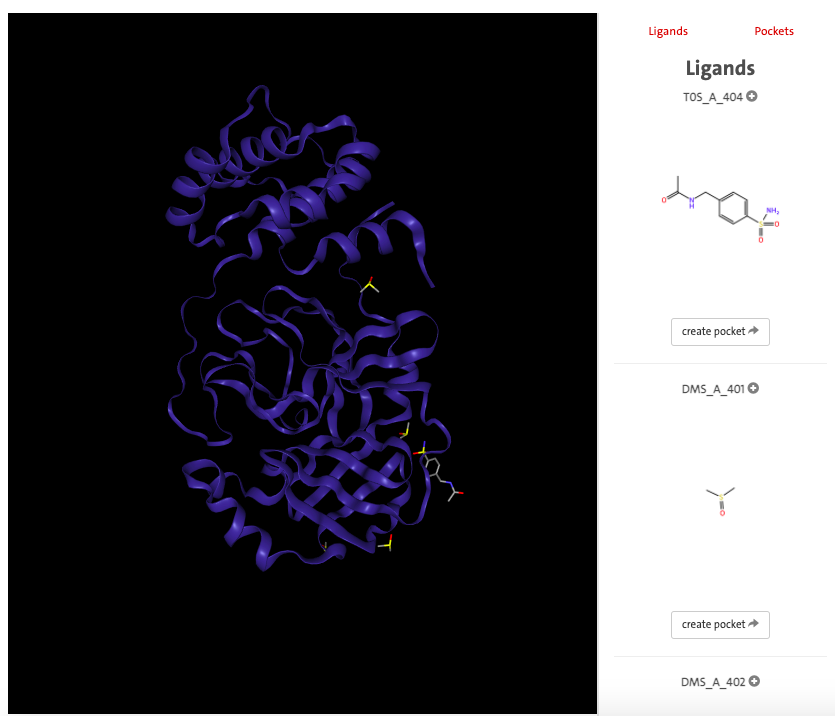
Рис 1. Структура 5RE7 и лиганды от ProteinsPlus - Structure-Based Modeling Support Server
Была сгенерирована 2D диаграмма взаимодействий с помощью Poseview на сайте Proteins.Plus.
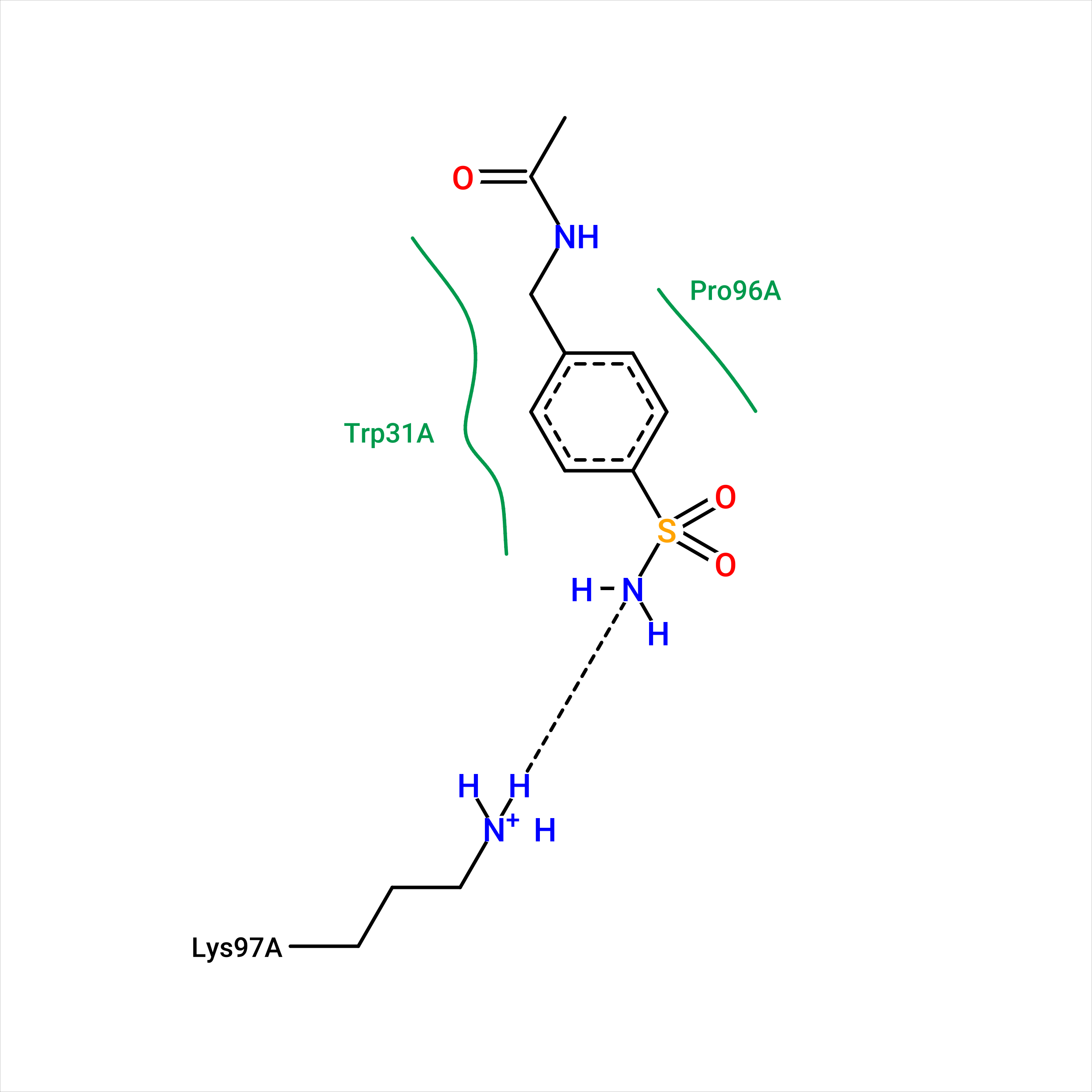
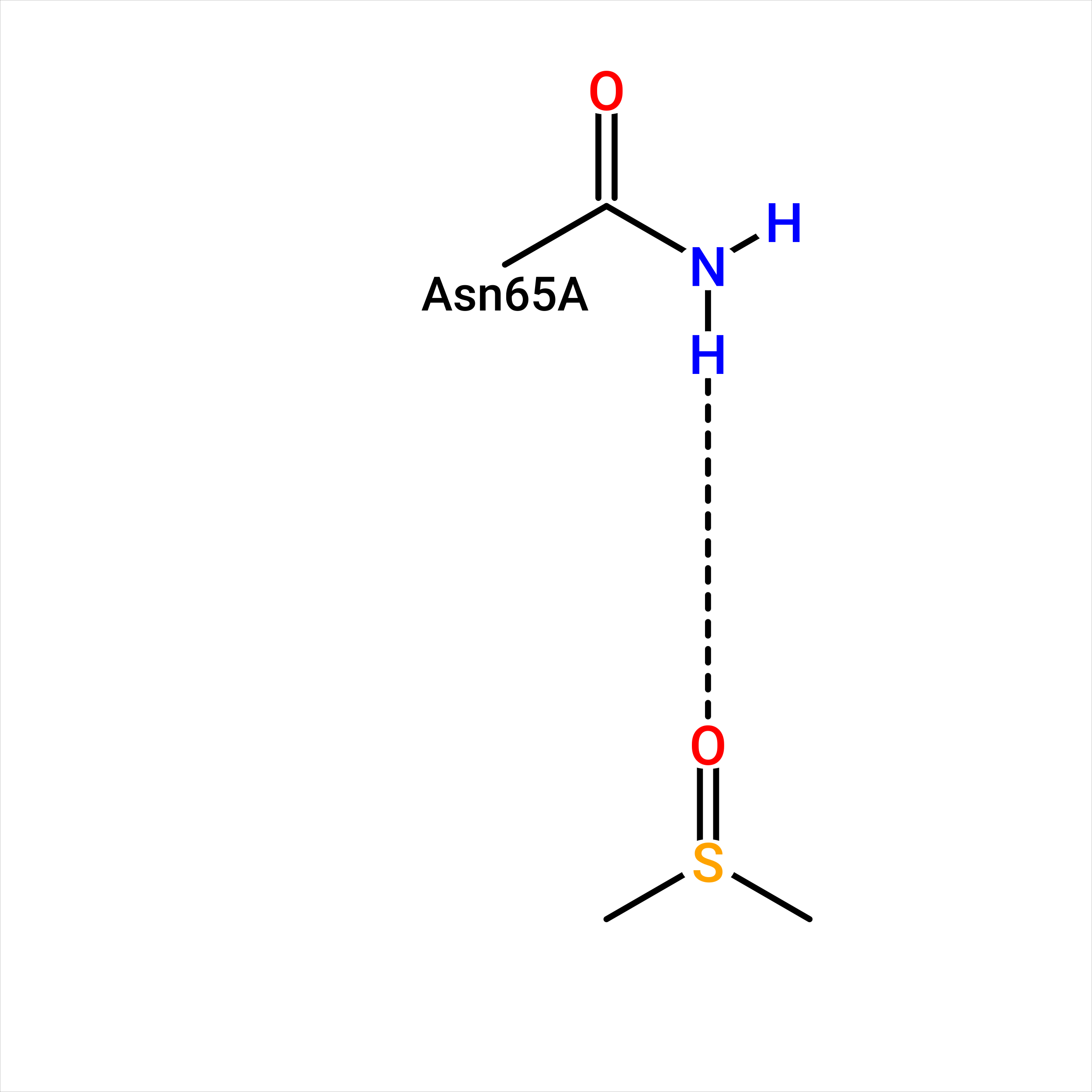
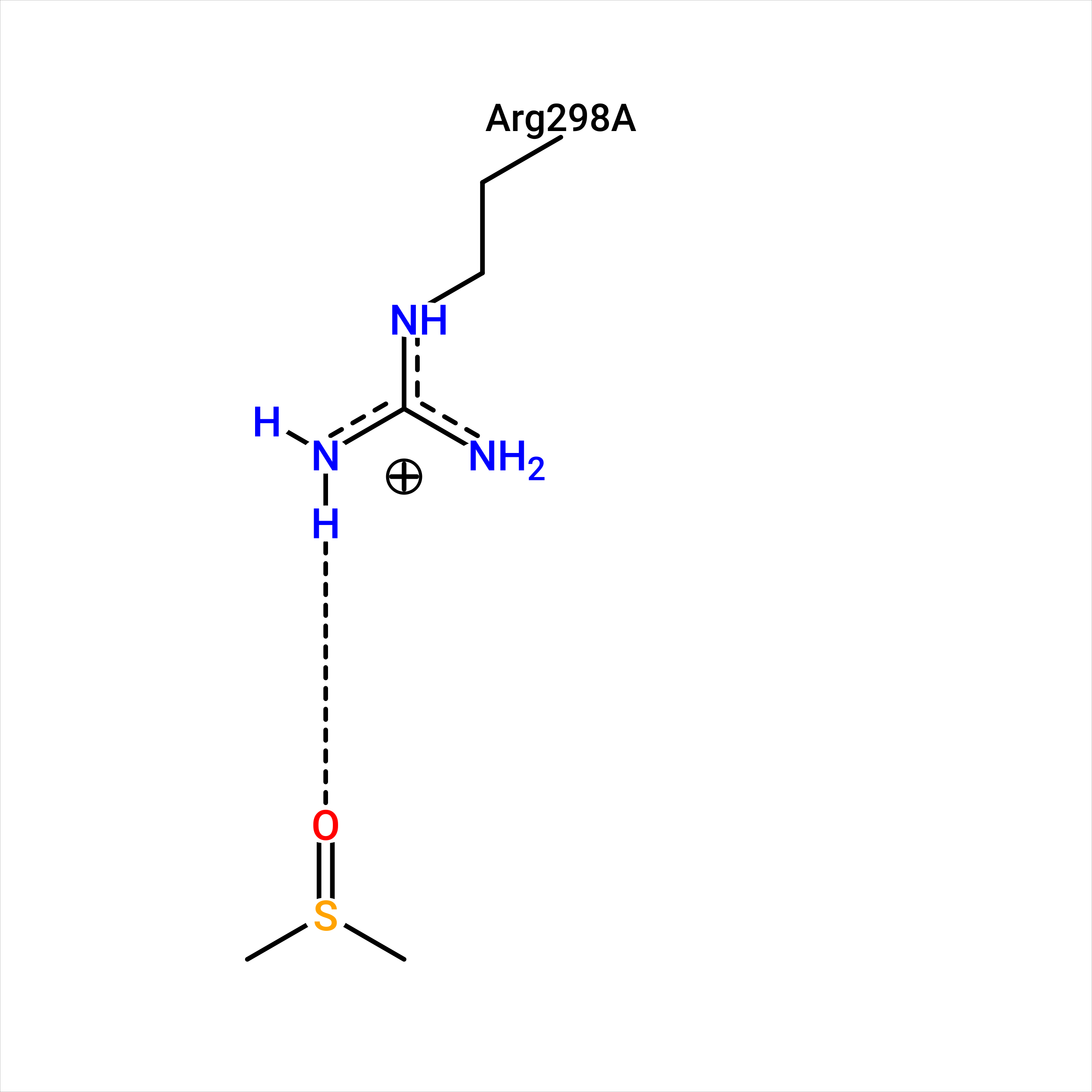
Рис 2. 2D диаграммы для лигандов TOS_404, DMS_402 и DMS_403.
Получилось так, что для лиганда DMS_403 я взаимодействия в прошлый раз не нашла, а оно есть с аргинином 298.
Для структуры DMS_402 я предполагала взаимодействия с гистидином 64 и аспарагином 65. Последнее подтвердилось на диаграмме (рис 2).
Лиганд TOS_404 оказался самым запутанным в прошлый раз. Для него я установила взаимодействие с лизином 97, которое явно видно на диаграмме. Еще я отмечала, что пусть связи с пролином 96 нет, я посчитала его важным. Он так же отображен на диаграмме, как и не упомянутый мною ранее триптофан 31.
Затем я на всякий случай запустила программу PlexView.
Рис 3. 2D диаграмма версия 2 для лиганда TOS_404.
Какие я могу сделать выводы?
Все 3 способа проанализировать связи лиганда показали различный результат. Больше всего связей выявила программа PlexView, меньше всего - PoseView. Главная связь во всех трех анализах осталась неизменной - это связь лиганда TOS-404 с Лизином 97. Помимо него при первом анализе я выделила Аланин 70, Пролин 96 и Валин 73. Интересно, что и эти три остатка, и могие из тех, которые я сочла не столь важными, но все же вероятными (видно в сессии с того занятия, ссылка на скачивание на странице практикума 1) оказались в PlexView отображены.
По имеющимся у меня данным я бы скорее доверяла результатам программы PoseView.
Задание 2. PyMol mutagenesis
0017 E 7 0076 C 5
Комплекс антитела с пептидным антигеном. Одну позицию антигена заменили на глицин. Используя возможности инструментария Wizard > Mutate нужно былое выяснить, каким был исходный остаток на этой позиции.
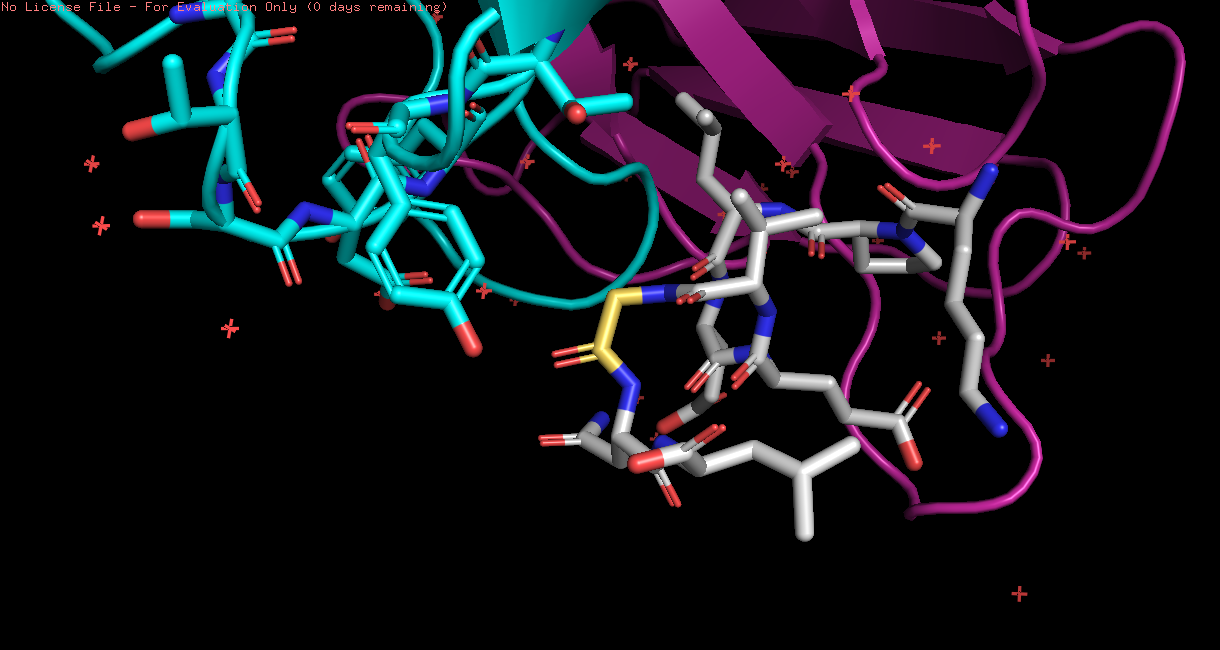
Рис 4. Комплекс 0017: серым выделена цепь с заменой, замена выделена оранжевым цветом.
В первом комплексе замена была на GLY 7(рис 4). На этом примере я решила привести подробные таблицы для каждой попытки замены, для комплекса 0076 я приведу только выводы.
| Аминокислота | Количество ротамеров | вероятность встретить (%) |
| ALA | 0 | 0 |
| ARG | 21 | от 1.1 до 14.3 |
| ASN | 10 | от 1.1 до 39.7 |
| ASP | 8 | от 1.0 до 40.5 |
| CYS | 3 | от 2.1 до 64.0 |
| GLN | 14 | от 1.5 до 23.2 |
| GLU | 14 | от 1.5 до 21.2 |
| HIS | 8 | от 1.2 до 34.2 |
| ILE | 6 | от 1.3 до 51.1 |
| LEU | 3 | от 1.4 до 92.8 |
| LYS | 16 | от 0.1 до 33.5 |
| MET | 14 | от 1.1 до 30.0 |
| PHE | 4 | от 6.0 до 65.7 |
| PRO | 2 | от 11.0 до 89.1 |
| SER | 2 | от 17.8 до 81.4 |
| THR | 3 | от 1.4 до 96.1 |
| TRP | 9 | от 1.0 до 46.8 |
| TYR | 4 | от 1.7 до 68.2 |
| VAL | 3 | от 19.1 до 57.8 |
Из таблицы представленности все оказалось очень непонятно, поэтому я начала рассматривать просто каждый вариант отдельно. Вот, что у меня получилось:
| Аминокислота | Номер ротамера | Стерическая напряженность | Представленность ротамера | Изображение |
| ARG | 11/21 | 30.98 | 2.3% | 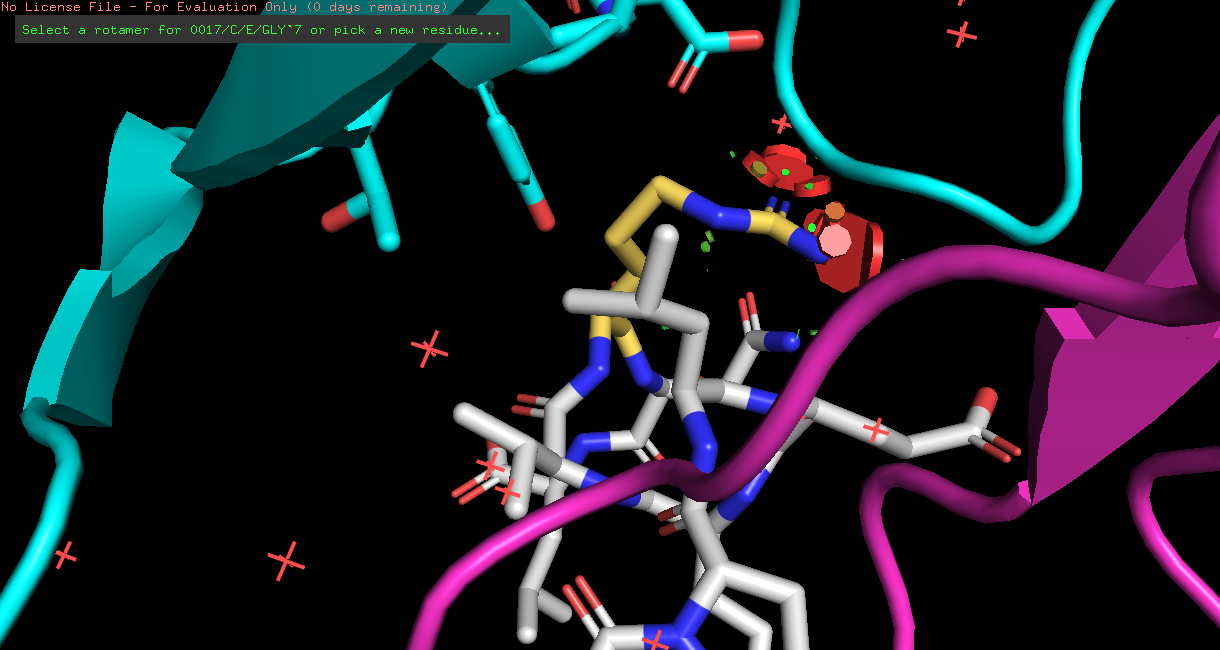 |
| ASN | 7/10 | 17.93 | 3.7% | 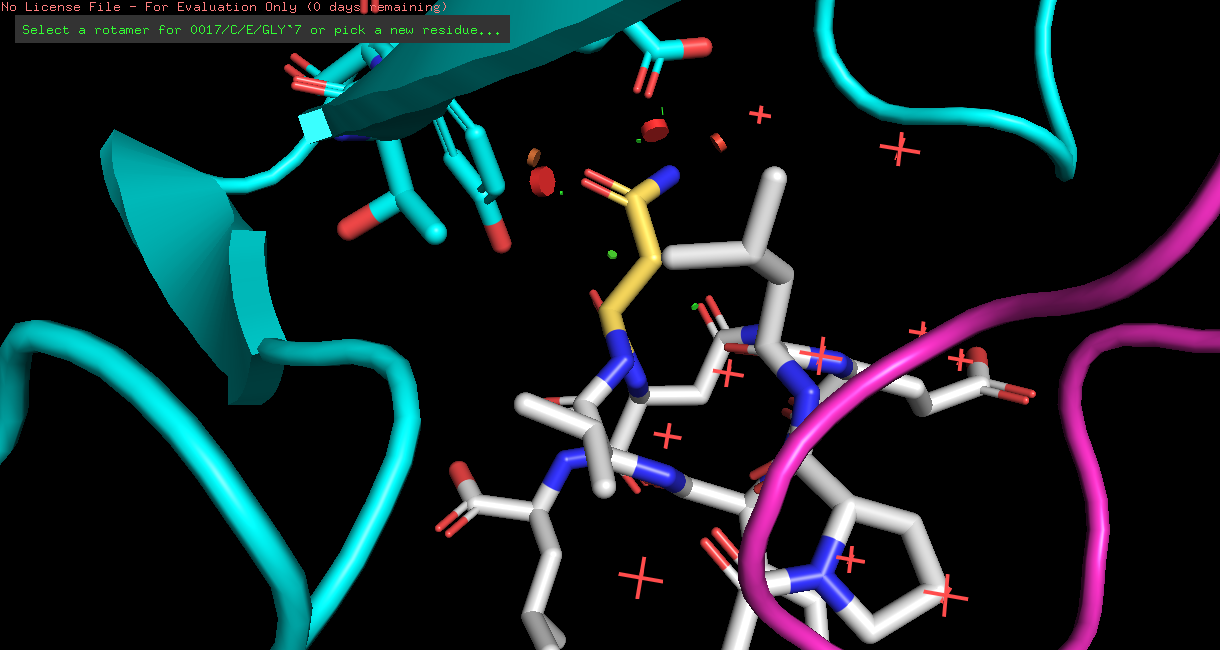 |
| ASP | 6/8 | 18.30 | 2.1% | 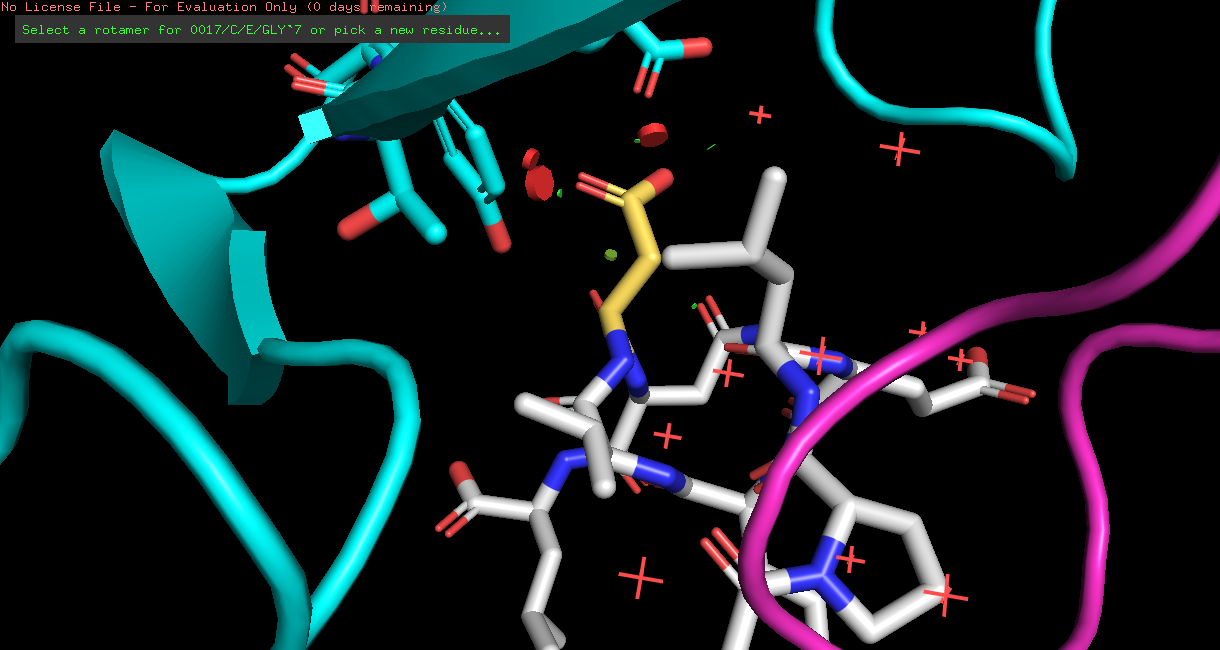 |
| CYS | 3/3 | 36.23 | 2.1% |  |
| GLN | 2/14 | 21.25 | 18.7% |  |
| GLU | 2/14 | 19.89 | 13.4% |  |
| HIS | 3/8 | 22.96 | 16.3% |  |
| ILE | 2/6 | 20.58 | 16.5% |  |
| LEU | 1/3 | 24.05 | 92.8% |  |
| LYS | 1/16 | 22.72 | 33.5% |  |
| MET | 5/14 | 17.46 | 5.4% |  |
| PHE | 2/4 | 24.77 | 14.8% | 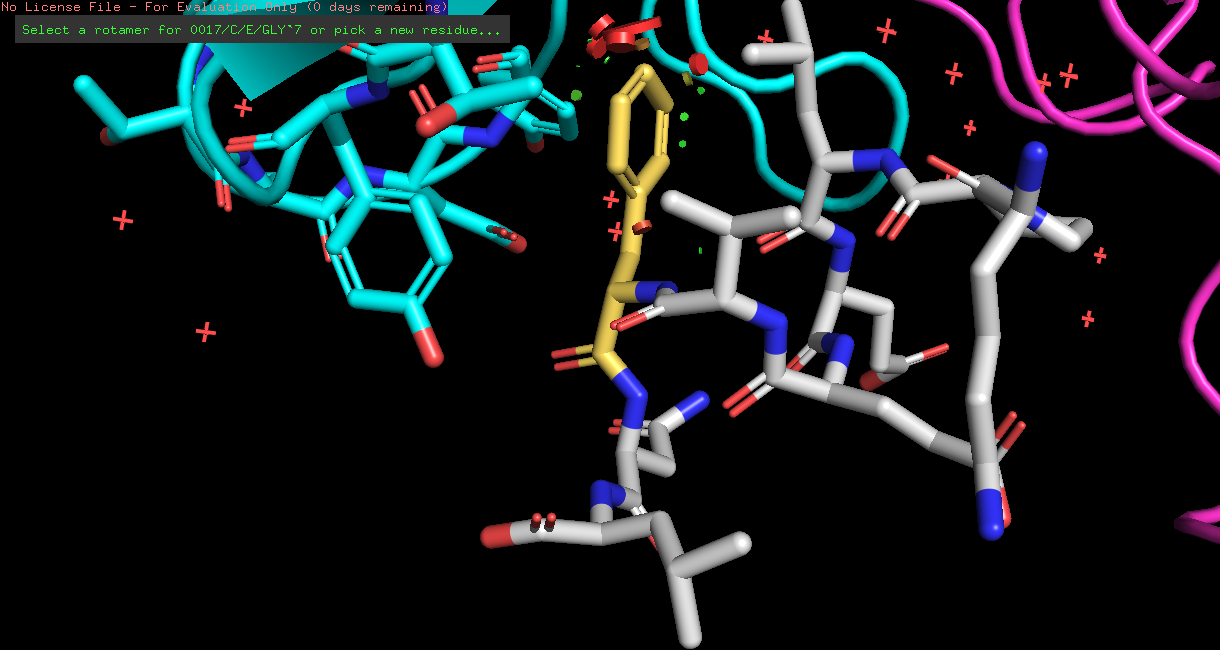 |
| THR | 2/3 | 21.04 | 2.5% | 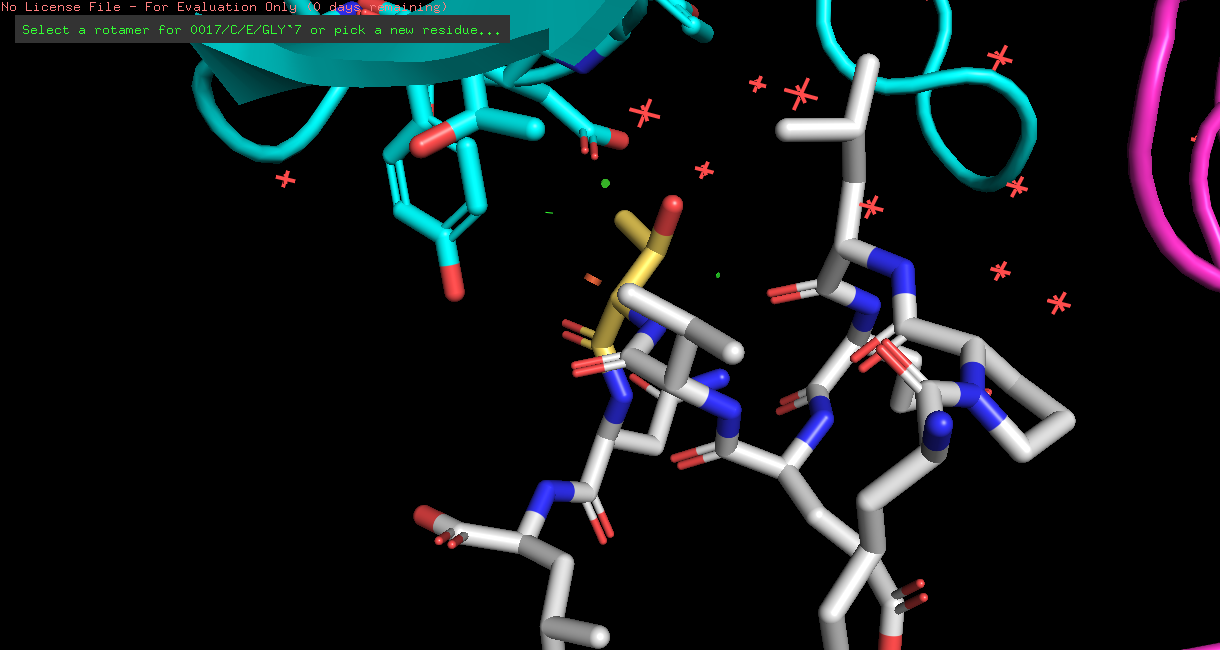 |
| TRP | 1/9 | 42.25 | 46.8% | 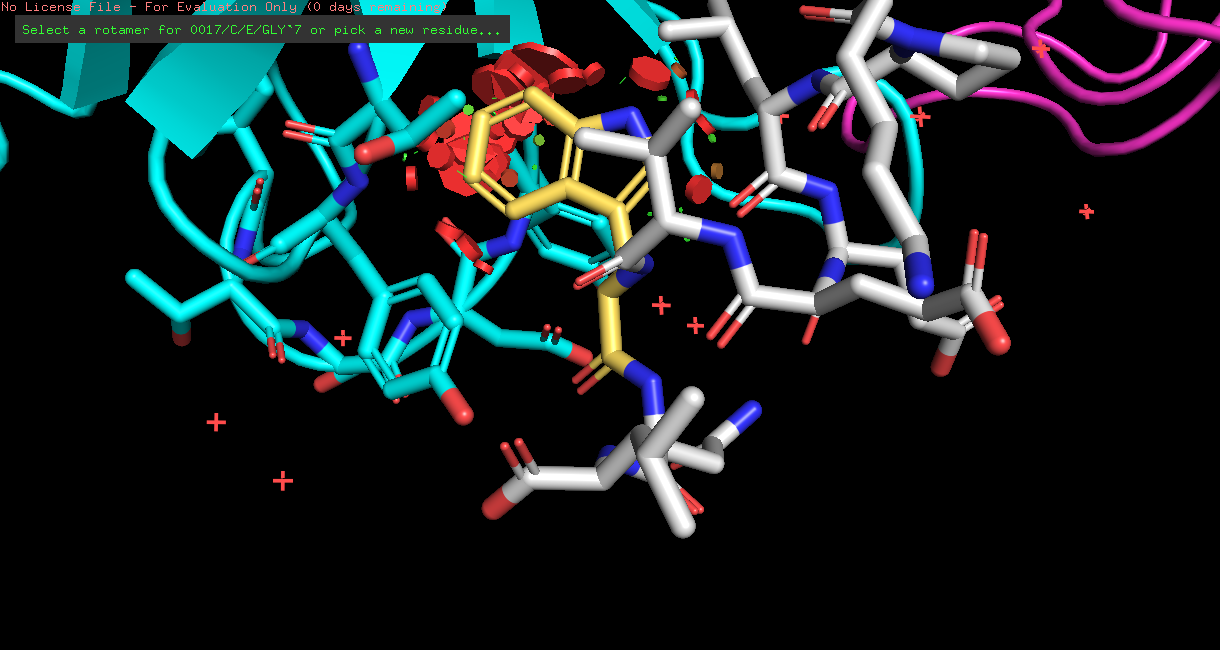 |
| TYR | 1/4 | 36.08 | 68.2% | 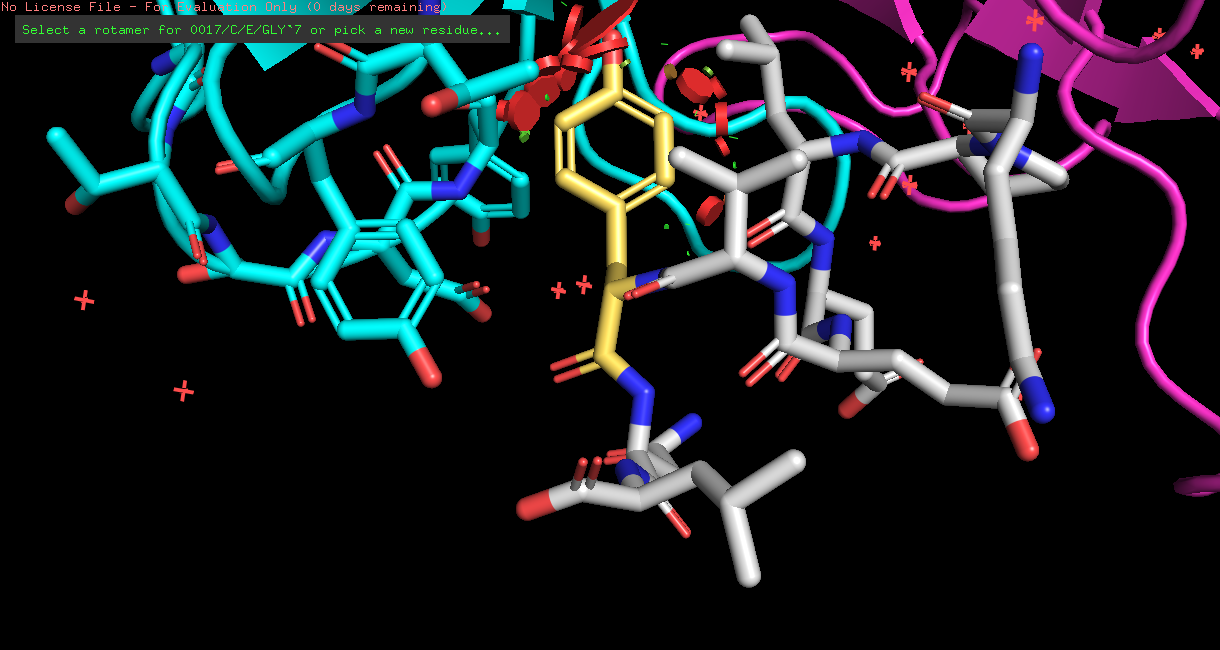 |
| VAL | 2/3 | 22.05 | 23.1% | 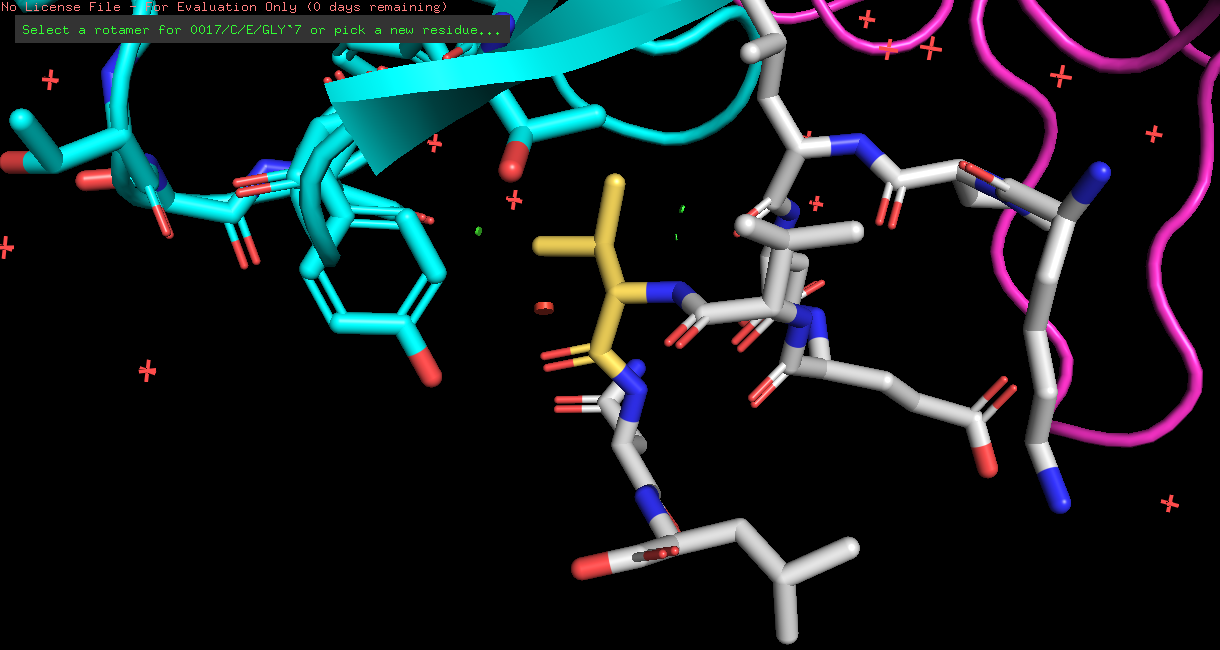 |
Из последней таблицы я делаю вывод, что на этом месте находился THR. У него напряженность 21.04, и очень удобно наблюдать связи на картинке 5 (ниже).
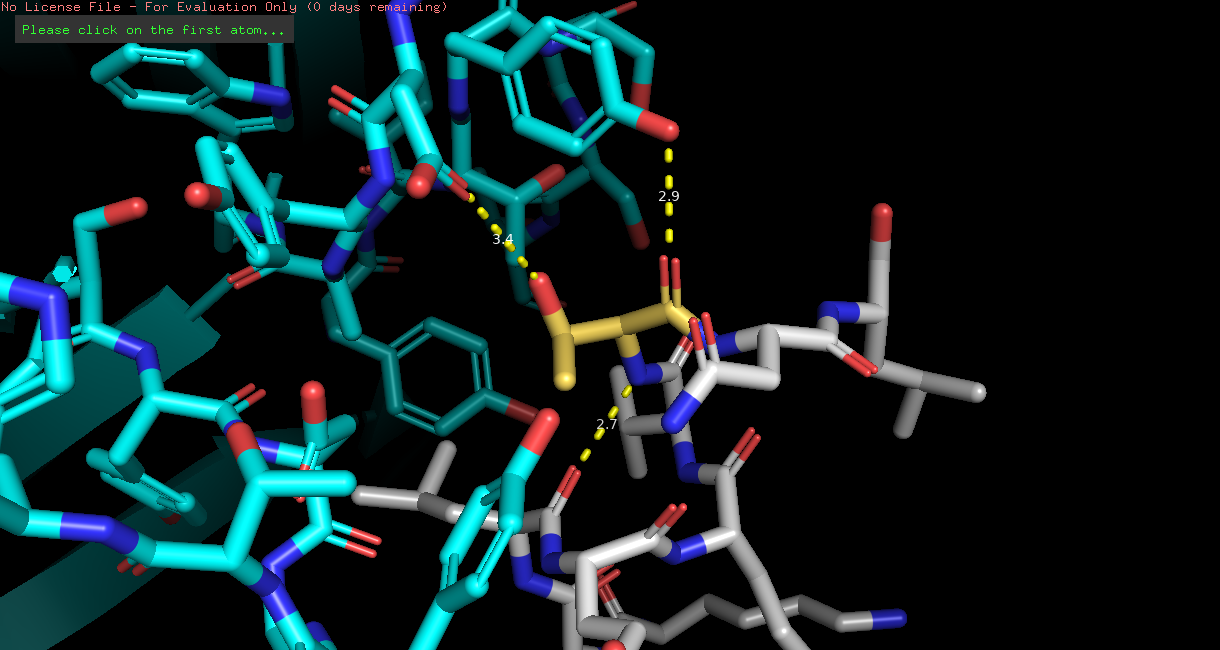
Рис 5. THR, который мне нравится.
Перейдем к комплексу 0076. Можно заметить, что в данном случае у нас есть довольно мало места для вставки нашей замены, значит остатки с кольцами, длинными структурами и тд точно нам бы не подошли.
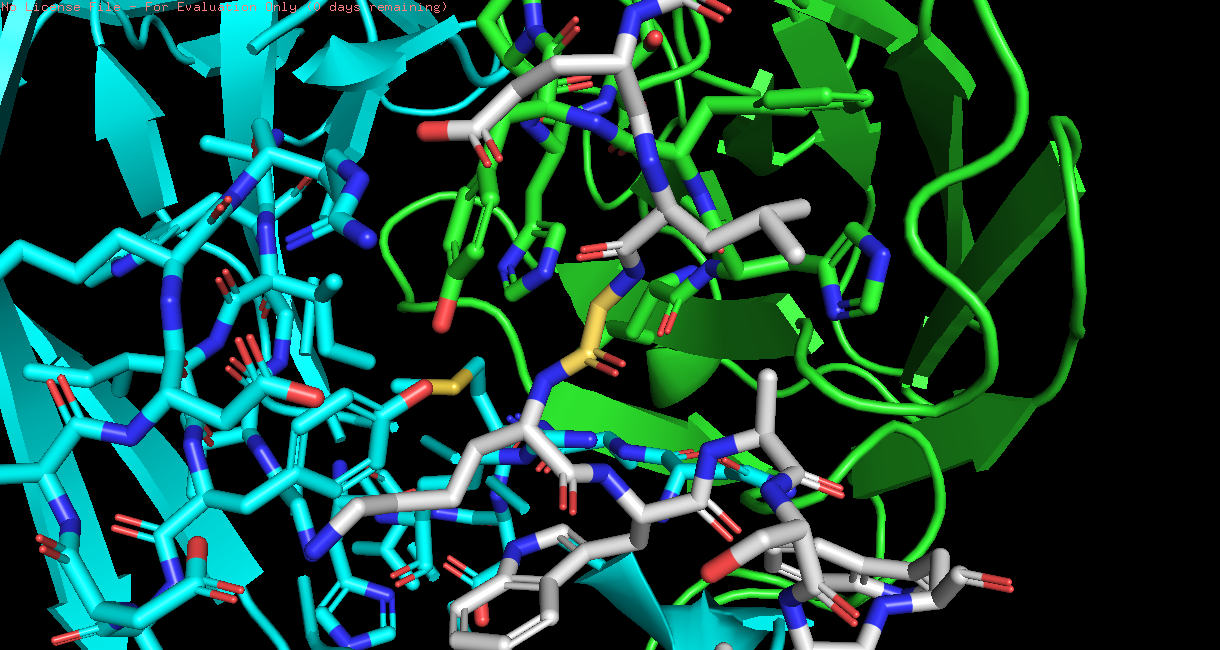
Рис 6. Комплекс 0076: серым выделена цепь с заменой, замена выделена оранжевым цветом.
Больше всего мне здесь показались подходящими ASP, CYS и SER, но cys меня разочаровал, так как там все не очень хорошо со связями.
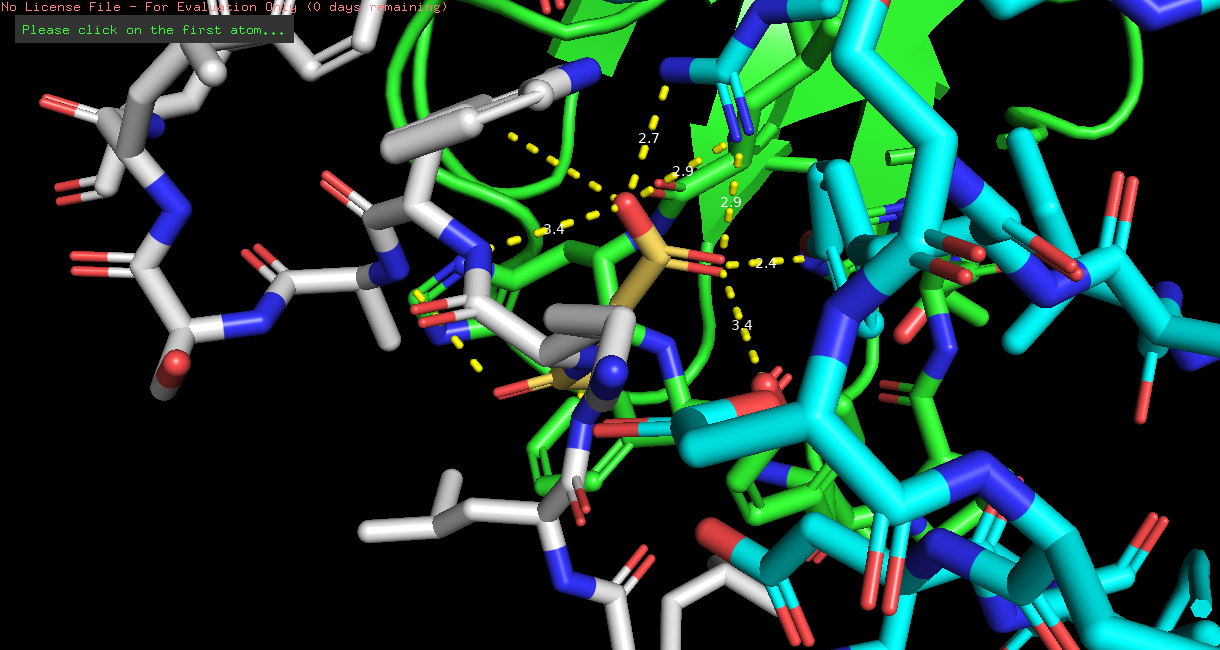
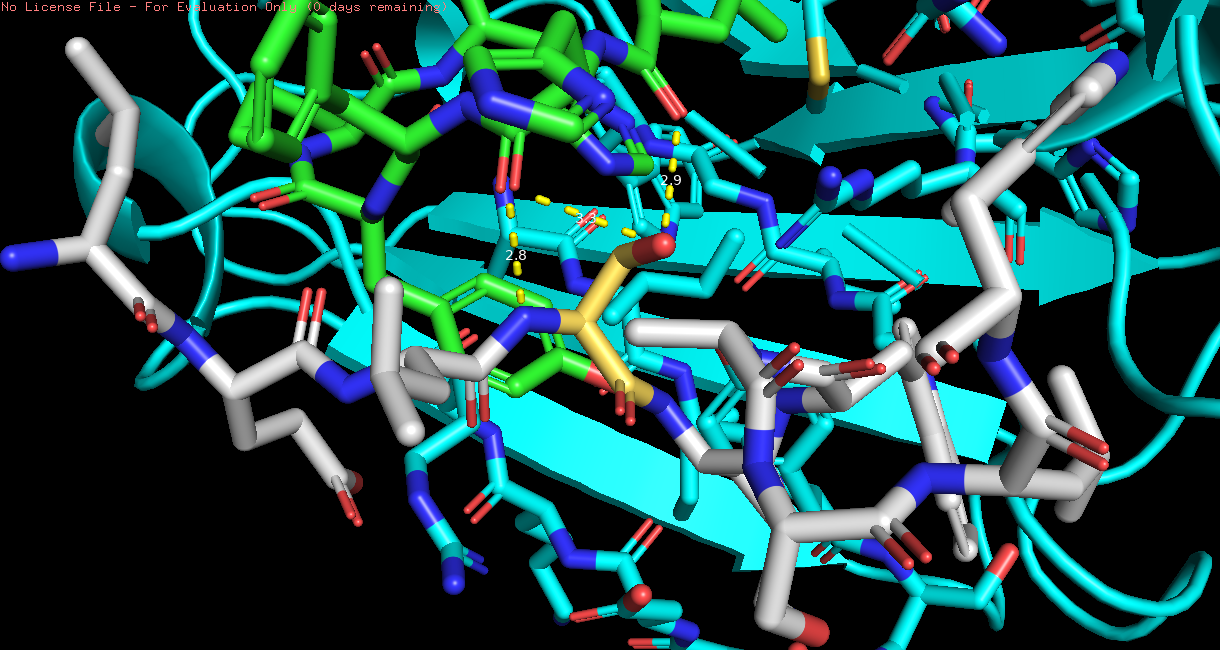
Рис 7. ASP и SER, которые мне понравились [тоже].
На рисунках видно, что SER создает связи только с одной из цепей (с зеленой), в то время как ASP заходит глубже в своеобразный "карман" и связывается с обеими цепями. Поэтому я сделаю ставку на ASP.
© Grigorjeva Masha