Практикум 1. Pymol, электронная плотность
Задание 1. Pymol
На данном этапе нужно было обработать с помощью Pymol структуру белковой молекулы (PDB_ID: 5RE7) и показать наиболее значимые взаимодействия с лигандом.
PanDDA analysis group deposition - Crystal Structure of SARS-CoV-2 main protease in complex with Z30932204

Рис 1. Лиганды в файле 5RE7.
В качестве окружения я предположительно выбрала Аланин 70, Пролин 96, Лизин 97, Валин 73 (рис. 2). PyMol не показал каких-либо связей между ними и лигандом, но мне кажется, что они могут играть роль в связывании лиганда // любых взаимодействиях с ним.
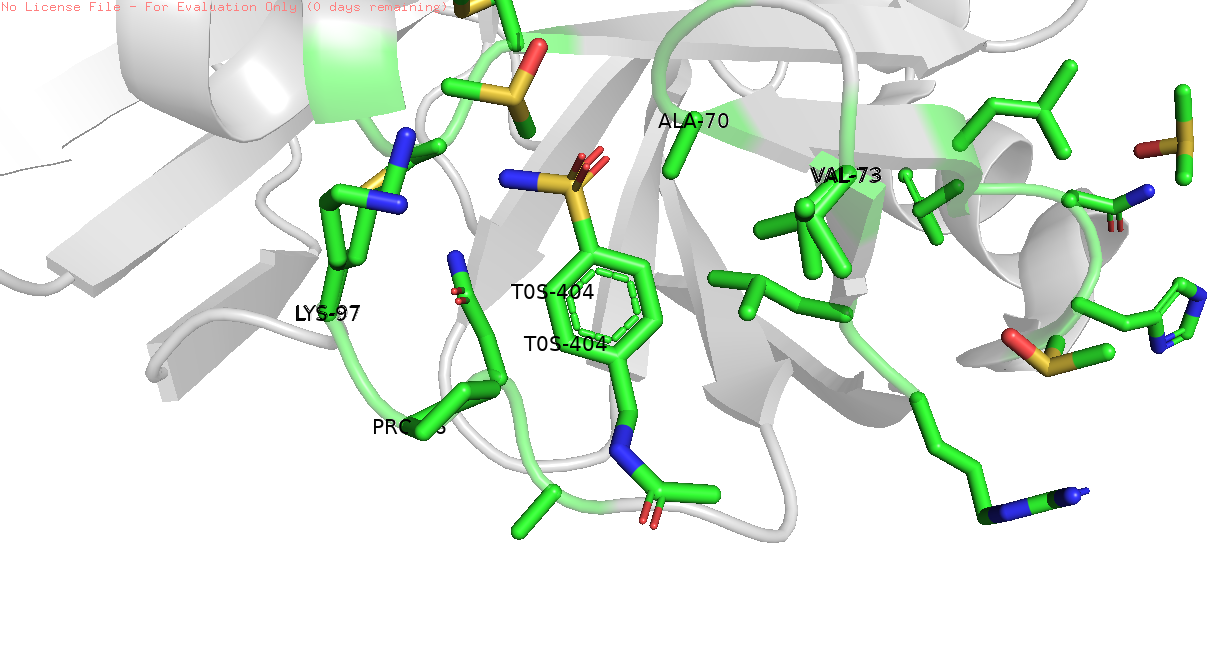
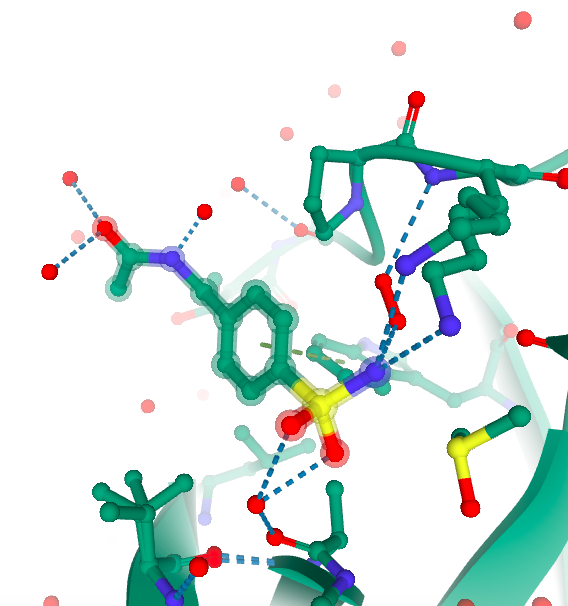
Рис 2. Лиганд TOS-404 и его окружение в текущем файле (слева) и в файле 5RE7 (справа), его связь с конформациями A и B лизина 97.
После этого я решила проверить, как связывается этот лиганд по версии PDB. Он связывается с лизином 97 (рис. 2).
Потом я решила проверит другой лиганд. Он был представлен в 3х местах стурктуры, при этом связывание я смогла найти только для одного из них (рис. 3) - он связывается с Гистидинов 64 и Аспарагином 65.
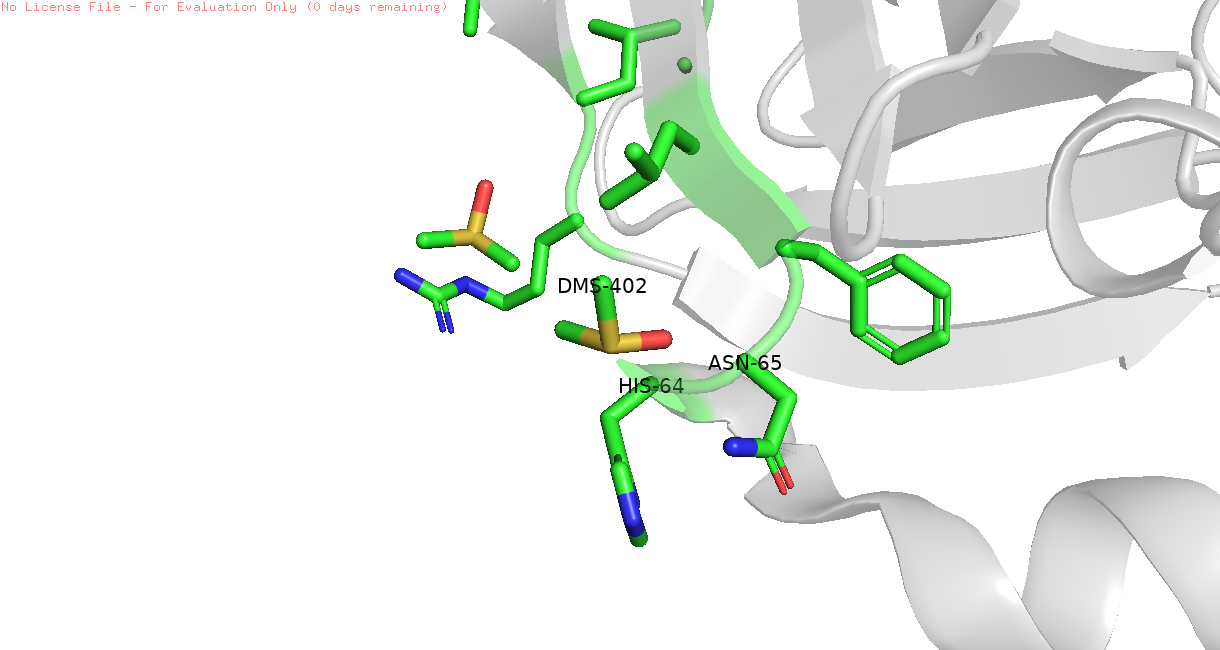
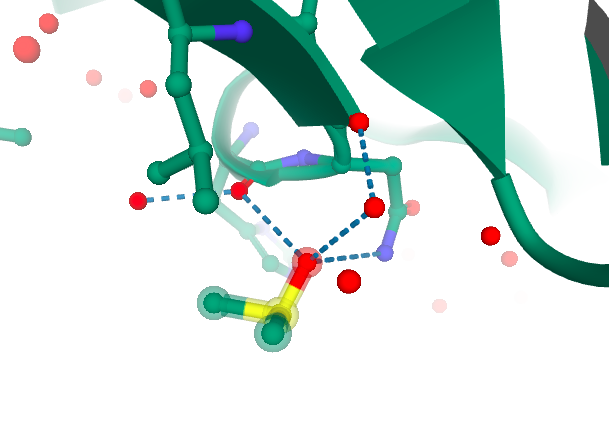
Рис 3. Лиганд DMS-402 в файле 5RE7 и его связь с гистидином 64 и аспарагином 65: на pdb (слева) и в нашей структуре (справа).
Ссылка на сессиЮ
Задание 2. ЭП: хорошая и плохая расшифровки
Дано два PDB ID: 3QPA и 1XZD. Они соответствуют двум кристаллографическим расшифровкам одного и того же белка, при этом по условию их качество сильно различается.
Update: замена структуры 1XZD на структуру 1OXM.
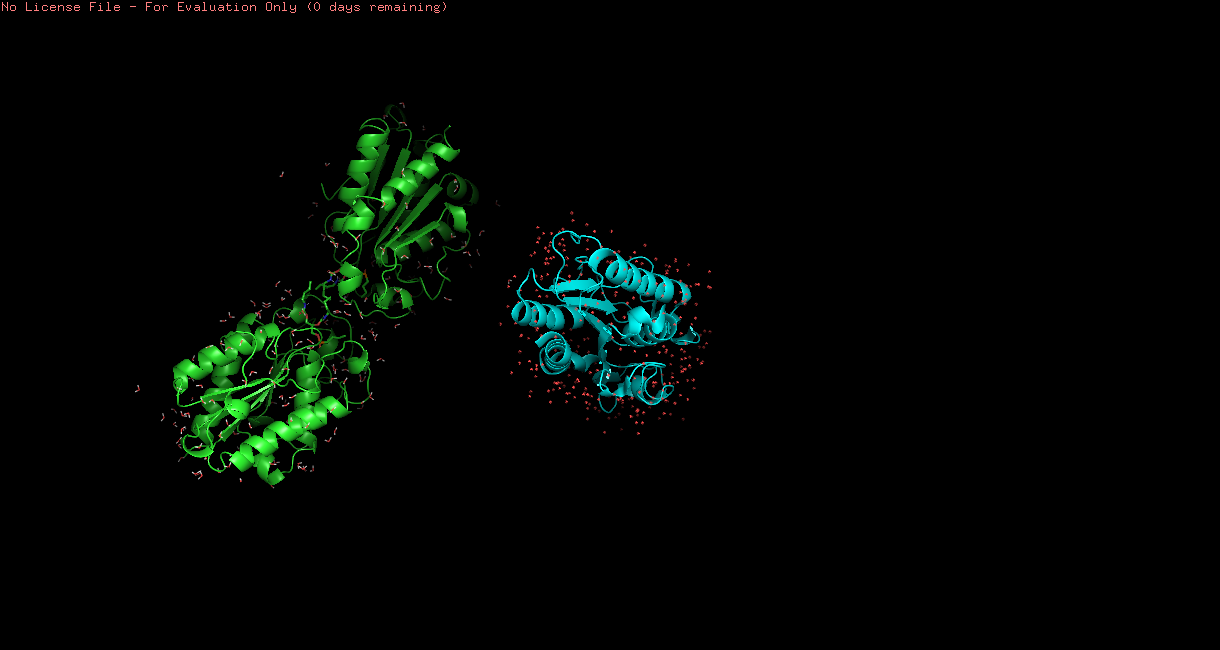
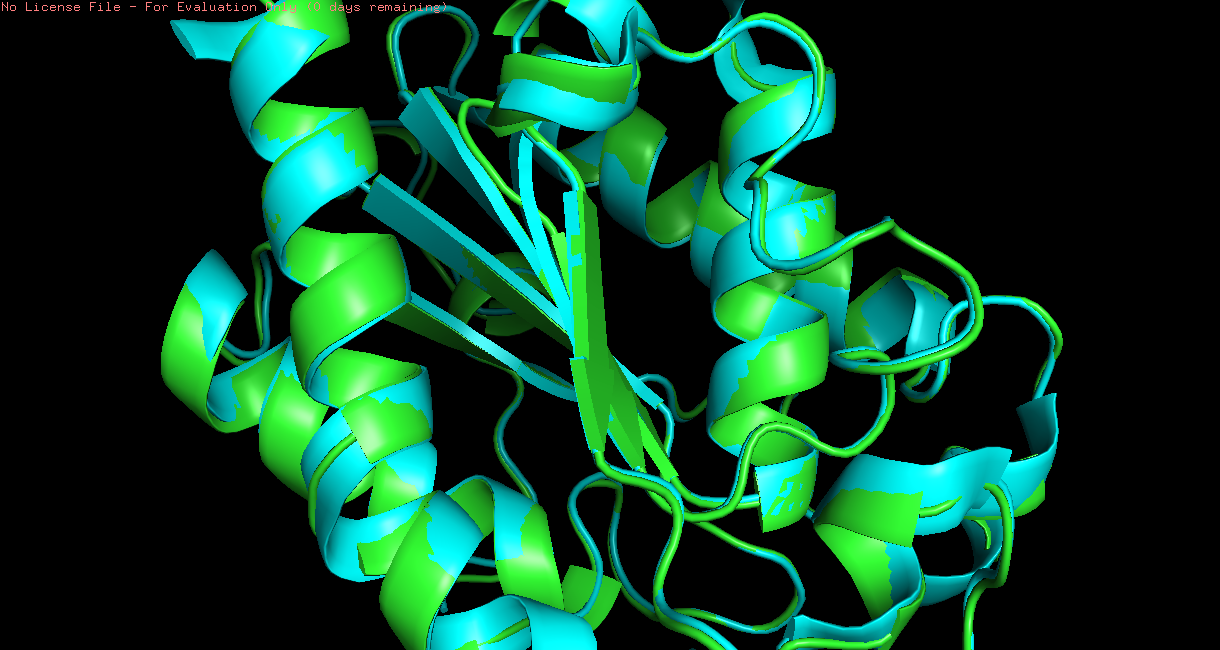
Рис 4. Структуры 3QPA (зеленая) и 1XZD (голубая) до (слева) после (справа) выравнивания. .
Итак, структуры оказались совершенно не похожи друг на друга (рис.4). Для того, чтобы анализировать одинаковый остов, я выравняла их друг на друга с помощью команды super. После этого они оказались очень похожи друг на друга, просто в одной из структур присутствовала еще одна часть структуры, которую мы затрагивать не будем. Таким образом был выбран остов остатков с 147 по 163.
Я использовала mesh (carve=1.5), чтобы сравнить качество структур.
select loop1, 1oxm and chain A and resi 147-163
isomesh 1OXM_mesh, 1oxm_2fofc, 2, loop1 and backbone, carve=1.5
select loop2, 3qpa and chain A and resi 147-163
isomesh 3QPA_mesh, 3qpa_2fofc, 2, loop2 and backbone, carve=1.5
Для 3QPA электронная плотность гораздо лучше согласуется с положениями отдельных атомов (рис. 5). Я предположила, что это - показатель лучшего разрешения в структуре.Поэтому я проверила себя по данным, выложенным в RCSB PDB: там оказалось, что для 3QPA разрешение 0.85 ангстр, а для 1OXM - 2.3 ангстр.
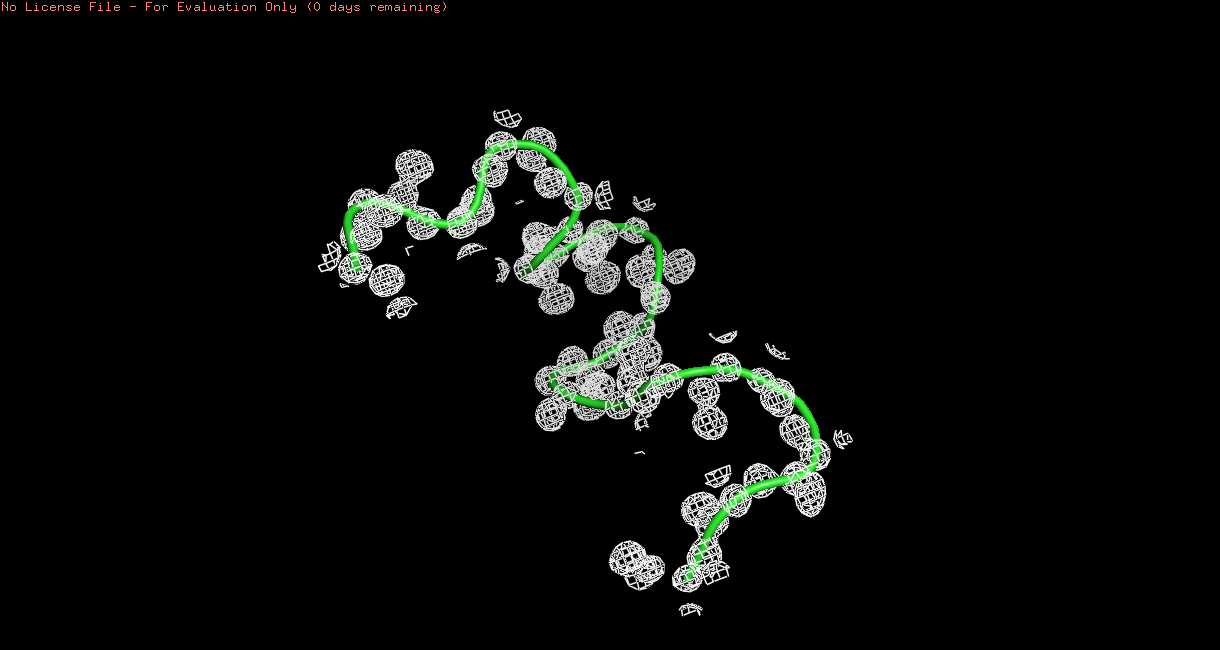
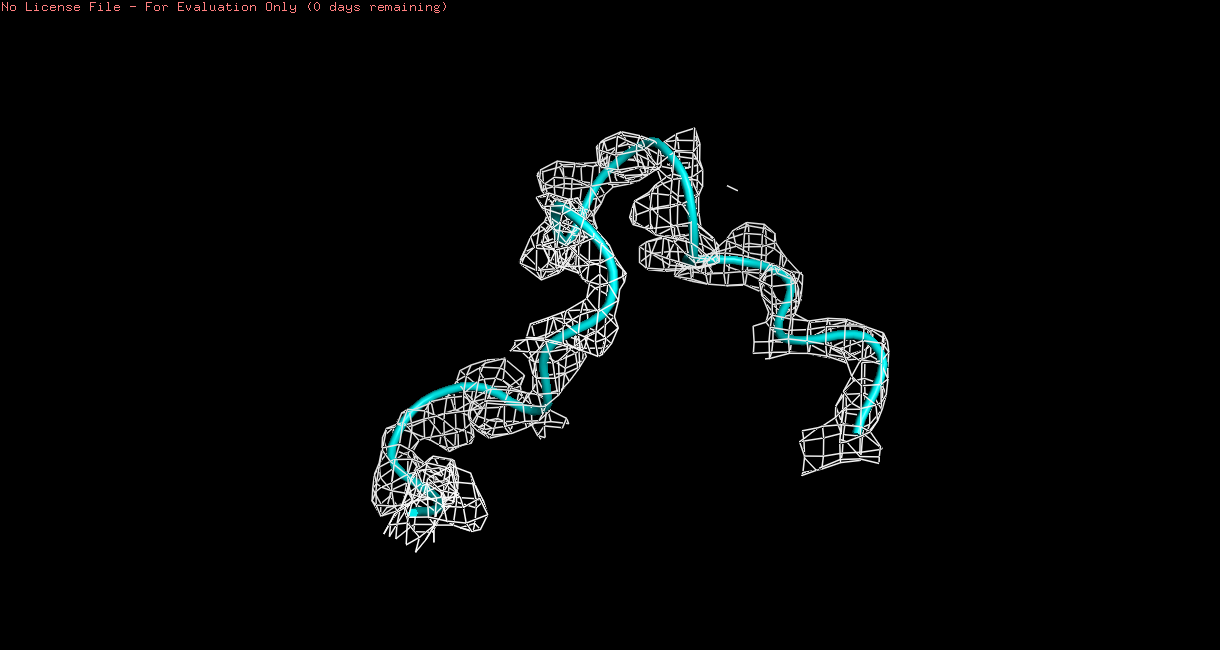
Рис 5. Петли в структурах 3QPA (зеленая) и 1XZD (голубая) после использования mesh с параметром carve = 1.5
Задание 3. ЭП и положение в структуре
Вернемся к PDB из задания 1. Была построена карта эп остова всего белка с тремя разными уровнями подрезки, снова был выбран параметр carve = 1.5
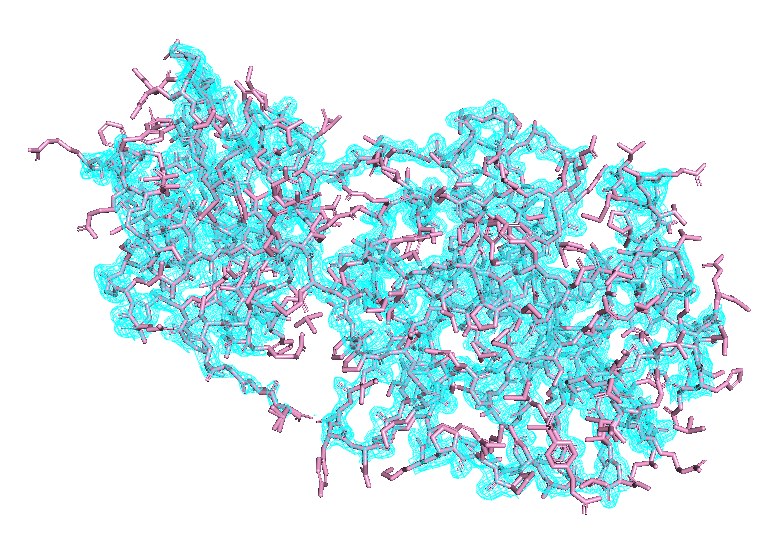


Рис 6. Карта ЭП вокруг 5RE7 после использования mesh с параметром carve = 1.5. Уровни 1, 2 и 3 слева направо.
По мере увеличения уровня покрытие падает. При переходе 1 -> 2 -> 3 некоторые регионы остова структуры первыми перестают быть покрытыми mesh'ем. В данном случае, чем ближе к переферии находится регион, тем быстрее он теряет покрытие. Скорее всего это объясняется плотностью электронов в области расположения атома, которая тем ниже, чем дальше от центра глобулы находится атом.
Задание 4. ЭП и типы атомов
Продолжаем работать с PDB из задания 1. Можно заметить, что даже на уровне 0.25 не весь лиганд покрыт, а с повышением уровня и вовсе покрыт все меньше и менше. Последним исчезает область возле атома кислорода, который связывается как на рис. 2.
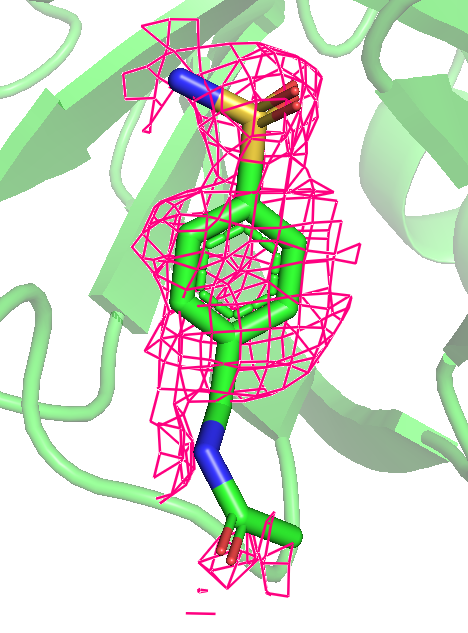
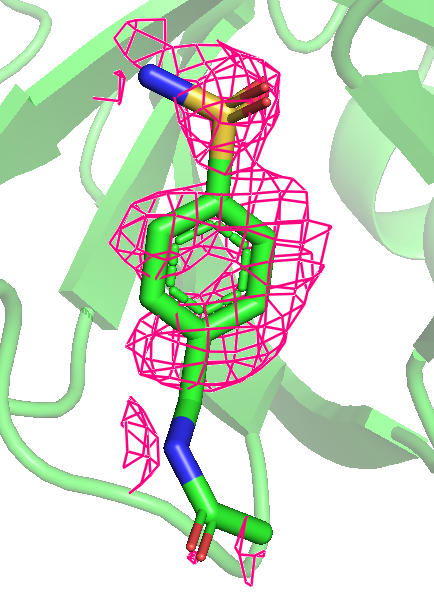
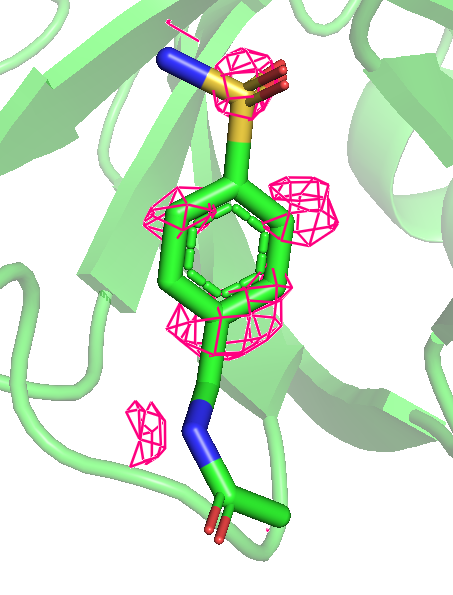
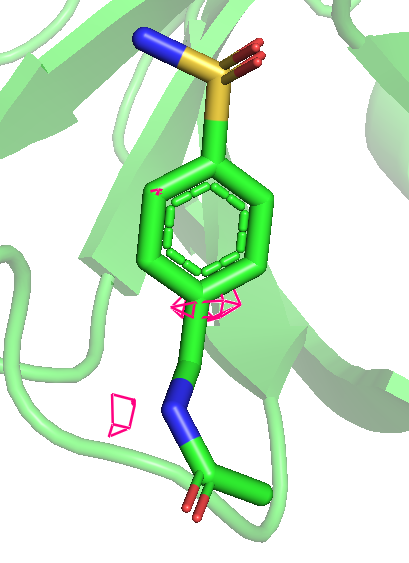
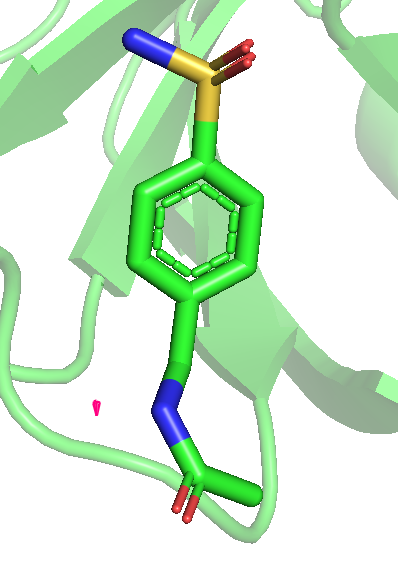
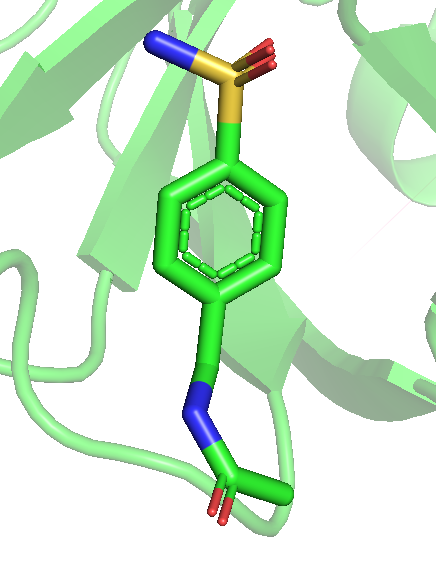
Рис 7. Карта ЭП вокруг лиганда после использования mesh с параметром carve = 1.5. Уровни 0.25, 0.5, 1, 1.5, 2 и 3 слева направо.
Можно предположить, что покрыты элеткроотрицательные атомы, (прим: атом серы с кислородами только до уровня 1 включительно), то есть ЭП сохраняется дольше там, где больее электроотрицательные атомы. Очень красиво видно, как распределяются эп по кольцу, исчезая через одну вершину шестиугольника.
© Grigorjeva Masha