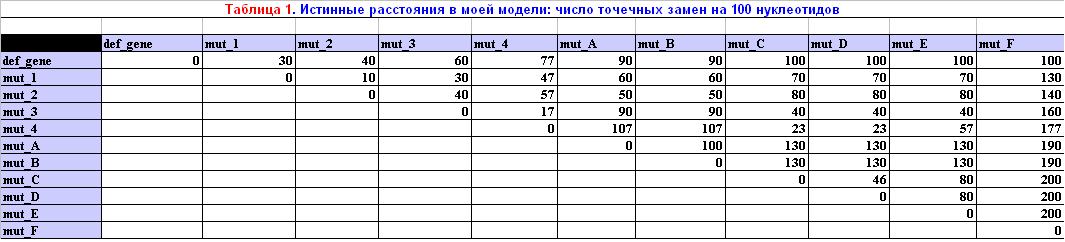

|
|
Задание 2. Определение соответствующих попарных расстояний с помощью программы
distmat пакета EMBOSS .
Как известно, программа distmat пакета EMBOSS используется для подсчета расстояний между последовательностями во множественном выравнивании. Выравнивание не содержит гэпов, так как модель не предусматривала ни вставки, ни делеции.
Для того, чтобы построить матрицу для среднего числа нуклеотидов на 100 позиций и матрицу попарных расстояний, вычисленных по формуле Джукса-Кантора, я запускала программу distmat и меняла значения параметра -nucmethod. Для построения первой матрицы я использовала значение параметра — 0, а во втором случае — 1.
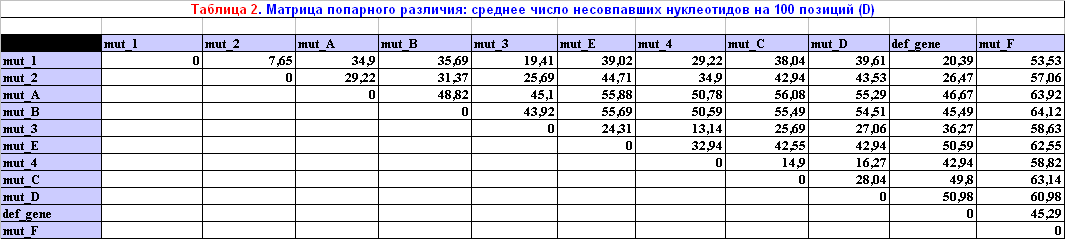
|
|
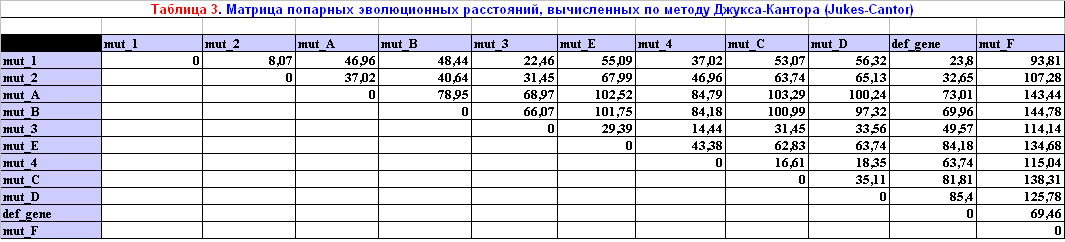
|
|
Задание 3. Построение и исследование графика зависимости попарных эволюционных расстояний, определенных способами [1] и [2] от "истинных" расстояний.
С помощью Excel была составлена сводная таблица по трём приведенным выше таблицам,
полученную таблицу я отсортировала по убыванию. В таблице указаны истинное расстояние и два расстояния, которые были получены программой
distmat пакета EMBOSS.
Затем по данным сводной таблицы был построен график.
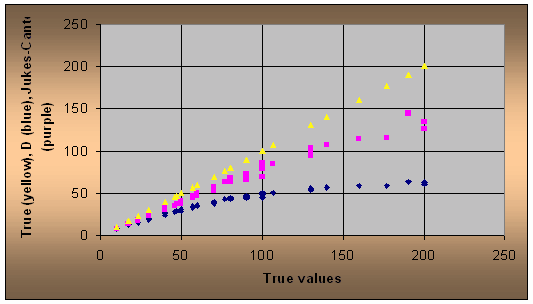
На полученном графике жёлтым цветом показана кривая "истинных" значений (для удобства сравнения отклонений), фиолетовым цветом - значения, полученные использованием метода Джукса-Кантора, а синим значения, полученные при оценке доли несовпавших нуклеотидов по матрице попарных различий (D).
Данный график наглядно демонстрирует, что оба метода не дают точную оценку эволюционных расстояний. Хотя при небольших количествах замен (в диапазоне примерно от 10 до 50) эти методы выдают более-менее сходное с "истинным" приближение. Нелишним будет отметить, что самое точное приближение на всем диапазоне даёт метод Джукса-Кантора, потому что при оценке он учитывает возможность обратных замен, то есть того, что нуклеотид может замениться сам на себя. Оценка же долей несовпавших нуклеотидов (D) учитывает только число прямых замен. Видно, что обе модели предсказывают меньшее попарное расстояние между последовательностями, чем то, что наблюдается в действительности. Это происходит потому, что обе оценки не учитывают скоростей эволюции отдельных доменов белка, а также различий в вероятности замены разных нуклеотидов.