Исследование структуры тРНК.
- Краткое описание структуры в файле
1H3E.PDB В файле приведены координаты атомов следующих молекул:
1. Фермент (лигаза) Тирозил-тРНК-синтетаза (из бактерии Thermus thermophilus) - 1 молекула.
2.�Tyr(GUA)тРНК (искусственно синтезирована) - 1 молекула.
3. Тирозинал - 1 молекула.
4. АТФ - 1 молекула.
5. Вода - 13 молекул.
Для исследования была выбрана цепь В, представляющая тирозил-тРНК со следующей последовательностью:5' 1 GGGCAGGUUC CCGAGCGGCC AAAGGGGACG GUCUGUAAAA CCGUUGGCGU 51 AUGCCUUCGC UGGUUCGAAU CCAGCCCUGC CCACCA [86] (76)* 3'
где 1 и 86 - номера первого и последнего нуклеотида.
В последовательности на 3'-конце есть триплет CCA, к которому присоединяетс6я аминокислота, но в файле PDB не приведены координаты атомов этих нуклеотидов. - Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями ( 1H3E_old.out). В соответствии с полученными данными:- акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 66-72
- Т-стебель - из 49-53 и комплементарного ему 61-65
- D-стебель - из 10-12 и комплементарного ему 23-25
- антикодоновый стебель- из 26-31 и комплементарного ему 39-44
Я создала скрипт для 6получения в RasMol изображения остова исследуемой тРНК, где акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым. Антикодон (G34, PSU35, A36) представлен в шарнирной модели и выделен желтым:
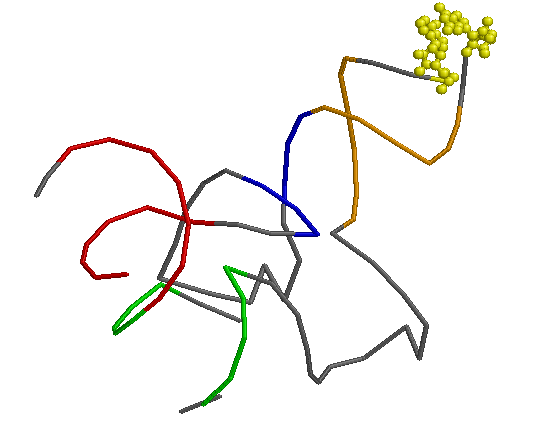
Рис.1. Вторичная структура Tyr(GUA)тРНК.Скрипт для получения изображения: restrict none background white select nucleic backbone 80 color grey select 66-72, 1-7 color red select 49-53, 61-65 color green select 10-12, 23-25 color blue select 26-31, 39-44 color orange select 34-36 and nucleic wireframe 80 cpk 200 color yellow
Структуру стеблевых дуплексов поддерживают 20 канонических и 1 неканоническая пара оснований G26-U44:
Также имеются другие особенности:
1) наличие вариабельной петли (нуклеотиды 47A-47I);
2) отсутствие остатка тимидина в Т-петле;
3) наличие 5-метилуридина (5MU54);
4) отсутствие дигидроуридинов в D-петле.
- Исследование третичной структуры
1.В файле 1H3E_old.out находятся данные о величине площади "перекрывания" 2-х последовательных пар азотистых оснований.
В файле
stacking.pdb - координаты взаимодействующих оснований.
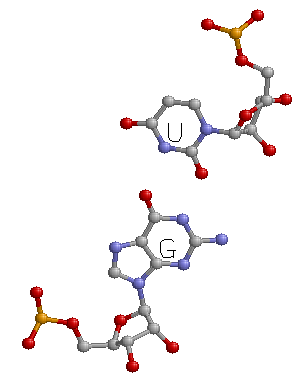
Взаимодествие между основаниями акцепторного (G7-C65) и T-стебля (C66-G449):
В D-петле имеется сильное перекрывание (G53-5MU54/C61-A58):
Существует также большое количество стекинг-взаимодействий между соседними парами одного стебля, например G3-C70/C4-G69 акцепторного стебля:
2. Третичную структуру так же стабилизируют дополнительные водородные связи. Например, связь между D- и Т-петлями (G19-C56):
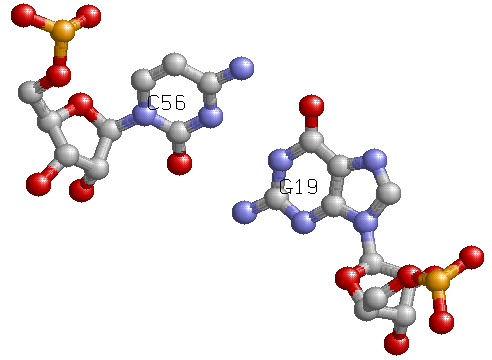
Другие дополнительные водородные связи: U47H-A20B (V-D петли), C48-G15(V-T петли), PSU55-G18 (T петля-D петля), U8-A14 и U9-G13 (основание акцепторного стебел-D-петля).
� - Предсказание вторичной структуры тРНК
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных последовательностях. С помощью этой программы был проведен поиск возможных комплементарных участков в последовательности исследуемой тРНК. Сначала я использовала стандартные параметры, но программа не смогла найти таких участков. При уменьшении штрафов и увеличении очков за совпадения программа смогла построить акцепторный стебель. (выходной файл программы 1H3E.einverted).
Также с помощью программы mfold пакета EMBOSS применялся алгоритм Зукера. Меняя несколько раз параметры, я не добилась нужного результата, так как ни в одном из полученных файлов я не увидела верную вторичную структуру. Вот пример одного из полученных мной результатов: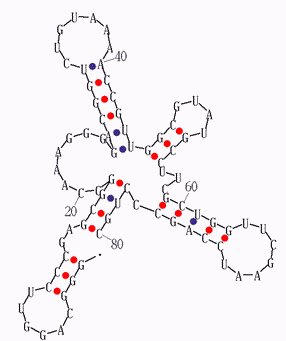
Реальная и предсказанная вторичная структура тРНК из файла 1H3E.pdb
Участок структуры
Позиции в структуре
(по результатам find_pair)Результаты предсказания
с помощью einvertedРезультаты предсказания
по алгоритму ЗукераАкцепторный стебель 5' 1-7 3'
5' 66-72 3'
Всего 7 пар7 6 D-стебель 5' 10-12 3'
5' 23-25 3'
Всего 3 пары0 0 T-стебель 5' 49-53 3'
5' 61-65 3'
Всего 5 пар0 0 Антикодоновый стебель 5' 26-31 3'
5' 39-44 3'
Всего 6 пар0 0 Общее число канонических пар нуклеотидов 21 7 6
<<Обратно на третий семестр
<<Обратно на главную страницу©Лелекова Мария,2009