Занятие 8
- Что значит код заданного фермента.
На странице БД UNIPROT с описанием выданного мне белка K6PF1_ECOLI я нашла ЕС код фермента, а на сайте International Union of Biochemistry and Molecular Biology его расшифровку:
- EC код: 2.7.1.11
- 2. - Transferases (Трансферазы - отдельный класс ферментов, катализирующих перенос функциональных групп и молекулярных остатков от одной молекулы к другой. Широко распространены в растительных и животных организмах, участвуют в превращениях углеводов, липидов, нуклеиновых и аминокислот.)
- 2.7. - Transferring phosphorus-containing groups (Трансферазы, совершающие перенос групп, содержащих фосфор)
- 2.7.1. - Phosphotransferases with an alcohol group as acceptor (Фосфотрансферазы,которые катализируют внутримолекулярный перенос фосфата с одной позиции на другую, с гидроксогруппой в качестве акцептора)
- 2.7.1.11. - 6-phosphofructokinase (6-фосфофруктокиназа, катализирующая реакцию: ATP + D-fructose 6-phosphate = ADP + D-fructose 1,6-bisphosphate)
- Уравнение катализируемой реакции: Atp + D-fructose 6-phosphate = adp + D-fructose 1,6-bisphosphate.
- графическое изображение катализируемой реакции:
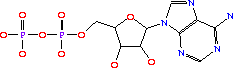
- Определите, в каких метаболических путях участвует изучаемый фермент.
Имя локуса: b2813
В БД KEGG был произведен поиск eco:b2813
Метаболические пути, ассоциированных с геном белка K6PF1_ECOLI:
Идентификатор KEGG Название по-английски по-русски eco00010 Glycolysis / Gluconeogenesis Гликолиз / Глюконеогенез eco00030 Pentose phosphate pathway Пентозофосфатный путь eco00051 Fructose and mannose metabolism Метаболизм фруктозы и маннозы eco00052 Galactose metabolism Метаболизм галактозы eco01100 Metabolic pathways Метаболические пути
Карта гликолиза / глюконеогенеза
Поиск в KEGG структурных формул заданных соединений
Название Идентификатор Структурная формула по-русски по-английски D-глюконат D-gluconate C00204 D-рибоза D-ribose C00117 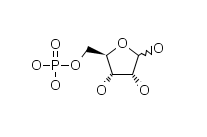
Поиск метаболического пути от одного заданного вещества к другому
Был использован инструмент "Color objects in pathways".
Запрос:C00204 red C00117 greenОткрылся список карт.
Была выбрана карта "ko00030 Pentose phosphate pathway" (пентозофосфатный путь - один из пяти возможных путей метаболизма глюкозы в клетке).
Выбранная цепочка ферментативных реакций:
путь: метаболизм рибозы
цепочка: D-рибоза > D-глюконатПромежуточные соединения:
- D-Ribose-5P (C00117),
- D-Glyceraldehyde-3P (C00118),
- D-Glucose (C00031),
- D-Glucono-1,5-lactone (C00198),
Первое соединение закрашено красным цветом, последнее - зеленым, промежуточные - желтым.
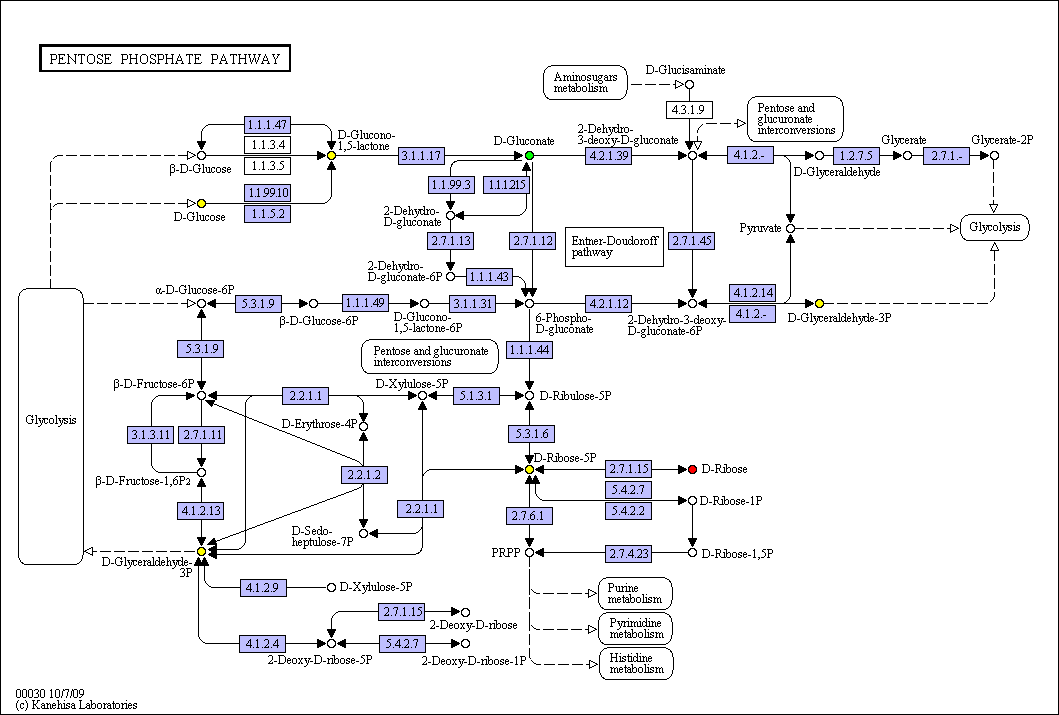
Сравнение метаболических путей у разных организмов
Возможность протекания выбранной цепочки ферментативных реакций в разных организмах с известными полными геномами:
Организм Возможна ли цепочка реакций Обоснование Карта Escherichia coli K-12 MG1655 да присутствуют все ферменты, необходимые для осуществления цепочки реакций карта Archaeoglobus fulgidus да присутствуют все ферменты, необходимые для осуществления цепочки реакций карта Arabidopsis thaliana да присутствуют все ферменты, необходимые для осуществления цепочки реакций карта Homo sapiens да присутствуют все ферменты, необходимые для осуществления цепочки реакций карта
Сравнение ферментов из далеких организмов
Был выдан EC-код: 2.5.1.6
С помощью одного запроса к SRS нашла ферменты с заданным EC-кодом у человека и археи Archaeoglobus fulgidus (окончание имени "_ARCFU").
Опция использования маски (Use wildcards) была снята, чтобы находились только ферменты с кодом 2.5.1.6, а не 2.5.1.6*, где * - какая-то цифра.
([uniprot-ECNumber:2.5.1.6] & ([uniprot-ID:*_human*] | [uniprot-ID:*_ARCFU*]))
Нашлось 7 белков, пять из которых принадлежат человеку, а два - археям.HEM3_ARCFU METK_ARCFU
HEM3_HUMAN METK1_HUMAN METK2_HUMAN PGTA_HUMAN PGTB2_HUMAN
Сравнение доменной организации найденных белков
ID Pfam AC Изображение Координаты доменов HEM3_ARCFU PF01379/PF03900 
2-212/217-289 METK_ARCFU PF01941 
1-396 HEM3_HUMAN PF01379/PF03900 
20-236/244-324 METK1_HUMAN PF00438/PF02772/PF02773 
16-115/128-250/252-389 METK2_HUMAN PF00438/PF02772/PF02773 
16-115/128-250/252-389 PGTA_HUMAN PF01239/PF07711/PF07711 
47-77/91-121/127-156/162-192/210-240/243-346/465-485 PGTB2_HUMAN PF00432 
66-109/114-157/162-205/210-253/258-302 Как видно из таблицы, есть 2 белка человека со схожей доменной организацией (METK1_HUMAN и METK2_HUMAN). Наиболее отличающаяся доменная организация у двух белков METK_ARCFU (только один домен) и PGTA_HUMAN (несколько доменов).
Для дальнейшего анализа были выбраны белки: METK_ARCFU и METK1_HUMAN.
Поиск ортологов
Для белка METK_ARCFU имя локуса его гена - afu:AF0050 .
С помощью инструментов KEGG для выбранного архейного белка был найден лучший ортолог из эукариот.
Им оказался белок fpl:Ferp_1054 (methionine adenosyltransferase).
Процент совпадения: 72.7%
Длина выравнивания: 399 аминокислотных остатка
Для человеческих белков не найдена информация о локусах.
<<Обратно на четвертый семестр
<<Обратно на главную страницу©Лелекова Мария,2010