Занятие 6
- Построить выравнивание заданного белка и белка-прототипа с разметкой трансмембранных сегментов.
- Сравнить нумерацию остатков белка-прототипа в UniProt и PDB
Был выдан идентификатор PDB белка-прототипа:3G5U
На домашней страничке PDBsum было получено выравнивание последовательности из UniProt и последовательности из PDB:
Выравнивание в формате algn1.msf
Нумерация в обеих базах данных совпадает. В базе данных PDB последовательность белка на 5 а.к.остатков меньше,чем в UniProt.
- Постройте полное глобальное выравнивание заданного белка и белка-прототипа.
Мне достались белок MDR1_CRIGR и белок-прототип MDR3_MOUSE.
Полученные последовательности были отданы на вход программе needle:
needle -asequence MDR1_CRIGR.fasta -bsequence MDR3_MOUSE.fasta -outfile mdr_mdr.needle
На выходе получился файл mdr_mdr.needle - Создать по данным БД ОРМ разметку трансмембранных сегментов в белке-прототипе.
Найдем по PDB ID (3G5U) описание ТМ-сегментов
белка-прототипа MDR3_MOUSE в БД OPM.
Белок MDR3_MOUSE имеет код 1.1.09.06, где:
1. - Белок относится к типу трансмембранных белков (Transmembrane, 2 класса)
1.1. - Белок относится к классу альфа-спиральных трансмембранных белков (Alpha-helical transmembrane, 57 надсемейств)
1.1.09. - Белок относится к надсемейству ABC-транспортеров (ABC transporters, 6 семейств)
1.1.09.06 - Белок относится к семейству не восприимчивых к лекарствам экспортеров(Multidrug resistance exporter (MDR))В БД OPM было получено описание ТМ-сегментов в белке-прототипе 3G5U.
Описание ТМ-сегментов для цепи A:
A - Tilt: 3° - Segments: 1( 48- 72), 2( 111- 131), 3( 185- 207), 4( 210- 233), 5( 290- 314), 6( 327- 348), 7( 708- 729), 8( 750- 773), 9( 827- 847), 10( 853- 872), 11( 931- 953), 12( 971- 988)
Исходя из этих координат были найдены трансмембранные сегменты.
Оставшиеся участки последовательности являются цитоплазматическими или внешними, выходящими наружу клетки. Для того, чтобы определить ориентацию этих участков относительно мембраны, белок рассматривался при помощи Jmol.Полученный файл marc.msf с выравниванием.
Заметим, что у белка-прототипа 3G5U цепь А содержит 12 трансмембранных доменов. При этом почти вся цепь находится в цитоплазме, а лишь небольшая ее часть обращена во внеклеточное пространство. Цепь начинается и заканчивается на цитоплазматической стороне.
- Предсказать топологию заданного белка с помощью наиболее популярной программы TMHMM.
Предскажем топологию белка MDR1_MOUSE с помощью сервера TMHMM.
На вход подадим ему файл MDR1_CRIGR.fasta с последовательностью белка в формате FASTA.
Выдача программы:
# sp_P21448_MDR1_CRIGR Length: 1276 # sp_P21448_MDR1_CRIGR Number of predicted TMHs: 11 # sp_P21448_MDR1_CRIGR Exp number of AAs in TMHs: 247.07675 # sp_P21448_MDR1_CRIGR Exp number, first 60 AAs: 12.77622 # sp_P21448_MDR1_CRIGR Total prob of N-in: 0.98442 # sp_P21448_MDR1_CRIGR POSSIBLE N-term signal sequence sp_P21448_MDR1_CRIGR TMHMM2.0 inside 1 47 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 48 70 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 71 112 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 113 135 sp_P21448_MDR1_CRIGR TMHMM2.0 inside 136 186 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 187 209 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 210 213 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 214 236 sp_P21448_MDR1_CRIGR TMHMM2.0 inside 237 291 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 292 314 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 315 323 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 324 346 sp_P21448_MDR1_CRIGR TMHMM2.0 inside 347 706 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 707 729 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 730 751 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 752 774 sp_P21448_MDR1_CRIGR TMHMM2.0 inside 775 847 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 848 870 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 871 931 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 932 954 sp_P21448_MDR1_CRIGR TMHMM2.0 inside 955 966 sp_P21448_MDR1_CRIGR TMHMM2.0 TMhelix 967 989 sp_P21448_MDR1_CRIGR TMHMM2.0 outside 990 1276
Также прилагается график, отображающий предположение о топологии:
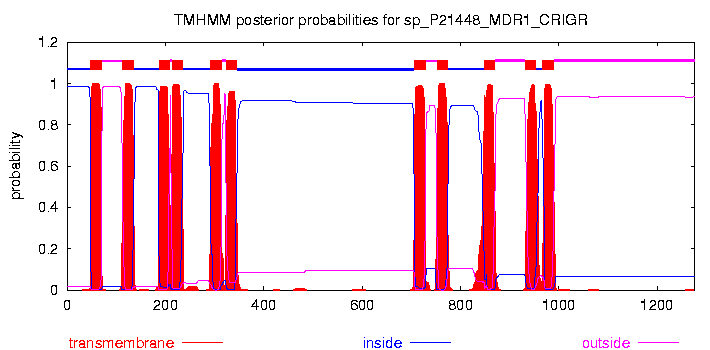
Полученное предсказание добавим к выравниванию файла mark.msf в виде искусственной последовательности с разметкой трансмембранных спиралей под названием "TMHMM". Полученное выравнивание сохранено в формате msf в файлe mark2.msf
- Сравните полученное предсказание с данными ОРМ.
Cравним предсказания, полученные с помощью программы TMHMM, с предсказаниями OPM.
Число а.к. остатков Всего а.к. остатков 1277 Остатки, предсказанные как локализованные в мембране (всего) 229 Правильно предсказали (true positives, TP) 200 Предсказали не то, что нужно (а.о. предсказаны как мембранные, а по данным ОРМ таковыми не являются, false positives, FP) 29 Правильно не предсказали ( не предсказаны, и по данным ОРМ не находятся в мембране, true negatives, TN) 978 Не предсказали то, что нужно (остатки по данным ОРМ находятся в мембране, false negatives, FN) 70 Чувствительность (sensivity) = TP / (TP+FN) 0.7407 (74,07%) Специфичность (specificity) = TN / (TN+FP) 0.9712 (97,12%) Точность(precision) = TP /(TP+FP) 0.8733 (87,33%) Сверхпредсказание = FP/ (FP+TP) 0.1266 (12,66%) Недопредсказание = FN / (TN+FN) 0.0667 (6,67%) Как видно из таблицы большая доля (12,66%) предсказанных трансмембранных сегментов не являлась таковой. К тому же 6,67% трансмембранных последовательностей было пропущено при предсказании. Можно заметить, что специфичность очень высока, при этом чувствительность и точность оказались ниже. Интересно, что предсказаны десять из двенадцати трансмембранных спиралей, при этом некоторые из этих спиралей предсказаны частично. Можно заметить, что локализацию достаточно большой части белка (примерно 350 позиции выравнивания) TMHMM предсказал как внеклеточную, в то время как OPM предсказал ее как трансмембранную.
<<Обратно на четвертый семестр
<<Обратно на главную страницу©Лелекова Мария,2010
- Сравнить нумерацию остатков белка-прототипа в UniProt и PDB
Был выдан идентификатор PDB белка-прототипа:3G5U