Занятие 10
Проект "Поиск белка с заданной функциональной специфичностью."
- Первый этап: описание функциональных особенностей заданной группы.
Мне был выдан белок-прототип MALI_ECOLI, выделенный из кишечной палочки Escherichia coli K-12. Имя гена - malI, длина последовательности 342 аминокислотных остатков.
Я произвела поиск информации о заданном белке в базе данных EcoCyc. Страница с описанием белка MALI_ECOLI.
MalI - мальтозный репрессор, который контролирует гены, относящиеся к мальтозной системе. Он отрицательно саморегулируется и координационно репрессирует транскрипцию оперона, который кодирует мальтоза-глюкоза PTS пермеазу (фермент, участвующие в транспорте метаболитов) и бифункциональный белок, который взаимодействует с активатором транскрипции MalT.
Термины GO, связанные с белком MALI_Ecoli:
GO Онтология GO (название словаря) Термин GO:0006355 biological process regulation of transcription, DNA-dependent GO:0045449 biological process regulation of transcription GO:0006350 biological process transcription GO:0003677 molecular function DNA binding GO:0003700 molecular function transcription factor activity GO:0005622 cellular component intracellular
Где?
(cellular component)Внутриклеточный Зачем, для чего?
(biological process)Транскрипция; регуляция ДНК-зависимой транскрипции Молекулярный механизм?
(molecular function)Связывание с ДНК; активность транскрипционного фактора Специфичность?
(molecular function)- Следовательно, в аннотации GO нет информации об эффекторе белка MALI_ECOLI - мальтозе.
Структурная формула мальтозы (БД KEGG, ID C00208):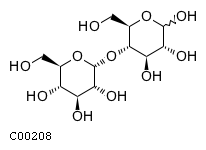
- Второй этап: создание множественного выравнивания доменов с разметкой по группам специфичности.
- Создание хорошего множественного выравнивания доменов заданной группы белков
Доменная структура белка MALI_ECOLI (информация из БД Pfam):
Cхема из Pfam: 
Пояснения к схеме Pfam AC Pfam ID Положение в последовательности белка MALI_ECOLI Клан PF00356 LacI 8-53 HTH (CL0123) PF00532 Peripla_BP_1 64-342 Periplas_BP-like (CL0144) Домен LacI (зеленый) является ДНК-связывающим, домен Peripla_BP_1 (красный) - эффекторсвязыюващий домен.
Полученное выравнивание всех эффекторсвязывающих доменов даного типа в fasta-формате.
С помощью скрипта из общего выравнивания добудем выравнивание нужных доменов.Их идентификаторы находятся в файле mali
В файле PF00532_mali.fasta находится множественное выравнивание эффектор-связывающих доменов группы спецефичности mali.
- Создание единого множественного выравнивания заданных доменов всех групп специфичности
При выполнение предыдущего задания так же было обнаружено, что в списке всех белков присутствуют не все, имеющие, по мнению эксперта, данную спецефичность. Их последовательности были найдены отдельно и добавлены в список.
Полученное выравнивание было импортировано в GeneDoc и там раскрашено по группам спецефичности (mali показана зеленым цветом).
Полное выравнивание с раскраской по группам спецефичности. В нем имеется консервативная для всех позиция 145 (покрашена черным).Колонки букв, окрашенные цветом, совпадающим с цветом названия белков, указывают на позиции, консервативные в доменах соответствующей группы специфичности. Наблюдаются так же консервативные позиции хотя бы для 6 групп спецефичности - колонки 36,58,62,98,117,137,189,219 и еще несколько. Консервативными в пределах набора последовательностей считали позиции, представленными одинаковыми аминокислотными остатками или аминокислотными остатками, входящими в одну группу сходства. Группы сходства (как в GeneDoc): (D,N), (E,Q), (S,T), (K,R), (F,Y,W), (L,I,V,M)
Длинных консервативных участков в моей группе спецефичности не нашлось - всего колонок с одними и теми же остатками для всей группы - 62, но самые длинные консервативные участки - с 4-8 и 123-127, т.е.4-5 а.к.остатков.
- Ресурс WebLogo
Я получила изображения для полного выравнивания.
И для моей группы спецефичности.
Изображение полного выравнивания эффекторсвязывающих доменов демонстрирует консервативность разных остатков на разных позициях в эффекторсвязывающих доменах всего семейства.
Из картинки видно, что "большие" буквы на лого совпадают с выделенными колонками, консервативными в полном выравнивании эффекторсвязывающих доменов.
Изображение выравнивания доменов группы специфичности mali иллюстрирует консервативность остатков в эффекторсвязывающих доменах исключительно данной группы специфичности.
- Третий этап: Создание профиля для доменов белков заданной группы специфичности
1). Сначала я получила выравнивание Днк-связывающих доменов группы специфичности mali.
Использовала домен из БД SMART, так как длина ДНК-связывающего домена SM00354 из БД SMART превосходит длины ДНК-связывающих доменов из других баз.
Воспользовалась ссылкой на SM00354.
Представительское выравнивание сохранено в файл SMART.fasta.
Последовательности всех бактериальных белков, содержащих домены данного семейства сохранены в файл family.fasta Было обнаружено, что в файле family.fasta, полученном из БД SMART, отсутствует один из белков, указанных в списке идентификаторов последовательностей, по мнению эксперта, обладающих данной специфичностью. Эта последовательность была получены из БД UniProt. Был запущен следующий скрипт:
#!/bin/bash for i in `cat mali`; do grep -A 5 ${i} family.fasta >> family_sort.fasta doneДанный скрипт ищет в файле family.fasta последовательности из списока, лежащего в файле mali. Для правильной работы скрипт и список должны быть сохранены в UNIX-формате. Выдача скрипта: файл family_sort.fasta. Я приписала к данному файлу последовательность белка B5XRE8. С помощью программы ClustalW2 последовательности белков, содержащих нужные ДНК-связывающие домены были выровнены под профиль представительского выравнивания SMART.fasta. Из полученного файла с выравниванием были вырезаны представительские последовательности и позиции выравнивания, не содержащие ДНК-связывающего домена. Полученное выравнивание было сохранено в файл dna_mali.fasta2). Далее по этому выравниванию был построен профиль.
1. C помощью программы pfw к выравниванию добавим веса:
pfw -m dna_mali.fasta > mali.pfw.fasta
2. C помощью команды pfmake построили профиль:pfmake -m mali.pfw.fasta /usr/share/pftools23/blosum45.cmp > mali.pfw.pfmake.pfl
3. С помощью команды autoscale нормируем полученный выше профиль относительно случайной базы:autoscale -m mali.pfw.pfmake.pfl > mali.pfw.pfmake.autoscale.pfl
4. С помощью программы pfsearch проведем поиск по протеому по профилям.Дан файл Pantoea.fasta, по которому и будем осуществлять поиск:
pfsearch -f mali.pfw.pfmake.autoscale.pfl Pantoea.fasta > mali.pfw.pfmake.autoscale.pfsearch.list pfsearch -x -f mali.pfw.pfmake.autoscale.pfl Pantoea.fasta > mali.pfw.pfmake.autoscale.pfsearch
5. Был получен файл со списком находок и файл с последовательностями находок. Всего нашлось 23 белка.С помощью программы ClustalW2 последовательности найденных белков были выровнены с ДНК-связывающими доменами группы специфичности mali. Далее выравнивание было импортировано в GeneDoc. Изображение выравнивания:
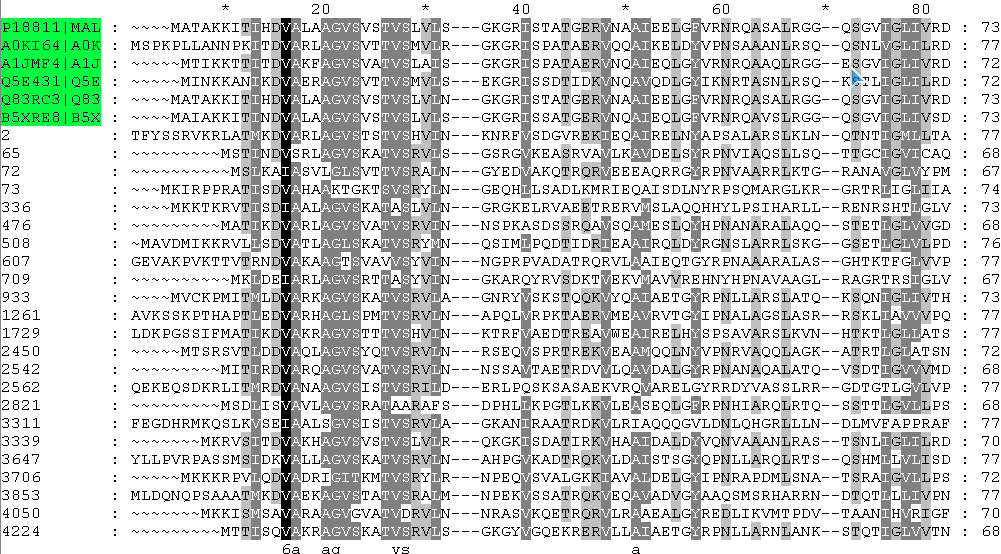
Для проверки важных позиций, воспользуемся БД PDB. Но в БД PDB не нашлось ни одной 3D-структуры белка группы специфичности mali. Я не обнаружила полностью консервативных позиций в выравнивании, поэтому можно сделать вывод, что найденная мной последовательность не принадлежит нашей группе специфичности.
<<Обратно на четвертый семестр
<<Обратно на главную страницу©Лелекова Мария,2010
- Второй этап: создание множественного выравнивания доменов с разметкой по группам специфичности.