Занятие 9
Качество структуры: PROCHECK, EDS, PDB_REDO.
Задание 1.
Необходимо было проанализировать выдачу программы PROCHECK для записи 1PFK PDB.Для этого мне нужно было воспользоваться базой данных PDBSum и со страницы моей структуры пройти по гиперссылке "PROCHECK" (слева).
Программа PROCHECK выдала следующее.
Карта Рамачандрана для остатков моей записи 1PFK:
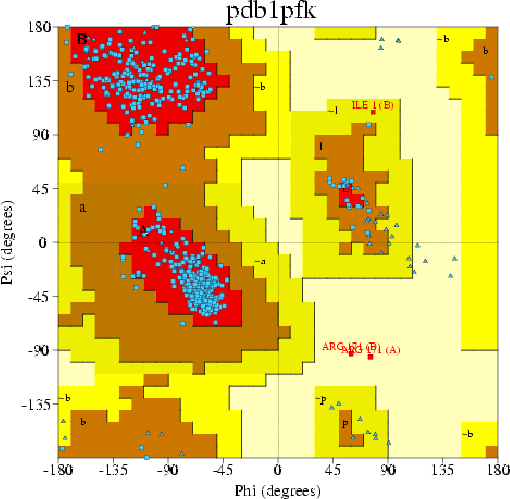
1. Ramachandran Plot statistics
No. of
residues %-tage
------ ------
Most favoured regions [A,B,L] 507 93.2%
Additional allowed regions [a,b,l,p] 34 6.2%
Generously allowed regions [~a,~b,~l,~p] 1 0.2%
Disallowed regions [XX] 2 0.4%*
---- ------
Non-glycine and non-proline residues 544 100.0%
End-residues (excl. Gly and Pro) 4
Glycine residues 76
Proline residues 16
----
Total number of residues 640
В предпочитаемые области на карте попало 507 остатков, что составляет 93.2%.На карте 2 остатка (отличных от Gly и Pro) отмечены красным цветом (составляют 0,4%). Это говорит об их ложности. Как видно, в целом структура белка правильная.
Задание 2.
На сервере EDS я зашла на страницу структуры записи 1PFK и прошла по гиперссылке "Significant regions".Открылась следующая страничка.
Количество остатков со значением Z-score, большим двух: 13 (10 - цепь А, 3 - цепь B).
С этого же сайта были получены графики z-score для цепей A, B:
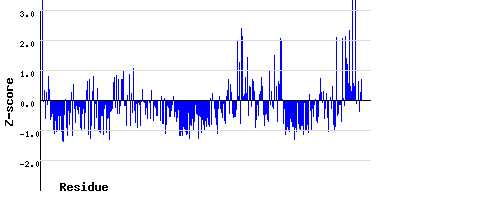
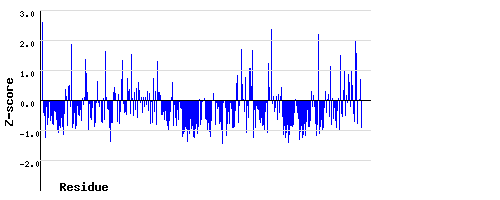
Почти все остатки имеют небольшой z-score (меньше 2.0).
Но в цепи A есть три остатка, имеющие z-score больше 3.0.
Значения z-score, которые превышают 3.0, достаточно подозрительны.
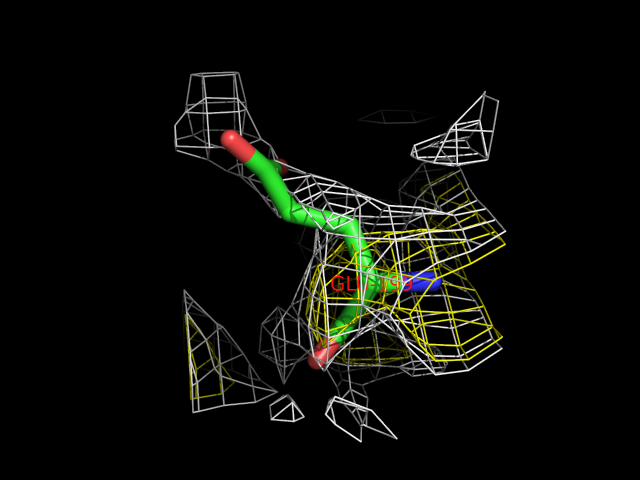
Для одного из таких остатков, глутаминовой кислоты 199 цепи А, я создала изображение, на котором видна его модель и электронная плотность вокруг него:
Задание 2.
Со страницы своей структуры в EDS я прошла по гиперссылке "PDB_REDO": выдыча PDB_REDO.R-values etc.
| From PDB header | Calculated from data | After conservative optimisation | After full optimisation | |
|---|---|---|---|---|
| R | 0.1740 | 0.1740 | 0.1485 | 0.1568 |
| R-free | --N/A-- | 0.1675 | 0.2000 | 0.1981 |
| σR-free | 0.0031 | 0.0036 | 0.0036 | |
| R-free Z-score | 22.03 | 0.36 | 4.00 |
Значение, рассчитанное R-free: 0.1675. Значение, рассчитанное R-free, указанного в PDB-файле, не указано.
После полной оптимизации R-free возрос, то есть ухудшился, а R-фактор уменьшился, то есть улучшился.
Это может быть вызвано тем, что во время оптимизации структура сильнее отклонилась от
экспериментальных данных.
z-score от R-free сильно уменьшился (с 22.03 до 4.00).
Снижение z-score говорит об улучшении оптимизации.
WHAT_CHECK validation
| Original PDB entry | Conservatively optimised | Fully optimised | |
|---|---|---|---|
| 1st generation packing quality1 | -0.221 | -0.371 | -0.228 |
| 2nd generation packing quality1 | -1.720 | -1.519 | -1.305 |
| Ramachandran plot appearance1 | -2.032 | -1.274 | -0.604 |
| Chi-1/Chi-2 rotamer normality1 | -3.094 | -2.444 | -1.469 |
| Backbone conformation1 | 0.201 | 0.111 | 0.311 |
| Bond length RMS Z-score2 | 0.695 | 0.973 | 0.523 |
| Bond angle RMS Z-score2 | 1.457 | 0.967 | 0.661 |
| Total number of bumps3 | 78 | 58 | 43 |
| Unsatisfied H-bond donors/acceptors3 | 49 | 24 | 33 |
1 Higher is better
2 Should be lower than 1.000
3 Fewer is better
Улучшились после полной оптимизации следующие параметры:
Ухудшился параметр:
<<Обратно на четвертый семестр
<<Обратно на главную страницу
©Лелекова Мария,2010