Занятие 1
Введение в PyMOL.
- Работа с записью 1KMY банка PDB.
C помощью команды get_pdb 1KMY на kodomo-count я скачала PDB-файл записи 1KMY.
Затем по полю HETNAM определила, что в структуре присутствует ион двухвалентного железа:HETNAM FE2 FE (II) ION HETNAM BPY BIPHENYL-2,3-DIOL HETNAM TBU TERTIARY-BUTYL ALCOHOL HETSYN TBU 2-METHYL-2-PROPANOL FORMUL 2 FE2 FE 2+ FORMUL 3 BPY C12 H10 O2 FORMUL 4 TBU C4 H10 O FORMUL 5 HOH *115(H2 O)
Далее в PyMOL была выполнена следующая командаcd H:\Term5\Practice1
для того, чтобы сделать активной мою рабочую директорию. Также была выполнена команда для того, чобы загрузить файл в PyMOL:
load 1kmy.pdb
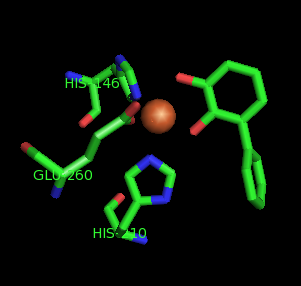
Затем мне необходимо было получить изображение окрестности иона, включая изображение остатков и лигандов, связанных с ионом координационными связями. Я использовала команду
show sticks, byres symbol FE around 3.5
которая позволила показать только остатки, связанные с ионом координационными связями: His 210, His 146, Glu 260 и Bpy 300 (Бифенил-2,3-диол).
- Задание 2.
В записи 1DLP изучите остатки 136 цепи A и 167 цепи С.
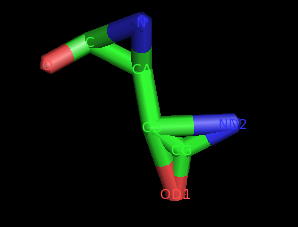
Я просмотрела запись 1DLP банка PDB: описывается структура предшественника лектина SCAFET, которая была получена при разрешении 3.3 ангстрем в 1999 году. Затем изучила необходимые остатки:Asn136
Arg167
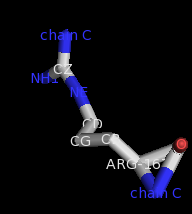
В этих остатках есть лишние ковалентные связи с кислородом и азотом. В остатке аргинина и в остатке аспарагина образован цикл атомами С, С-альфа и азота. У аспарагина также есть два цикла в боковой цепи: между атомами СG, CB и ND2 и между атомами CB, CG и OD1.
Проверила поле REMARK записи PDB: там сказано, что данные о положении и свойствах атомов CG, OD1 и ND2 в аспарагине и атомов CB, CG, CD, NE, CZ, NH1 и NH2 в аргинине могут быть ненадежными.В записи 1GT0 изучите участок цепи C с 75 по 100 остатки.
Описываемая структура тройного комплекса POU/HMG/DNA была получена при разрешении 2.6 ангстрем в 2002 году. Нам надо изучить участок цепи C с 75 по 100 о6статки. Но в графическом окне получили изображение только 75-77 и 97-100 участков. Данные об остатках в промежутке 78-96 в файле PDB отсутствуют, и, соответственно, я не увидела их изображение. Такое могло произойти в следствии того, что, в ходе выполнения рентгеноструктурного анализа, данный участок в кристалле плохо закристаллизовался.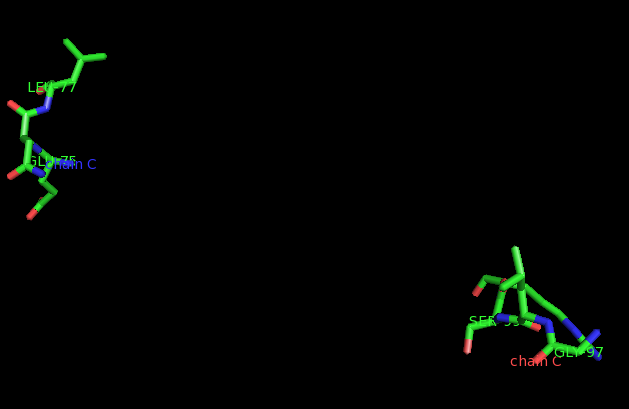
- Создание скрипта для PyMOL.
Я создала скрипт, который воспроизводит команды для работы с остатками 136 и 167 из записи 1DLP.pdb:
load 1dlp.pdb
hide all
select duo, (resi 136 and chain a) or (resi 167 and chain c)
show sticks, duo
<<Обратно на пятый семестр
<<Обратно на главную страницу©Лелекова Мария,2010