Результаты
Выход программ blastn (word size - 11) и megablast (word size - 28)
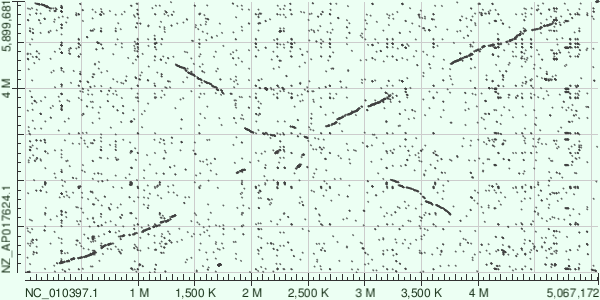
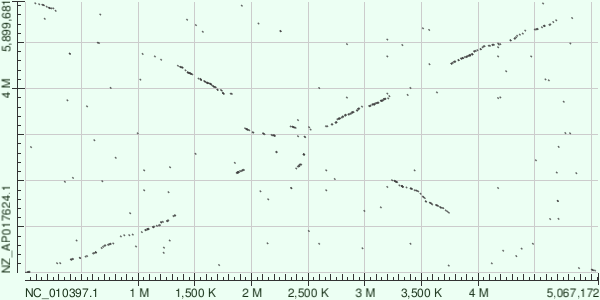
Megablast и blastn выстроились очень схожим образом, однако на dotplot BlastN больше мусора, так как параметр "word size" у этой программы меньше. Если сравнивать геномы бактерий, можно заметить многочисленные транслокации, причем некоторые кусочки еще и инвертировались. Разрывы линий связаны с наличием мутаций в геноме (делеций).