Реконструкция деревьев
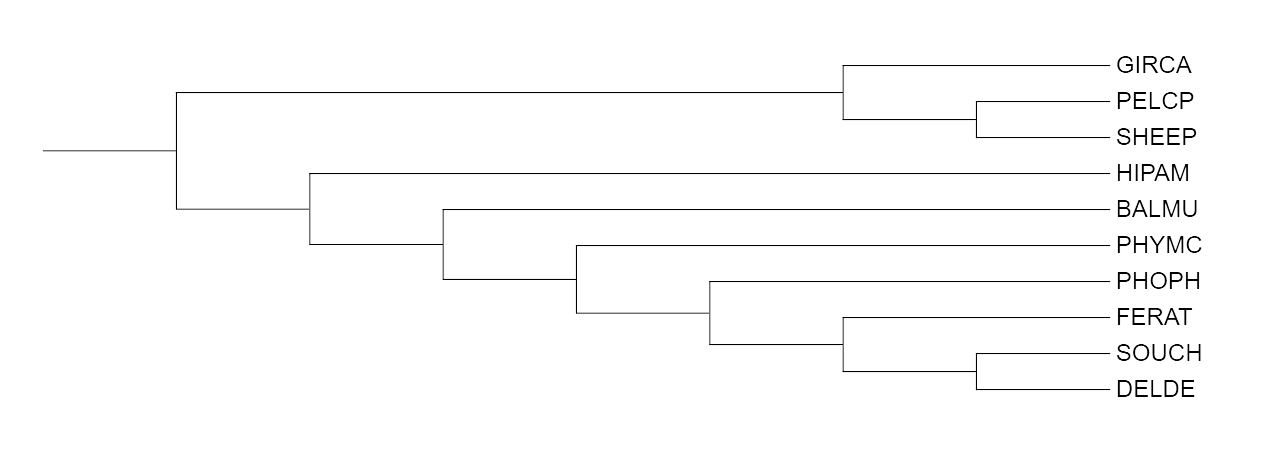
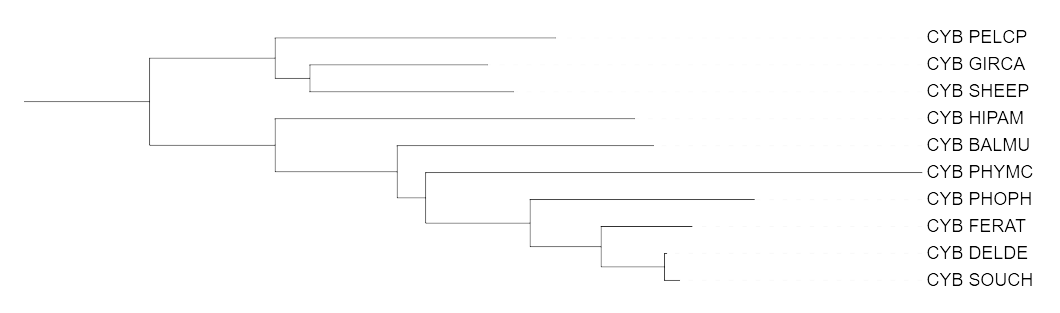
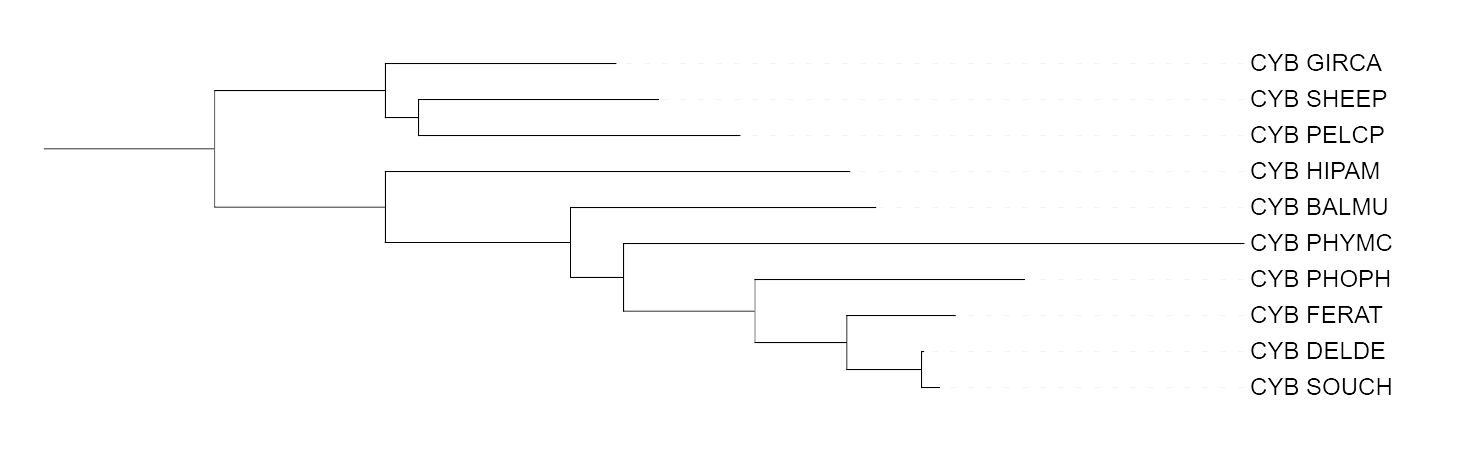
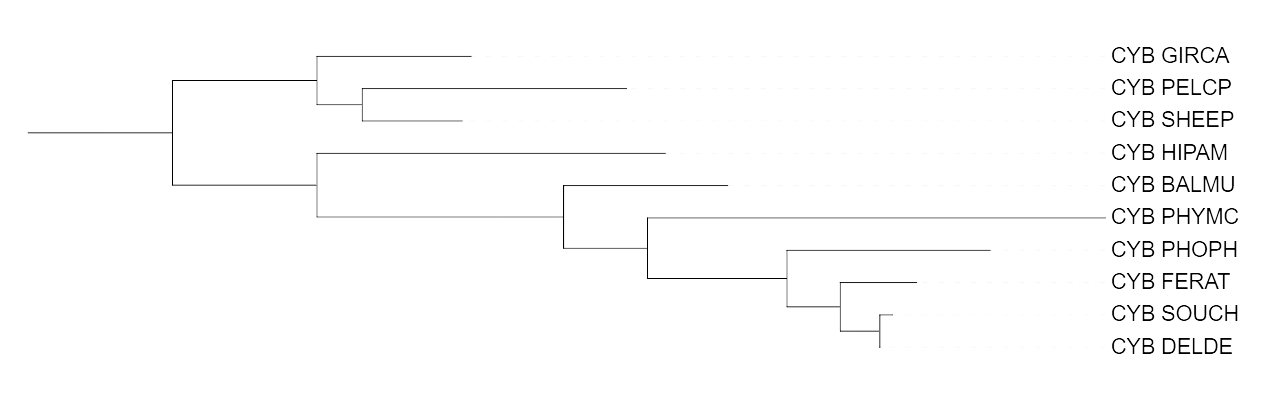
В данном практикуме было выполнено выравнивание последовательностей цитохромов B и реконструировано филогенетичсекое дерево тремя способами:
Выравнивание выполнялось при помощи программы muscle на kodomo.
*Последовательность цитохрома B у организма Cervus nippon nippon (CERNN) была некорректно подобрана для предыдущего задания, поэтому в данном практикуме использоваться не будет.
Реконструкция деревьев




Сравнение деревьев
После реконструкции все деревья оказались одинаковыми за исключением работы программы fastme с моделью p-distance, на визуализации которой Giraffa camelopardalis (GIRCA) оказался ближе к Ovis aries (SHEEP), чем Pelea capreolus (PELCP).