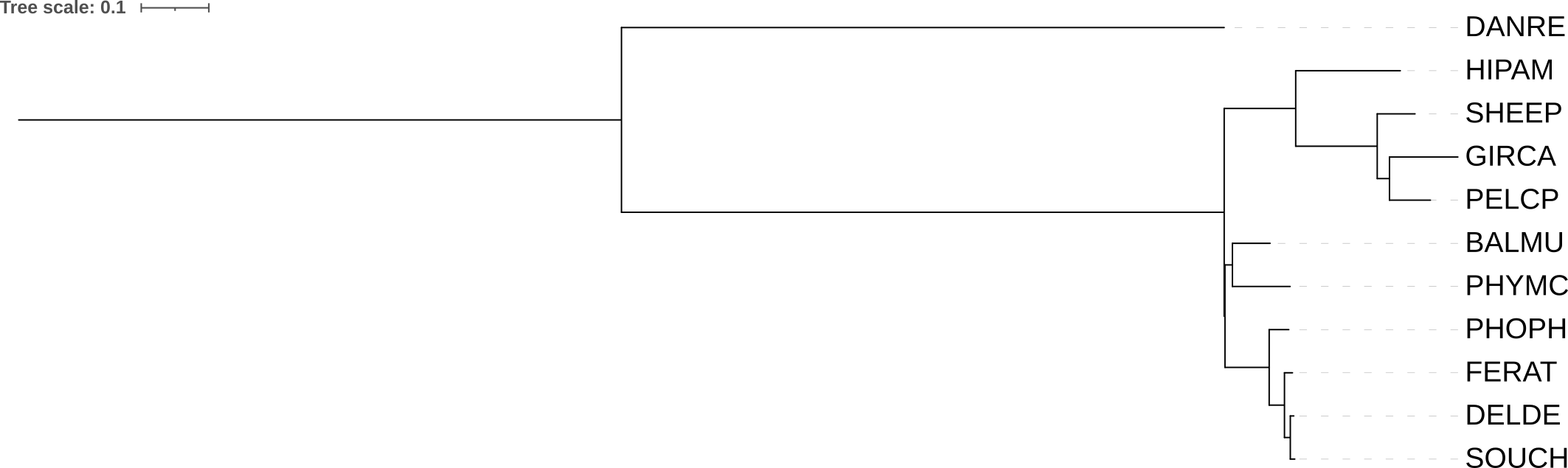
Практикум 3
Задание 1. Реконструкция дерева по нуклеотидным последовательностям
С помощью программы seqret из EMBL были получены последовательности 12s рРНК для животных, выбранных мной в прошлых практикумах.
Далее они были выровнены с помощью программы MUSCLE, а файл с выравниванием был переведен в формат phylip-relaxed. Дерево было реконструировано с помощью программы IQ-Tree.
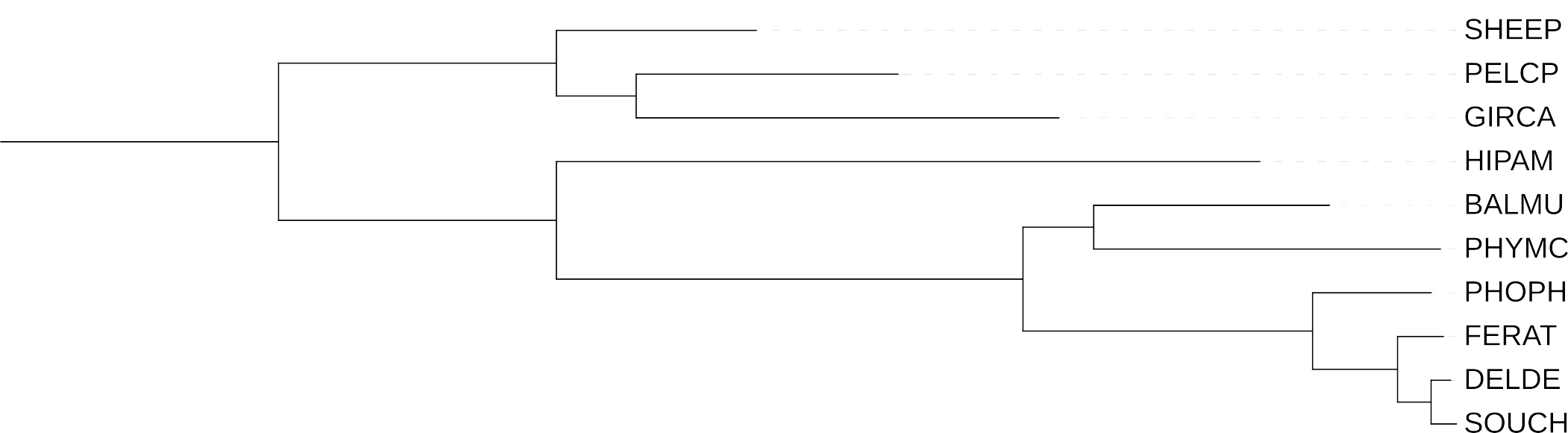
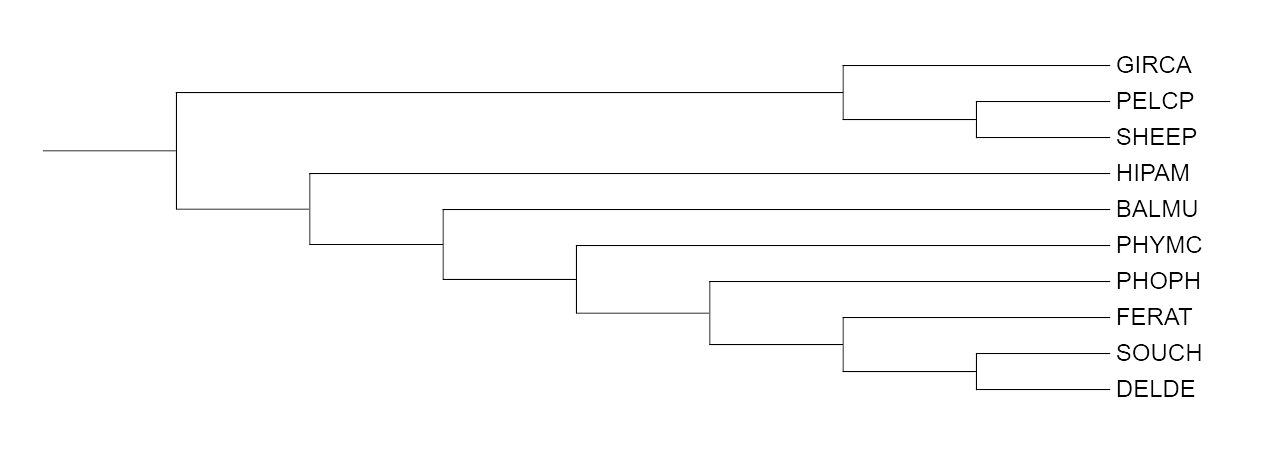
Сравним данное дерево с полученными в предыдущем практикуме:
Интересно заметить, что в дереве, реконструированном по последовательности 12s рРНК, кит и кашалот (BALMU и PHYMC) образуют вместе отдельную ветку, хотя в дереве NCBI Taxonomy они отходят поочерёдно.
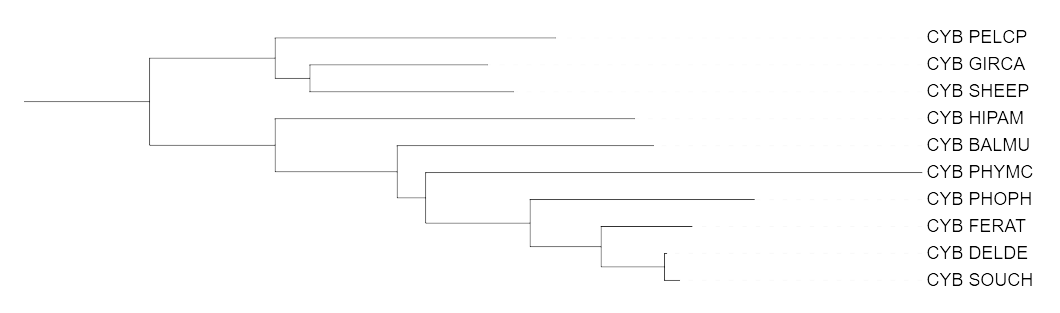
Также в полученном дереве вместе отделились сернобык и жираф (PELCP и GIRCA), стоит заметить, что в дереве, построенном по последовательности cytB с помощью fastme, жираф был отделён в ветку вместе с овцой (SHEEP), таким образом, полученные деревья демонстрируют все возможные варианты перегруппировок.