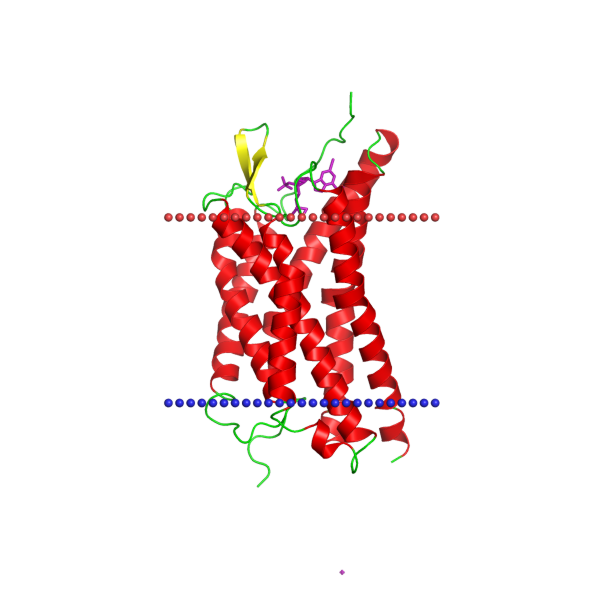
Сравнение предсказаний трансмембранных участков в бета-листовом белке
- Название на русском: Мальтопорин
- Название на английском: Maltoporin (LamB protein)
- Идентификатор PDB: 1AF6
- Идентификатор UniProt: P02943 (LAMB_ECOLI)
- Организм: Escherichia coli (Кишечная палочка)
- Расположение в мембране: Наружная мембрана
- Функция белка (по данным UniProt): Специализированный поровый белок наружной мембраны. Образует тример, и каждая субъединица формирует гидрофильный канал (β-баррел), который позволяет пассивно диффундировать малым молекулам. Специфически опосредует захват мальтозы и мальтодекстринов (линейных олигосахаридов из остатков глюкозы), что является ключевым этапом утилизации крахмала и гликогена бактерией. Также может функционировать как рецептор для некоторых бактериофагов (например, фага λ).
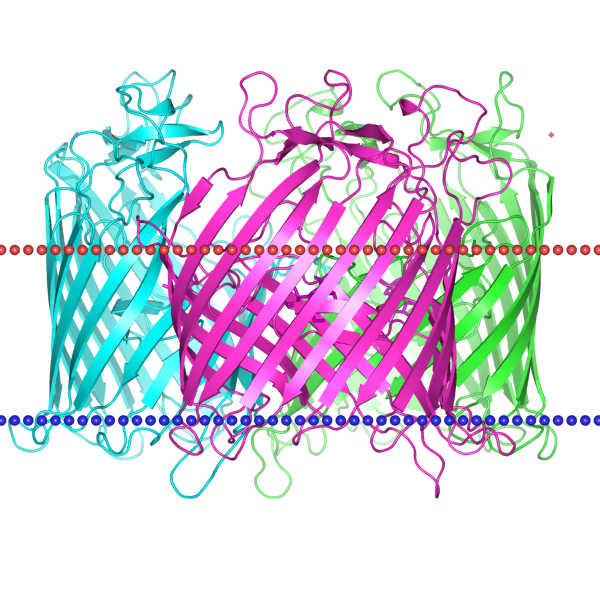
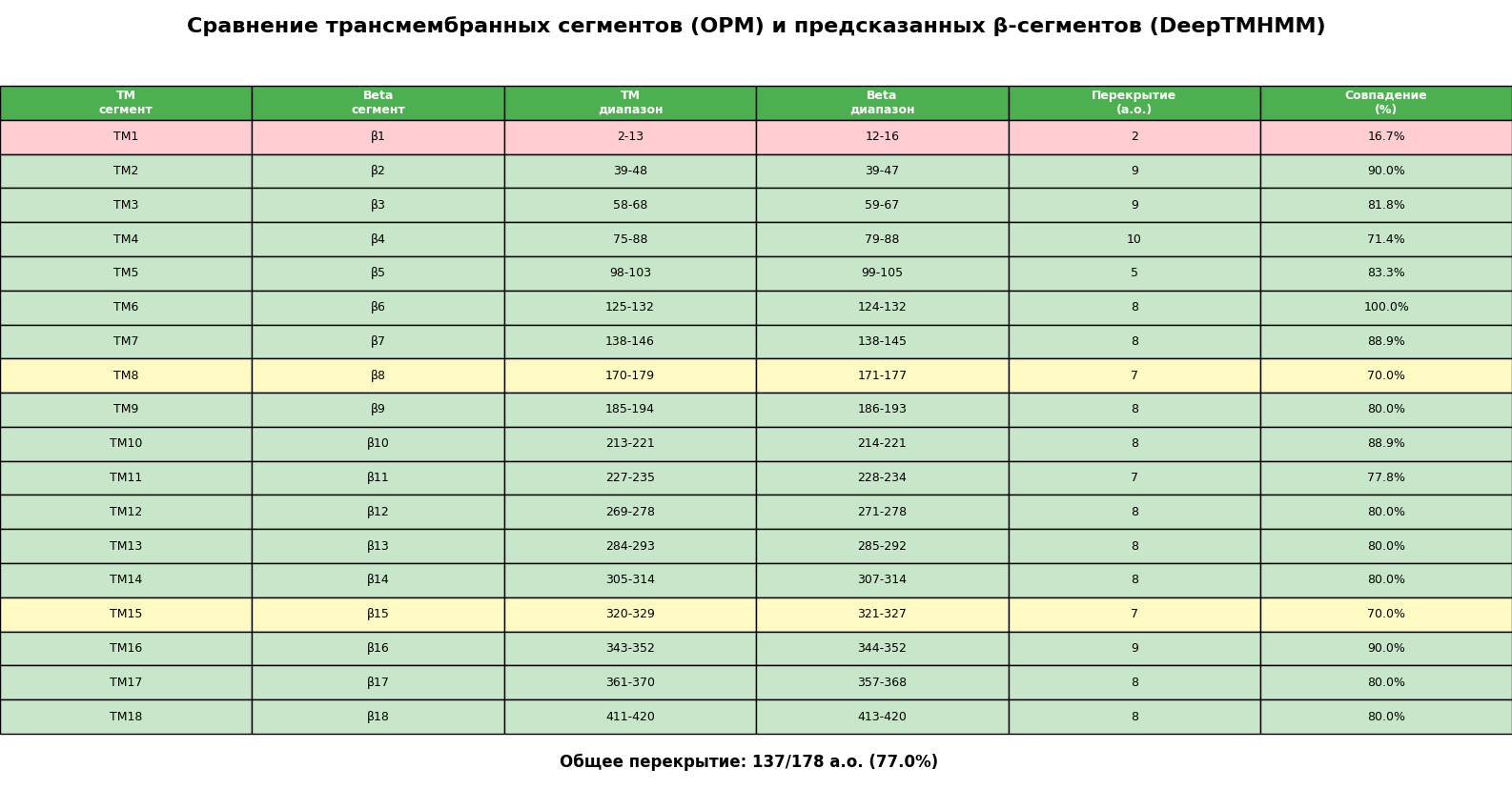
Координаты трансмембранных участков:
1(2-13),2(39-48),3(58-68),4(75-88),5(98-103),6(125-132),7(138-146),8(170-179),9(185-194),10(213-221),11(227-235),12(269-278),13(284-293),14(305-314),15(320-329),16(343-352),17(361-370),18(411-420)
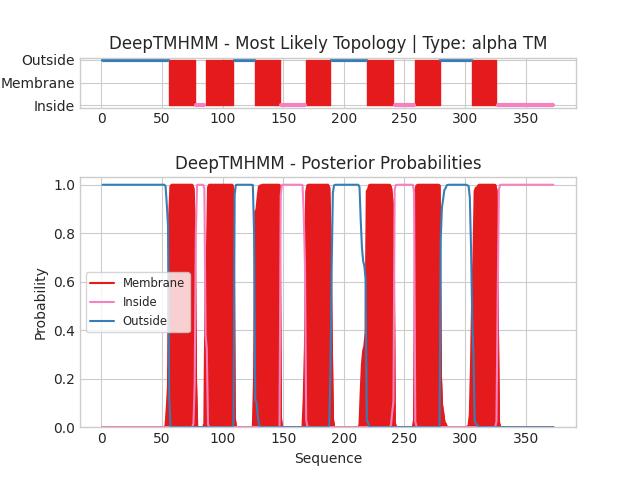
Последовательность для анализа с помощью DeepTMHMM была взята из UniProt(P02943).
Ссылка на выдачу предсказанных программой координат трансмембранных участков.
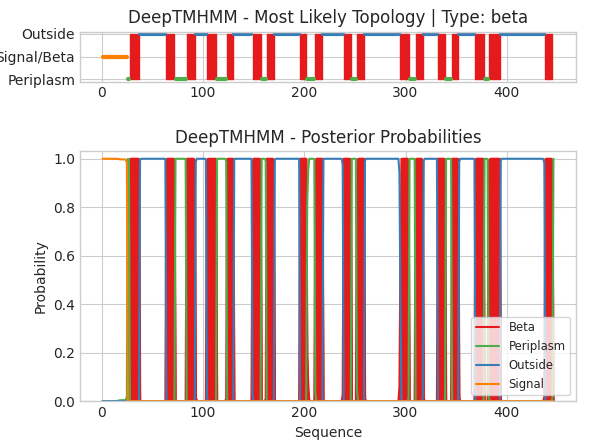
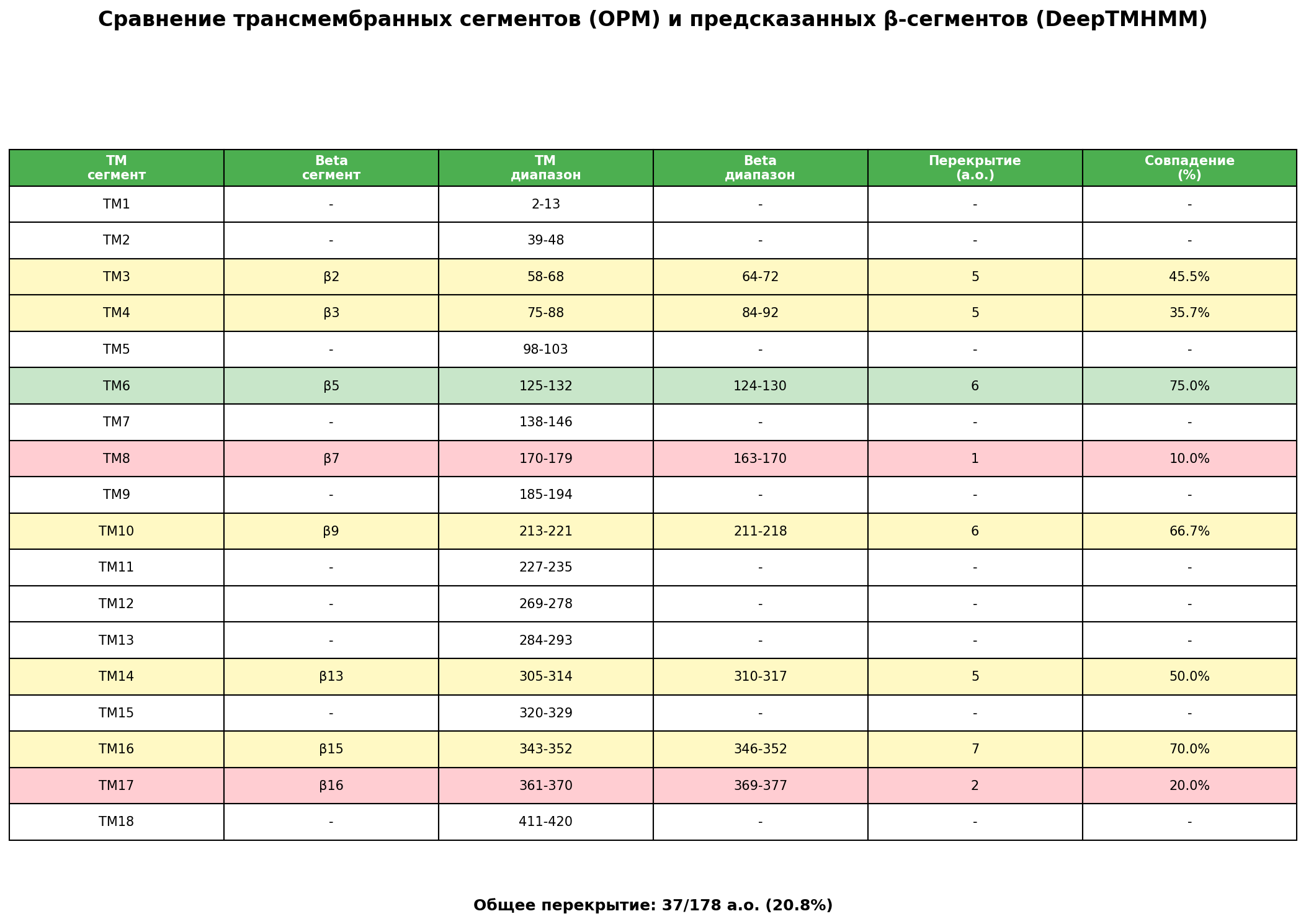
Совпадение низкое - в 20.8%.
Из результатов сравнения можно сделать вывод, что основной причиной расхождения в нумерации аминокислотных остатков между данными OPM и предсказаниями DeepTMHMM является наличие в исходной последовательности UniProt сигнального пептида длиной 25 аминокислот.
OPM использует аннотации для зрелой формы белка, где этого пептида уже нет, в то время как DeepTMHMM работает с полной последовательностью, включая этот участок. Если учесть сдвиг на 25 остатков совпадение позиций сегментов значительно улучшается, что подтверждает данную причину как ключевую.