0. Упражнения по EMBOSS
Из предложенного списка упражнений были выбраны и выполнены необходимые семь. Результаты - как и полагается, в ~/term3/block2/pr9_emboss.txt
1. Написание скрипта
В качестве упражнения было выбрано задание 1: "Проверить, сколько находок с E-value < 0.1 в среднем находит blastn для случайной последовательности данной длины в данном геноме бактерии."
Итоговый скрипт вызывается соответствующей командой и получает на вход название файла и длину для случайных последовательностей первым и вторым аргументом соответственно, а на выход дает число случайных последоватеьностей с E-value больше 0,1 среди находок.
Стоит отметить, что при использовании скрипта на данном в качестве тестового геноме hs_ref_GRCh38.p7_chr22.fa вне зависимости от задаваемой длины последовательности, со средним числом находок около 0,077.
2. Поиск гомологов белков в неаннотированном геноме
Для поиска базовых для всех эукариот белков был предложен неаннотированный геном Amoeboaphelidium protococcarum. На основе его последовательности была подготовлена база данных для BLAST+ командой
makeblastdb X5.fasta -dbtype nucl
Искомыми же были выбраны белки из всего таксона грибы - раз уж исследуемый геном принадлежит их примитивному родственнику, сходства должно быть достаточно. Конкретно белки, выбранные мной как претендующие на присутствие почти у всех эукариот: гистон H3 Saccharomyces cerevisiae (найден по запросу name:"histone h3" taxonomy:"Fungi [4751]" AND reviewed:yes), актин Schizosaccharomyces pombe (запрос name:actin taxonomy:"Fungi [4751]" AND reviewed:yes) и, судя по тому, что в описании таксона Афелид указано, что их митохондрии не редуцированы, цитохром C Aspergillus niger (запрос name:"cytochrome c" taxonomy:"Fungi [4751]" AND reviewed:yes). Все организмы, из которых взяты последовательности, хорошо изучены, так что, как мне кажется, этим записям можно доверять.
Скопировать последовательности в рабочую директурию позволили команды:
seqret sw:P61830 -out 1.fasta
seqret sw:P10989 -out 2.fasta
seqret sw:P56205 -out 3.fasta
Каждую из полученных последовательностей я сравнил при помощи tBLASTn (т.к. нужно выравнять белковые последовательности по нуклеотидной) с базой данных из последовательности интересующего генома, с E-value не более 0,001 и с выдачей в формате удобной для чтения таблицы командами
tblastn -db X5.fasta -query 1.fasta -evalue 0.001 -outfmt 7 -out b1, выдача
tblastn -db X5.fasta -query 2.fasta -evalue 0.001 -outfmt 7 -out b2, выдача
tblastn -db X5.fasta -query 3.fasta -evalue 0.001 -outfmt 7 -out b3, выдача
Первые два запуска алгоритма ознаменовались успехом - в выдаче оказались по несколько схожих участков в базе данных для каждого запроса с очень низкими E-value и высокой идентичностью (от 87%), да и покрытие по образцовому белку полное, так что с большой долей вероятности можно утверждать, что гены гистона Н3 и актина белков полностью или почти полностью присутствуют в данном геноме Amoeboaphelidium protococcarum.
А вот с цитохромом С все не так однозначно. У выдачи с лучшими идентичностью и E-value покрытие чуть больше 70%. Но покрытие явно маловато. Дабы разобраться в ситуации, я нашел последовательность скэффолда с лучшим покрытием и поискал совпадения в blastx - лучшем варианте нуклеотидного бласта при необходимости найти информацию о транслируемом с последовательности белке - скэффолда с максимальным покрытием под номером 287. Так как его последовательность слишком велика для веб-версии БЛАСТа, а сохранять к себе всю базу данных белков даже опистоконт не особо хочется, я взял на вход сервиса только участок, в котором обнаружено сходство, с некоторыми довесками по краям - а вдруг это у модельного организма белок укорочен. Получилась вырезка примерно 469740-471840 bp скэффолда - далеко от его границ, а значит обрыв скэффолда не связан с неполным покрытием. По выдаче, однако, можно понять, что в абсолютном большинстве выравниваний нет соответствия в транскрипте куска скэффолда заведомо определенным последовательностям цитохромов С как раз в позициях 1-31, и, похоже, этот участок оказался в геноме большинства опистоконт (а в выдаче найденные последовательности принадлежат множеству организмов - от комара до гриба пилолистника) или исчез из исследуемого генома позднее, чем произошло расхождение большинства представителей этого таксона с Amoeboaphelidium protococcarum.
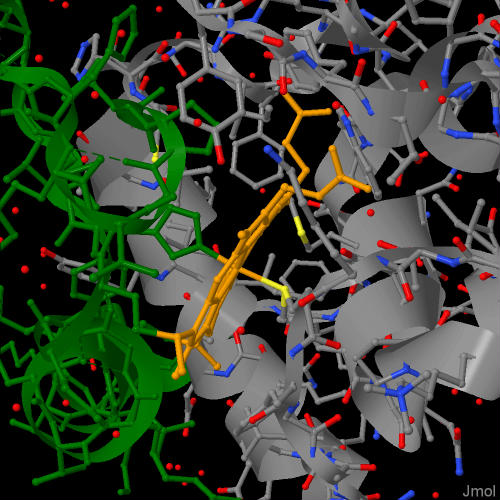
Далее я попытался выяснить, какую функцию выполняет "потерянный" участок. Для этого я нашел в PDB структуру этого же белка у относительно недалекого родственника - Saccharomyces cerevisiae, а затем визуализировал в JMol разницу и функциональную часть цитохрома - гем (рис. 1, 2).

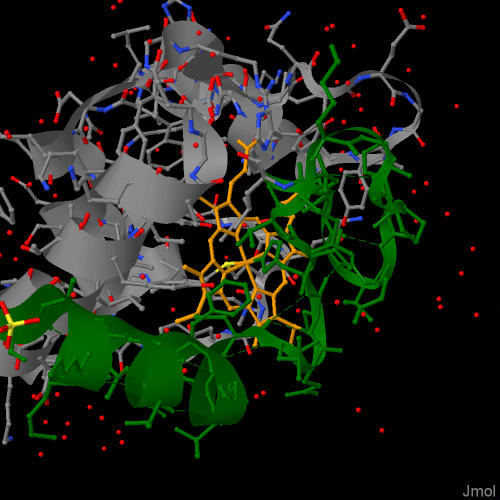
Как видно по картинкам, не найденный в исследуемом геноме участок транскрипта должен быть крайне важен - он в том числе участвует в связывании гема, то есть имеет функциональные участки. Как организм обходится без него - непонятно. Возможно, вопреки моим данным, данный организм все же отказался от использования митохондрий, а значит и цепи переноса электрона, а может быть необходимый участок кодируется другой последовательностью ДНК или представлен сильно измененными аминокислотами.