Визуализация гомологов белка
Гомология
Поиска паралогов белка CLPX_ECOLI был произведен в ранее выбранных бактериях, представленных в таблице 1.бактерии:
| Вид | Мнемоника |
|---|---|
| Bartonella henselae | BARHE |
| Burkholderia cenocepacia | BURCA |
| Escherichia coli | ECOLI |
| Haemophilus influenzae | HAEIN |
| Polynucleobacter asymbioticus | POLAQ |
| Saccharophagus degradans | SACD2 |
| Shewanella denitrificans | SHEDO |
| Yersinia pestis | YERPE |
Протеомы указанных бактерий были скопированы в файл, на основе которого подготовлена база данных для программы Blast командой:
makeblastdb -in bacs -dbtype prot
Далее среди всех протеомов были найдены совпадения с последовательностью CLPX_ECOLI с E-value не меньше 0.001 командой:
blastp -query CLPX_ECOLI.fasta -db bacs -out output.txt -evalue 0.001
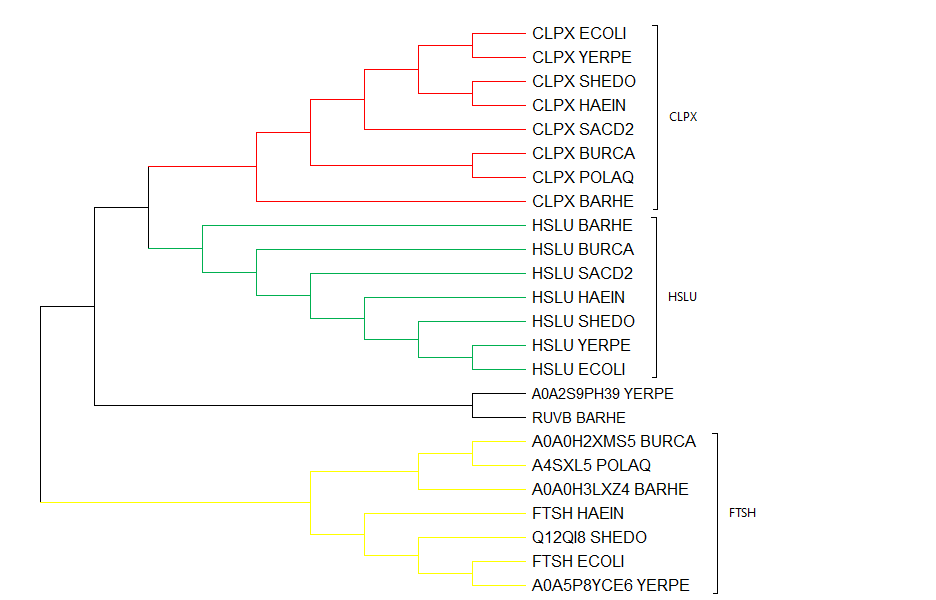
Выдача программы находится тут, краткий список выдачи - тут. Последовательности всех этих белков из Uniprot были выровнены по алгоритму Muscle, выравниванте загружено в MEGA-X. На основе выравнивания по алгоритму максимальной экономии было построено делево, показанное на рисунке 1. Дерево в Newick-формате тут.

На этом дереве отмечены группы белков с одинаковыми (судя по описаниям) функциями. Сразу отмечу, что укроенение дерева я проеделал вручную для большей наглядности группы белков FTSH.
Ортологи
Итак, теперь стоит описать группы ортологов - гомологичных белки из разных организмов, разошедшихся в процессе видообразования и, чаще всего, выполняющих одну и ту же функцию.
CLPX
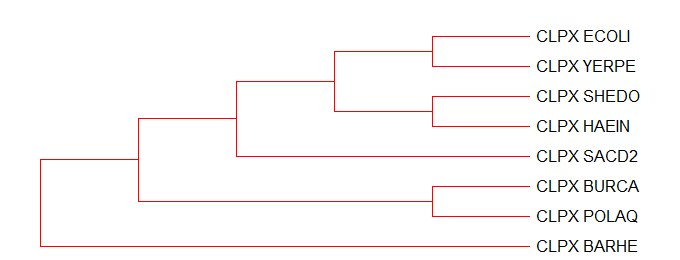
Группа белков с мнемоникой CLPX - это АТФ-связывающая субъединица ClpX АТФ-зависимой протеазы Clp, обладающая шаперонной активностью и готовящая субстраты для транслокации в протеазу ClpP. В эту группу вошли представители всех восьми видов выбранных бактерий. Дерево этой группы белков совпадает с филогенией бактерий за исключением двух ветвей - филогенетически HAEIN ближе к ветви (ECOLI, YERPE), а по дереву ортологов CLPX - к SHEDO.
HSLU
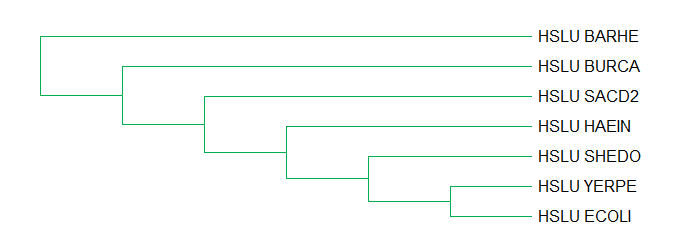
Группа белков с мнемоникой HSLU - это АТФ-азная субъединица HslU АТФ-зависимой протеазы, которая, обладая шаперонной активностью, готовит N-конец своих белковых субстратов к гидролизу в HslV, нарушая его третичную сторуктуру. В эту группу вошли представители 7 из 8 видов выбранных бактерий (не оказалось соответствующего белка из Polynucleobacter asymbioticus). Дерево этой группы белков совпадает с филогенией бактерий, за исключением отсутствия одного листа и двух ветвей - филогенетически HAEIN ближе к ветви (ECOLI, YERPE), SHEDO ортологично этой троице; а по дереву ортологов - наоборот, к ветви (ECOLI, YERPE) ближе SHEDO, а HAEIN им ортологична.
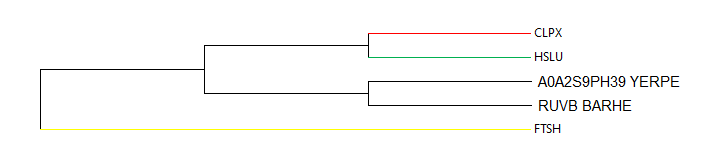
FTSH
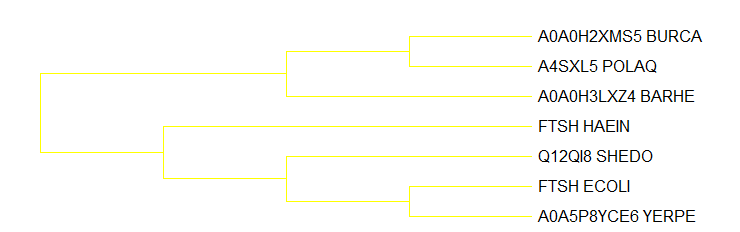
Группа белков с мнемоникой FTSH - это АТФ-зависимая цинковая металлопротеаза FtsH, учавствующая в контроле качества встраиваемых мембранных белков. В эту группу вошли представители 7 из 8 видов выбранных бактерий (на этот раз не оказалось соответствующего белка из Saccharophagus degradans). Дерево этой группы белков, за исключением отсутствия одного листа, не совпадает с филогенией аналогично дереву по HSLU - листья SHEDO и HAEIN поменяны местами.
Стоит также отметить, что деление этих белков на группы ортологов довольно хорошо заметно и на выдаче Blast - так, битовый счет выравниваний CLPX_ECOLI с другими последовательности из группы CLPX находится в пределах 587-805; из группы HSLU - 82-100; а из группы FTSH и для двух не отнесенных к группам белков - в районе 41-46. Из-за этого можно было бы предположить, что несгруппированные белки A0A2S9PH39_YERPE и RUVB_BARHE тоже стоит отнести к FTSH, но, судя по записям на Uniprot, они выполняют совсем другие функции.
Паралоги
Теперь немного обсудим паралоги в полученном дереве. Напомню, паралоги - это гомологичные белки, принадлежащие одному организму, чаще всего возникающие в результате дупликации гена и расходящиеся в процессе эволюции настолько, что начинают выполнять разные функции. Очевидно, по рис. 1 ввиду гомологии всех рассматриваемых белков найти их очень просто - это любые пары белков с одинаковой мнемоникой вида и разной мнемоникой функции. Например, HSLU_BARHE, CLPX_BARHE, A0A0H3LXZ4_BARHE и RUVB_BARHE - паралоги, как и набор CLPX_YERPE, HSLU_YERPE и A0A5P8YCE6_YERP; или пара белков HSLU_ECOLI и CLPX_ECOLI.