Эволюционные домены. Банки Pfam(Protein family) и InterPro
Опиcание доменной архитектуры белка TRMB_BACSU в соответствии с банком Pfam.
База данных Pfam содержит информацию о семействе белков(о группах белков являющихся гомологичными). С её помощью можно опсисать и рассмотреть эволюционные домены(участки консервативных групп в белке) гомологичных белков TRMB_BACSU.
Таблица 1. Доменная структура TRMB_BACSU.
Данные о домене Methyltransf_4
Домен входит в 26 архитектуры, более подбробно о них можно узнать здесь. В базе данных Pfam находятся последовательности 4759 белков и определены пространственная структуры шести различных белков.
Для фрагментов белков, относящихся к домену Methyltransf_4, было получен файл с множественным выравниванием в формате msf. На Рис. 1 представлено изображение выравнивания 19 белков, входящих в состав предыдущего файла.
Рис 1. Множественное выравнивание фрагментов белков, соответствующих домену Methyltransf_4; Color: Clustalx, By Conservation = 33.

Описание частоты встречаемости доменов в организмах по отдельности
Белок TRMB_BACSU имеет одну доменную архитектуру, поэтому рассамтривать, для этих целей, не интересно. Возьмем белок UVRB_BACSU. Результаты представленны в Таблицах 2 и 3.

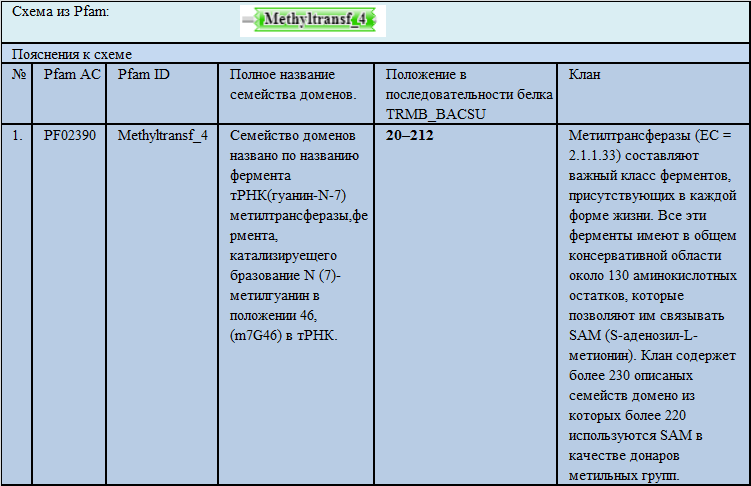
Таблица 2. Представленность домена ResIII (PF04851) в организмах разных таксонов
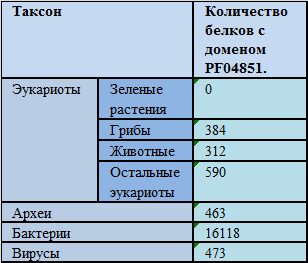
Таблица 2. Представленность домена Helicase_C (PF00271) в организмах разных таксонов
Сравнение описание мотивов в разных банках семейств, по данным InterPro.
InterPro - база данных с информацией о различных семейств белков, доменов, мотивов, сайтах. InterPro база, включающая в свой состав другие база данных(SUPERFAMILY, CATH-Gene3D, PRINTS, PANTHER, Pfam, SMART, PROSIT). На Рис. 2 изобржен результат работы со сравнением особенностей последовательности белка UVRB_BACSU.
Рис 2. Изображение доменоы белка UVRB_BACSU из различных без данных
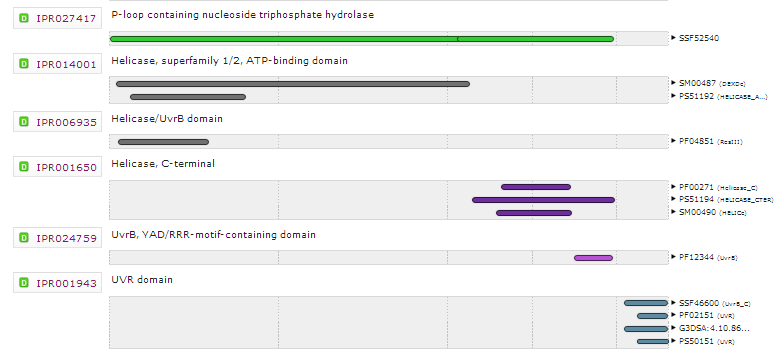
Самый котроткий мотив был PF02151 (UVR), находящийся в базе данных Pfam. SSF52540 является самам большим мотивом, он описан базой SUPERFAMILY. Границы структурных отделов отличаются, например, расмотрим домен Helicase_C(IPR001650), его длина составляет с 459 по 545 аминокислоту в SMART, что отличается от границ домена в Pfam(с 463 по 545).
В InterPro для белка UVRB_BACSU интегрированы следующие структурные подписи:P-loop containing nucleoside triphosphate hydrolase; Helicase, superfamily 1/2, ATP-binding domain; Helicase/UvrB domain; Helicase, C-terminal; UvrB, YAD/RRR-motif-containing domain; UVR domain.