EMBOSS
Программа getorf пакета EMBOSS
Рассмотрим работу программы пакета EMBOSS - getorf. Рассотрим
это на примере одного белка D89965(принадлежащего по словам аторов желудку крысы Rattus norvegicus). Воспользуемся командой entret embl:D89965 для
получения файла с записью D89965 банка EMBL, для дальнейшей работы. Как указано на сервере
kodomo: программа getorf извлекает из нуклеотидныой последовательности открытые рамки считывания.
Воспользуемся командой:
getorf D89965.fasta -minsize 90 -table 0 -find 1 такой записью мы сказали програме использовать файл D89965.fasta
(файл с последовательностью нуклеиновой кислоты); -minsize 90 - c минимальной длину orf - 90 нуклеотидов; -table 0 - с использованием стандартный генетический код;
-find 1 - выбираем нужный тип поиска: orf начинается со старт-кодона и заканчивается стоп-кодоном.
Таким образом, мы поличили следующий набор orf:
>D89965_1 [66 - 155] Rattus norvegicus mRNA for RSS, complete cds. MQFHPRLPAVLQVCAACDRYASLLPAQRRL >D89965_2 [56 - 169] Rattus norvegicus mRNA for RSS, complete cds. MISDAVSSATASSASSLRSMRSVRQSFASSTAALTRWP >D89965_3 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds. MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM AVTAYAYYSCHELTPWLRIQSTNPVQKYGA >D89965_4 [218 - 3] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds. MLLRCSNCLNVNWKCIRAIWSKPPLSWQKTGVPIACCANLKHCWQSRMKLHRLSSPVTVT WCSQKTILLLSA >D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds. MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
Можно заметить, что последовтельность 3(>D89965_3) совпадает с записью в EMBL. В свою очередь записть EMBL имеет ссылку на записаь в базе данных Swiss-Prot(индентификатор P0A7B8). Она является записью белка HSLV_ECOLI, котроая принадлежит организму отличного от прежденего, кишечной палочки.
Эта последовательность прекрывается с одной из найденых ранее (orf в записи D89965 - >D89965_5), что означает их идентичность на участке. Получилось что два очень разных по таксономии организма имеют одинаковые белки.Как тваое могло произойти?
Не стоит забывать о месте обитания кишечной палочки в организме млекопитающих. Она обитает в различных участках желудочного тракта. И получается, что в ходе эксперемента была сиквенирована последовательность бактериального гена, вместо той которая планировалась в эксперементе. К счастью, база данных Swiss-Prot является аннотированной и её стоит доверять больше, чем архивной базе данных EMBL.
Итог: ошибка в интерпретации результатов секвинирования(бактериальная мРНК была принята за крысиную) и как следствие ошибка поиска ORF.
Файлы-списки
seqret sw:adh*_* позволяет получить все последовательности алкогольдегидрогеназ из Swiss-prot;infoseq -only -usa > list.txt позволяет получить список всех универсальных адресов USA;grep list.txt -f org.txt > select_org.txt позволяет получить список только тех организмов, которые
есть в файле organisms;seqret @select_org.txt final_adh.fasta позволяет извлечь из файла adh.txt только те последоваиельности, названия которых содержатся в файле select_org.txt.
В итоге имее файл.EnsEMBL
Расмотрим ген MED13_HUMAN. Для поиска информации об этом гене стоит использовать портал EnsEMBL.
Прямой поиск: добавление в поисковую строку название гена, дал два результата. Из них можно получить следующую информацию: расположение, координаты, структура, длина, варианты сплайсинга и много чего еще.
Пример:
Последовательность эконов и трансируемые части можно увидеть пройдя по этой ссылке. Как можно заметить при наведениии на определенный нуклеотид в последовательности сразу подсвечивается триплед в состав которого она входит и аминокислота которая им кодируется. Работает и обратный принцып с белковыми остатками последовательности.
В ходе работы с сайтом, был получен длинный экзон гена MED13_HUMAN:
TGATGGAAGTAATATGGATCAAGAATATGGCACTGCTTATACACCTCAAACT
CATACTTCTTTTGGGATGCCTCCTAGCAGTGCACCTCCTAGTAACAGCGGAGCAGGAATTCTTCCTTCTC
CATCCACCCCTCGGTTTCCAACTCCAAGGACTCCAAGGACTCCTCGGACTCCTCGTGGAGCTGGTGGACC
TGCTAGTGCTCAAGGTTCAGTCAAATATGAAAATTCAGACTTGTATTCACCAGCTTCTACCCCATCTACA
TGCAGACCCCTTAATTCTGTTGAACCTGCAACTGTCCCTTCCATCCCTGAAGCACACAGTCTTTATGTAA
ACCTCATCCTTTCAGAATCAGTTATGAATTTGTTTAAAGACTGTAACTTTGATAGTTGTTGCATCTGTGT
TTGCAACATGAACATCAAGGGTGCCGATGTTGGAGTTTACATTCCAGATCCAACGCAGGAAGCACAATAT
AGGTGTACCTGTGGCTTCAGTGCTGTCATGAACAGAAAATTTGGAAACAATTCAGGATTATTTCTTGAAG
ATGAACTAGATATCATAGGACGCAATACAGACTGTGGCAAAGAAGCAGAAAAACGTTTTGAAGCTCTCAG
GGCTACCTCTGCTGAACATGTTAATGGAGGACTAAAGGAATCTGAAAAATTATCTGATGATTTGATATTA
TTGCTACAAGATCAGTGCACTAATTTATTTTCACCCTTTGGAGCAGCAGACCAAGATCCTTTTCCTAAAA
GTGGTGTAATTAGCAATTGGGTACGTGTTGAAGAGCGTGACTGTTGCAATGACTGCTACCTTGCATTAGA
ACATGGGCGTCAGTTCATGGATAACATGTCAGGAGGAAAAGTTGATGAAGCACTTGTGAAAAGTTCATGC
TTACACCCCTGGTCCAAAAGAAACG
С помощью инструментов поиска BLAST/BLAT было установлено место расположение этого участка в хромосоме(см. Рисунок 3).
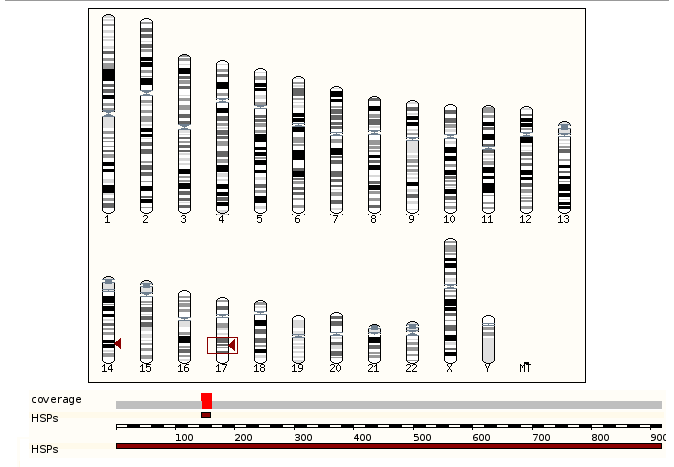
Рисунок 3. Локализация экзона в хромосоме, в красной рамке заключена лучшая находка.
Используя ссылку ContigView (символ [C]), мы переходим на страницу с изображением расположения данного экзона(см. Рисунок 4).
Стоит также отметить, что на портале есть возможность простроения генетических деревье. Можно заметить, что в таком случае проще искать организмов являющимися близими по данному гену.