Построение дерева по нуклеотидным последовательностям
Построение дерева по нуклеотидным последовательностям
В этом разделе попробуем построить филогенитическое дерево тех же организмов, которые были выбраны ранее, по их нуклеотидным последовательностям. Для этого мы, как и в предыдущем задании будем использовать знания о малой РНК субъединицы рибосомы (16S rRNA). Ниже в таблице представленна информация о 16S rRNA в каждом организме выборки.

Таблица 1 В таблице для каждой бактерии указан AC записи EMBL, в которой нашлась последовательность 16S rRNA, координаты этой РНК в этой записи, на прямой или комплементарной последовательности оказалась последовательность этой рРНК.
Полученные последовательности были выравнены программой Muscle. На освнове множественного выравнивания muscle, было построено дерево программой MEGA по алгоритму максимального правдоподобия.
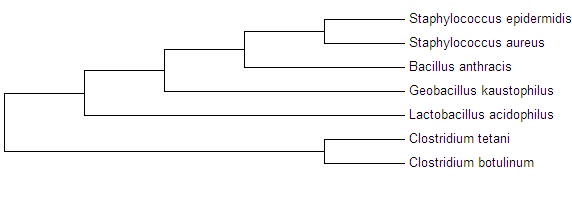
Изображение 1 Дерево бактерий, построенное программой MEGA по алгоритму максимального правдоподобия на основе множественного выравнивания программой muscle 16S РНК.
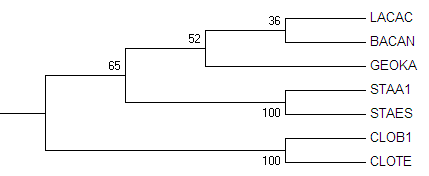
Изображение 1 Верное дерево
Как видно деревья отличаются. Только две совпадающие ветви. Дерево по нуклеотидной последовательности построенно хуже, чем то что по было простенно по белкам.
Построение и анализ дерева, содержащего паралоги
Для начала, введем определения. Два гомологичных белка мы будем называть ортологами, если они были получены из разных организмов или разделение их общего предка на линии, произошло в результате видообразования. А паралогами будем называть гомологичными белка из одного организма.
Используя файл proteo.fasta, содержащий записи банка UNIPROT, относящиеся ко всем бактериям используемых в задании этого блока, был проведен поиск гомологов. Программой blastp(с порогом на E-value 0,001) и отобраны только те бактерии которые использовались мою ранее в заданиях. На основе множественного выравнивания, было простроенно дерево.

Изображение 1Дерево гомологов белка CLPX_BACSU построенное программой MEGA по алгоритму максимального правдоподобия на основе множественного выравнивания программой muscle.
Среди найденых гомологов есть ортологи: CLPX_STAES и CLPX_STAA1; CLPX_GEOKA и CLPX_BACAN и т.д. Так же есть и паралоги, например: CLPX_BACAN и B0AY46_BACAN. Интересно, что на этом дереве так же можно наблюдать пример дупликации генов: HSLU_BACAN и C3F5U0_BACAN, и пример разделение путей эволюции белков в результате видообразования: Q5L436_GEOKA и A7WYU0_STAA1.