Анализ трехмерных структур
Определение вторичной структуры (d1)
Для работы был выбран белок TenI с PDB-кодом 3QH2, использовалась программа DSSP (выдача программы).
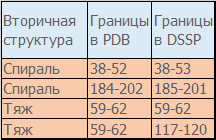
Таблица 1 Сравнение границ элементов вторичной структуры.
Из Таблицы 1 видно, что определенные в DSSP и в PDB-файле границы бэта-листов совпадают полностью. Одна из рассмотренных спиралей отличается на один остаток, вторая – на два остатка. Это тоже очень хорошее совпадение в определении границ.
SheeP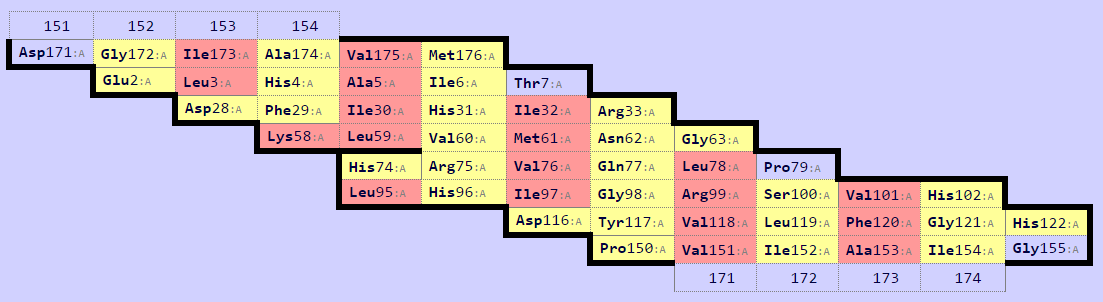
Рисунок 1 Карта бэта-листа цепи А структуры 3QH2
Изображние выдаеваемое программой SheeP(Рисунок 1) представлеят карту укладки бэта-листа. А далее с помощью программы Jmol получилось некрасивая, но информативная картинка об укладки этого листа.
С помощью детектора бэта-листов STRIDE на сайте SheeP была получена карта водородных связей, а в консольной версии - список водородных связей.
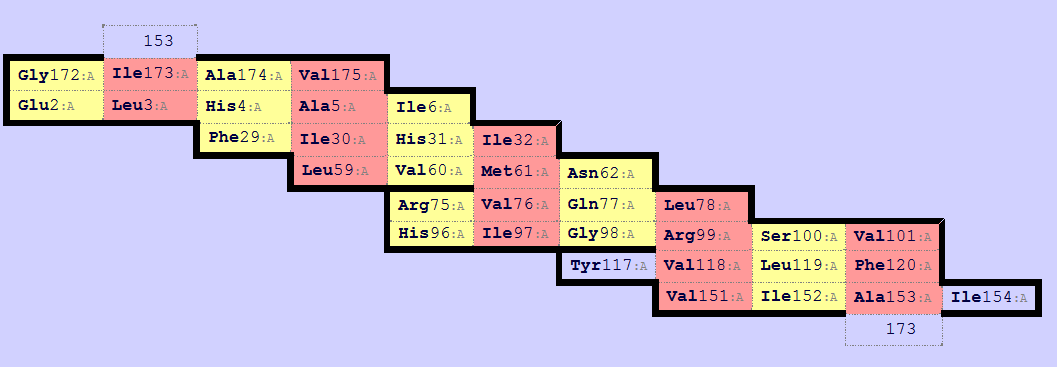
Рисунок 2 Расположение водородных связей структуры 3QH2
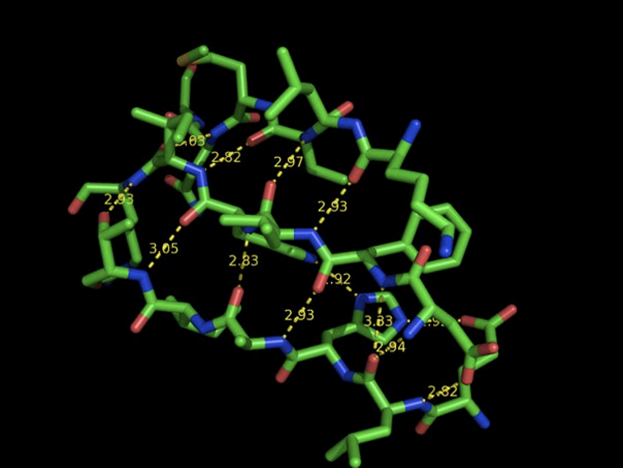
Рисунок 3Расположение водородных связей структуры 3QH2
Совмещение структур (d2)
С помощью PDBeFold были найдены структурные гомологи для всего белка 3QH2(SMM: 70-70): (ниже указаны струкутры, цепи и RMSD )
PDB 3qh2:A 1.1360
PDB 1yad:C 1.0829
PDB 1g69:A 1.3937
PDB 1wa3:B 1.4386
PDB 2v82:A 1.4547
Структурное выравнивание PDBeFold этих структру представленно ниже

Рисунок 4 Визуализация пространственного совмещения 3QH2 и структурных гомологов (с помощью JMol на PDBeFold)
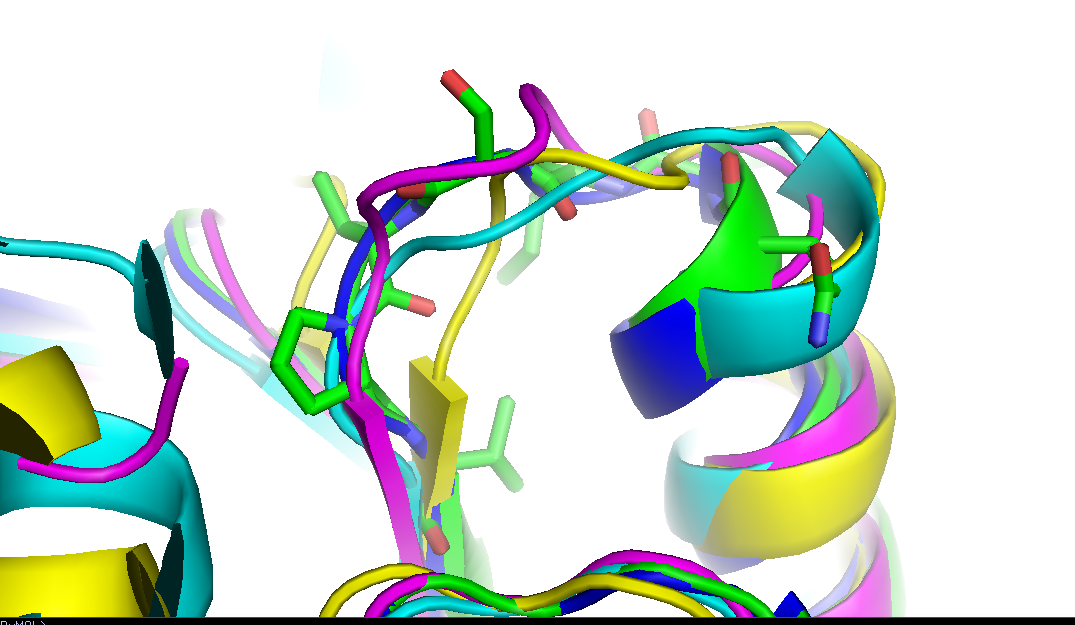
Рисунок 5 Визуализация пространственного совмещения 3QH2 и структурных гомологов в области плохого выравнивания(петля)
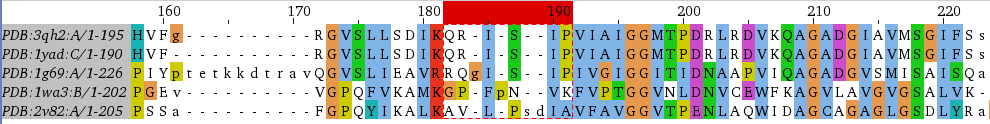
Рисунок 6 Визуализация выравнивания 3QH2 и структурных гомологов в области плохого петли(см. выше)
Поиск по сходству в PDBeFold
Для работы был выбран самый первый домен из списка – 1В0М А:203-315. Результаты отсортированы по RMSD. Поиск осуществлялся при параметрах, заданных по умолчанию.
Совмещение по заданному выравниванию
Для работы были выбраны опять же самые первые в списке константные домены Т-клеточного рецептора 1OGA из альфа-цепочки регион d:118-202 и из бэта-цепочки регион е:119-245.
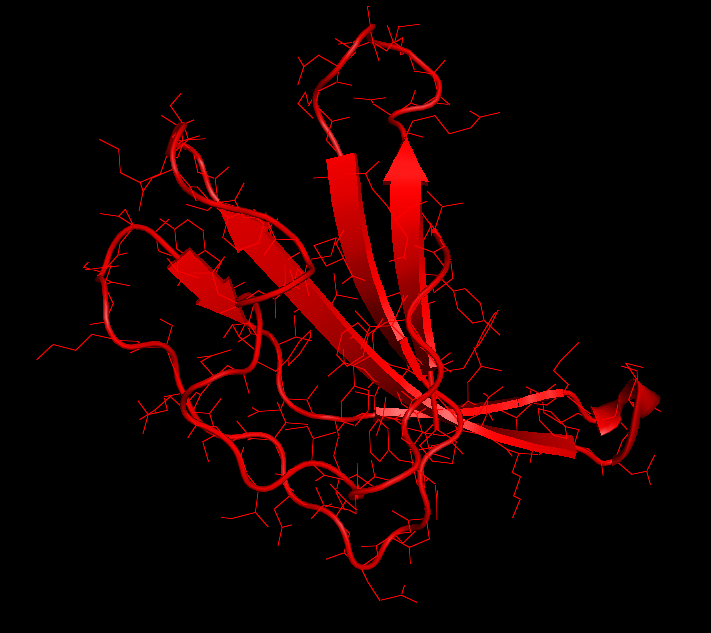
Рисунок 7 Константные домены Т-клеточного рецептора 1OGA(d:118-202)
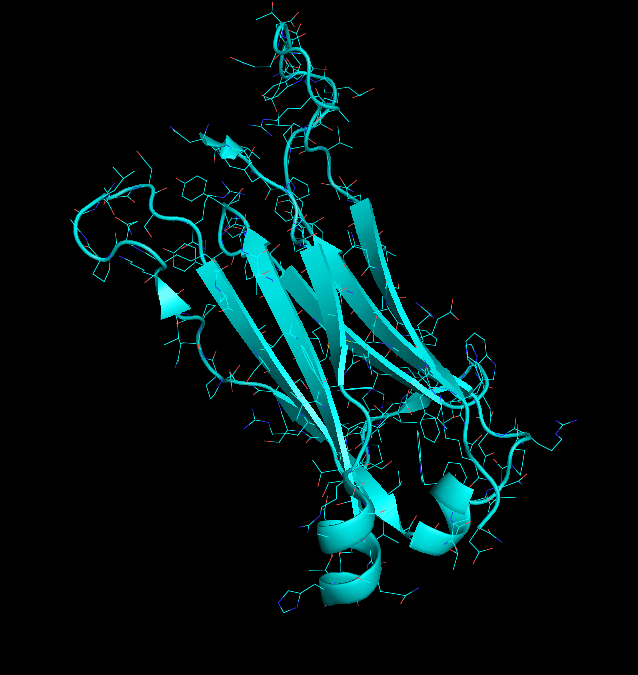
Рисунок 8 Константные домены Т-клеточного рецептора 1OGA(е:119-245)
Карты бэта-листов для выбранных доменов были построены с помощью SheeP. Далее были выбраны соответствующие друг другу бэта-листы из альфа-цепочки и из бэта-цепочки.
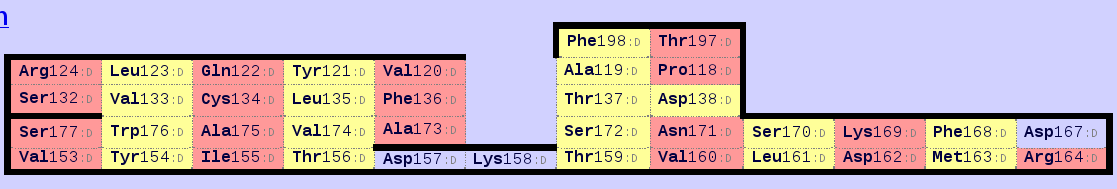
Рисунок 9 Карты бэта-листов константных доменов Т-клеточного рецептора 1OGA(d:118-202)
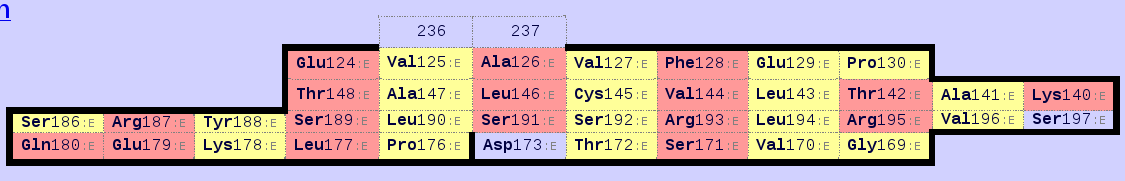
Рисунок 10 Карты бэта-листов константных доменов Т-клеточного рецептора 1OGA(е:119-245)
Предоставленные карты находятся в одной ориентации. С помощью команды PyMol было построено совмещение

Рисунок 11 Выравнивание"аналогичных" листов, константных доменов, Т-клеточного рецептора 1OGA(с помощью команды pair_fit )
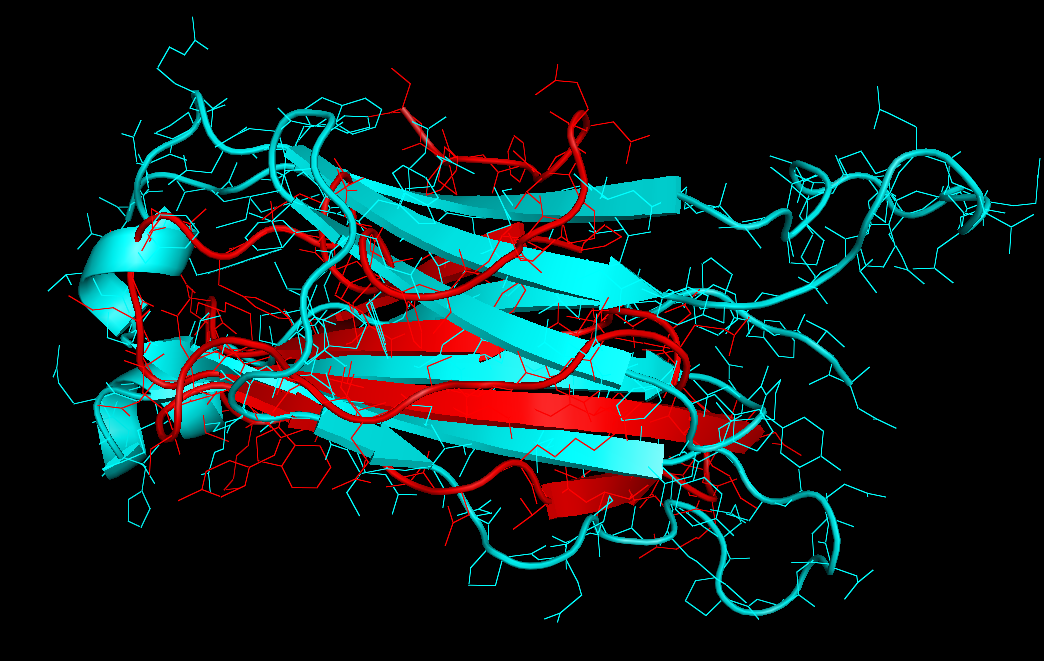
Рисунок 11 Налодение(совмещение)"аналогичных" листов, константных доменов, Т-клеточного рецептора 1OGA(с помощью команды align)
Анализируя структурное выравнивание, можно заметить, что топологии структур(доменов) хорошо совпадают.
Нахождение гидрофобные кластеры (d3)
С помощью ресурса. Clud был произведен поиск гидрофобных кластеры в структуре 3QH2.
.png)
Рисунок 12 Гидрофобные кластеры в структуре 3QH2 (clusters not smaller: 3, distance threshold: 4)
.png)
Рисунок 12 Гидрофобные кластеры в структуре 3QH2 (clusters not smaller: 3, distance threshold: 5.8)
.png)
Рисунок 13 Гидрофобные кластеры в структуре 3QH2 (clusters not smaller: 3, distance threshold: 10)
.png)
Рисунок 14 Гидрофобные кластеры в структуре 3QH2 (clusters not smaller: 10, distance threshold: 5.4)
.png)
Рисунок 15 Гидрофобные кластеры в структуре 3E7K (clusters not smaller: 3, distance threshold: 5.4)
Построение поверхности (d4)
Для работы был выбран комплекс димера пуринового репрессора с ДНК 1BDH. Визуализация выполнена с помощью PyMol.
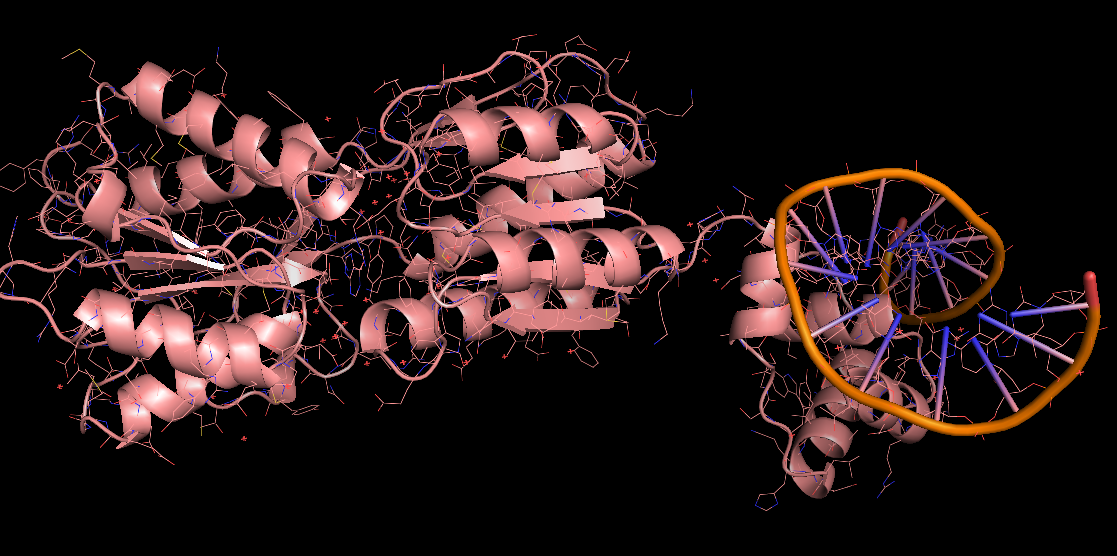
Рисунок 16 Комплекс димера пуринового репрессора с ДНК 1BDH
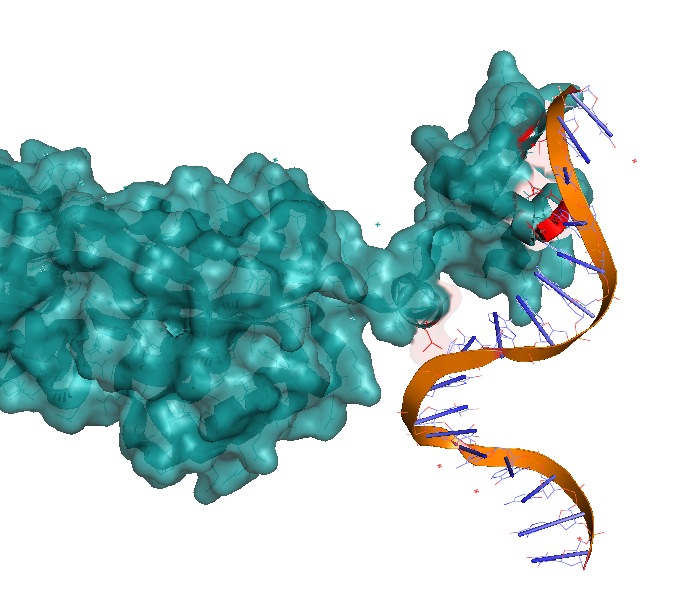
Рисунок 17 Визуализация поверхности контакта димера с ДНК
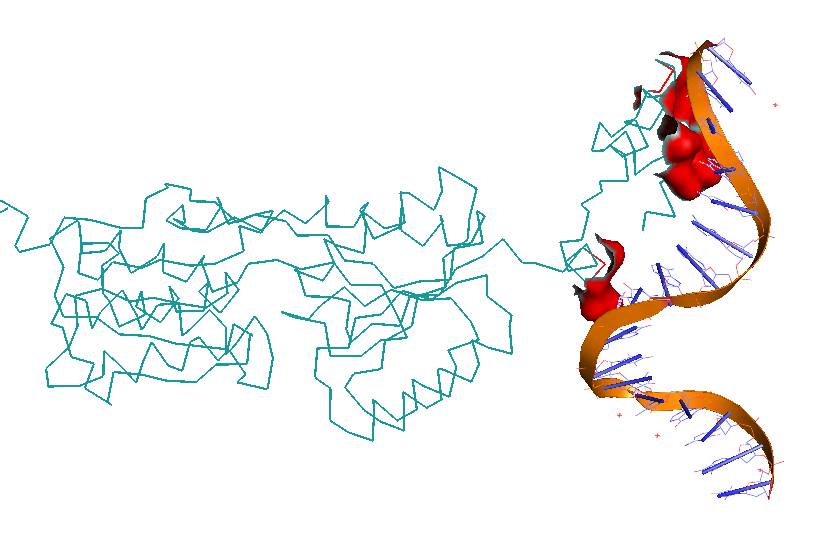
Рисунок 18 Визуализация поверхности контакта димера с ДНК на фоне остовной модели части белка
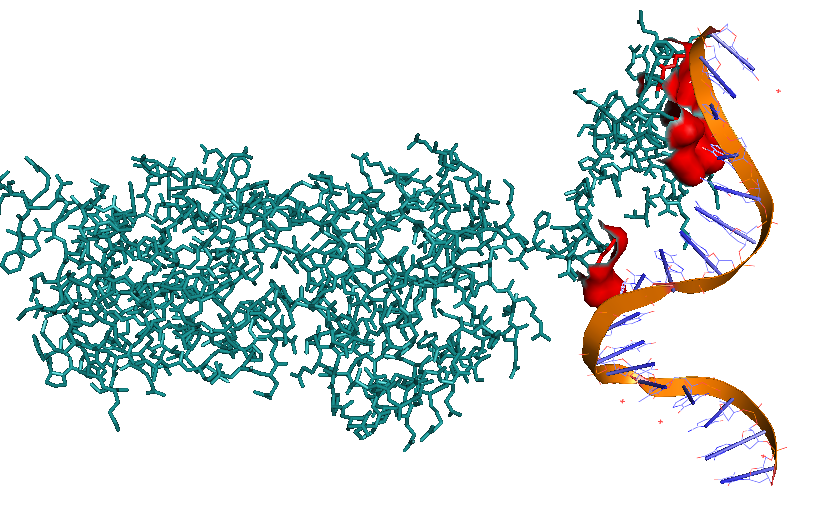
Рисунок 19 Визуализация поверхности контакта димера с ДНК формат stiks
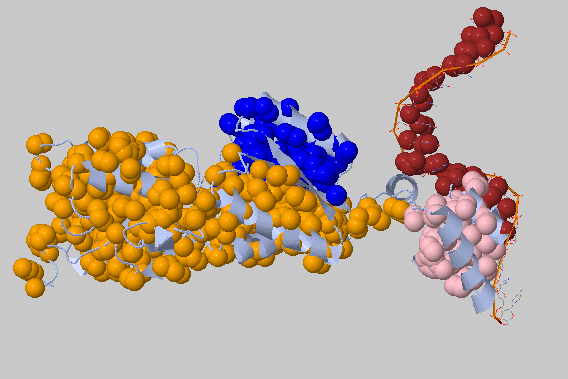
Рисунок 20 Визуализация гидрофобных ядер в комплекс димера пуринового репрессора с ДНК 1BDH
Сравнение доменов (d5)
Структуру 1GD1 CATH относит к Superfamily 3.30.360.10 Dihydrodipicolinate Reductase и утверждает, что она имеет 2 домена(domain 2).