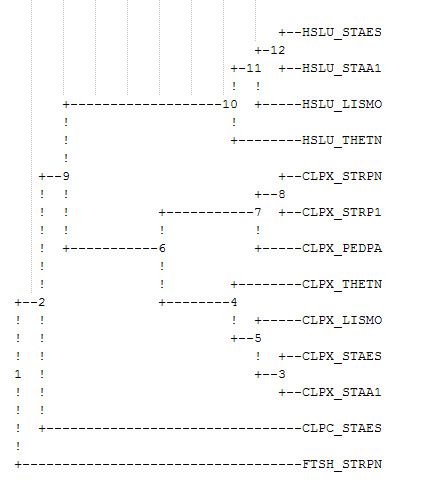
| Мнемоника | AC записи EMBL | Координаты FT, в которой описано 16S рРНК |
| LISMO | AL591981 | complement(99187..100732) |
| PEDPA | CP000422 | 116896..118475 |
| STAA1 | BX571856 | 514251..515805 |
| STAES | AE015929 | complement(2380573..2382126) |
| STRP1 | AE014074 | 17169..18503 |
| THETN | AE008691 | 53858..55384 |