

| PDB код | Число цепей | Тип
(спираль, баррель) |
Число трансмембранных участков в цепи | Число остатков в одном трансмембранном участке
(типичное, минимальное, максимальное) |
(*) Толщина мембраны в ангстремах
(расстояние между атомами на границах трансмембранных участков |
| 1zll | 5 | спираль | 5 | 25 | 30.5 ± 1.4 A |
| 3fhh | 1 | баррель | 22 | 6-8 (типично 7) | 24.6 ± 0.9 A |
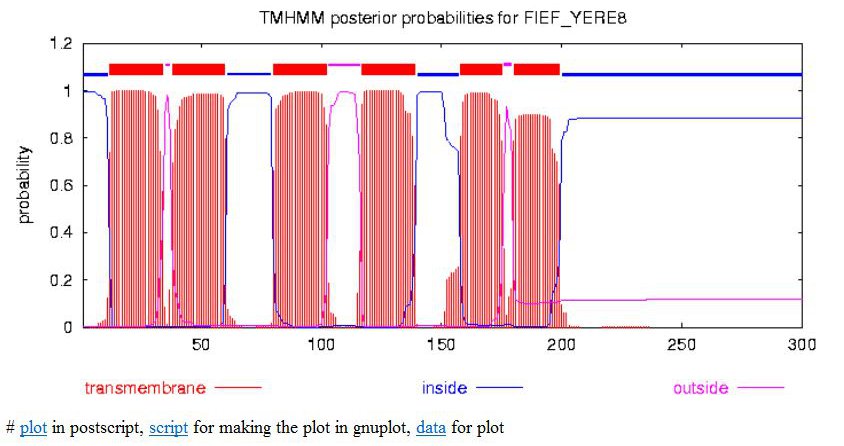
Координаты α-спиралей, предсказанные Uniprot и ТМНММ для A1JHZ2 (мой белок)
| Uniprot, координаты | Uniprot, длина | ТМНММ, координаты | ТМНММ, длина | Гомолог, коордитаны | Гомолог, длина |
| 12-32 | 21 | 12-34 | 23 | 12-34 | 23 |
| 40-60 | 21 | 38-60 | 23 | 38-60 | 23 |
| 82-102 | 21 | 80-102 | 23 | 80-102 | 23 |
| 114-134 | 21 | 117-139 | 23 | 117-139 | 23 |
| 164-184 | 21 | 158-175 | 18 | 158-175 | 18 |
| 180-199 | 20 | 180-199 | 20 |