Отчет студента 2 курса ФББ Кузеванова
Алексея Владимировича по работе
«Предсказание генов».
|
Создайл в
директории Term3 поддиректорию Practice12. Завел файл отчета megacounterfeit.doc. В
директории P:\y05\Term3\GeneRecognition нашёл два файла: ecoli_kuzevanov.txt — фрагмент последовательности ДНК
Escherichia coli в
формате EMBL (с аннотацией); human_kuzevanov.txt — фрагмент последовательности
ДНК человека в формате plain. Скопировал эти файлы в свою рабочую директорию. Часть 1. Поиск прокариотических
генов 1). Мне
был дан фрагмент последовательности ДНК Escherichia coli и аннотация к нему в
формате EMBL. Мне нужно
было идентифицировать гены в этом фрагменте при помощи программ ORF Finder и
GeneMark и сравнить
полученные результаты с аннотацией. Результаты я оформил в виде таблицы:
2). С
помощью программы ORF Finder идентифицировал открытые рамки считывания в
последовательности ДНК Cкопировал
последовательность ДНК в текстовое поле формы и нажал кнопку OrfFind. Получил
список обнаруженных открытых
рамок. Щёлкнул мышью найденную ORF, чтобы получить её более подробное
описание. Нажал кнопку BLAST,
чтобы найти последовательности, похожие на транслированную ORF, в банке
белковых последовательностей nr и
кнопку Format! в открывшемся окне. Начало,
конец и рамку для самых длинных не пересекающихся по ДНК предсказаний занес в
такую же таблицу, как в пункте
1). Выделил зелёным цветом строки таблицы, соответствующие предсказанным ORF,
точно совпадающим с аннотированными генами. Приложил к отчёту
выравнивание, соответствующее лучшему хиту blastp.
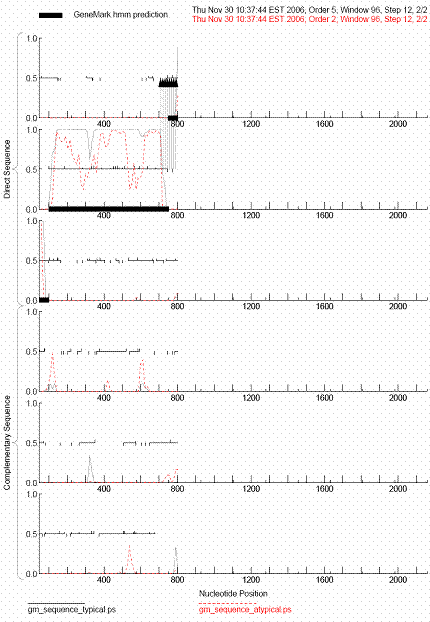
3). С
помощью программы GeneMark распознал гены в последовательности ДНК Скопировал
последовательность ДНК в поле Sequence Text. Включил опции Print
GeneMark 2.4 predictions in addition to GeneMark.hmm predictions и Generate
PDF graphics (screen). Запустил
программу кнопкой Start
GeneMark.hmm. Получил таблицы генов, предсказанных двумя программами:
GeneMark и GeneMark 2.4. В отчете
использовал результаты работы GeneMark 2.4. Занес
результаты работы программы GeneMark 2.4 в такую же таблицу, как в пункте 1).
Знак < или > возле границы
предсказанного гена означает, что программа предполагает продолжение гена за
пределами данной ей
последовательности. Выделил зелёным цветом строки таблицы, соответствующие
предсказаниям, точно
совпадающим с аннотацией. Посмотрел,
как распределен кодирующий потенциал по ДНК, для этого нажал гиперссылку View PDF
Graphical Output на странице с предсказаниями GeneMark. На открывшемся PDF-рисунке
увидел графики
распределения кодирующего потенциала для каждой рамки считывания на обеих
цепях ДНК.
Приложил график к отчёту.
Часть
2. Поиск эукариотических генов 1). Мне
дан фрагмент ДНК из генома человека, содержащий альтернативно сплайсируемый
ген. Моя задача — найти две
различные изоформы этого гена (неодинаковые выранивания двух белков с ДНК) и
некодирующие экзоны,
используя программы GENSCAN, BlastX и Human Genome Browser (HGB). С помощью
программы GENSCAN выделил экзоны в последовательности ДНК и определите их
тип. Скопировал
последовательность ДНК в текстовое поле формы и нажал кнопку Run GENSCAN.
Программа GENSCAN
представила результаты в виде таблицы экзонов. Занес в свою таблицу начало,
конец и тип всех предсказанных
программой экзонов:
2).
Выделение экзонов в последовательности ДНК с помощью программы BlastX и сравнение предсказания
программ GENSCAN и BlastX В меню "Choose database" оставил предложенный по умолчанию
банк nr. Ниже в опциях нашел меню, позволяющее ограничить поиск только какой-нибудь
одной таксономической группой, и выбрал в нём позвоночных (Vertebrata). Отключил
фильтр малой сложности (low complexity). Программа
BlastX
предсказала изоформы выданного гена. Нашел
изоформы, различающиеся по числу экзонов. Выберал две изоформы с разным
числом экзонов. Одна из них порождена белком, принадлежащим не человеку.
В отчете
для каждой изоформы привел выравнивание и отдельную таблицу координат "экзонов"
на белке и ДНК: Возникли
проблемы с заполнением таблицы: так как в каждой изоформе находится очень
большое количество «экзоов», было
очень трудно заполнить таблицу и вычислить какие «экзоны» перекрываются,
соответственно было трудно определить
какие «экзоны» не встречаются, сопоставляя таблицы координат «экзонов» по
двум изоформам. Для
заполнения таблицы выбрал наиболее большие «экзоны», точнее мной было выбрано
6 первых. Покрасьте в этих таблицах красным
цветом отличия изоформ — альтернативные экзоны. Альтернативными называются экзоны,
которые либо отсутствуют в другой изоформе, либо покрывают собой интрон,
идентифицированный в другой изоформе, либо имеют альтернативный 5'- или 3'-конец.
Внимание: из-за описанной выше неточности программы BlastX отличия концов "экзонов" на 1–10 нуклеотидов не могут быть
сочтены настоящей альтернативой. gi|46361987|ref|NP_996995.1| gi|45708670|gb|AAH33796.1| gi|119606136|gb|EAW85730.1|
hydroxyacylglutathione hydrolase-like, isoform CRA_f [Homo sapiens] Length=290 Score = 271
bits (692), Expect = 3e-71 Identities =
124/124 (100%), Positives = 124/124 (100%), Gaps = 0/124 (0%) Frame = +1 Query
1540
KVFCGHEHTLSNLEFAQKVEPCNDHVRAKLSWAKARPLSRRGKRVGGEGTGFGVGGALRQ 1719
KVFCGHEHTLSNLEFAQKVEPCNDHVRAKLSWAKARPLSRRGKRVGGEGTGFGVGGALRQ Sbjct
167
KVFCGHEHTLSNLEFAQKVEPCNDHVRAKLSWAKARPLSRRGKRVGGEGTGFGVGGALRQ 226 Query
1720
GLMVTGACGHSRRGMRMTCPLCRRLWARSASTTPSCGWREYGCCPGASTVTWTLRKASGD 1899 GLMVTGACGHSRRGMRMTCPLCRRLWARSASTTPSCGWREYGCCPGASTVTWTLRKASGD Sbjct
227
GLMVTGACGHSRRGMRMTCPLCRRLWARSASTTPSCGWREYGCCPGASTVTWTLRKASGD 286 Query
1900 CVLG 1911
CVLG Sbjct
287 CVLG 290 Score = 88.6
bits (218), Expect(3) = 2e-50 Identities =
37/38 (97%), Positives = 38/38 (100%), Gaps = 0/38 (0%) Frame = +3 Query
1059
QFGAIHVRCLLTPGHTAGHMSYFLWEDDCPDPPALFSG
1172
+FGAIHVRCLLTPGHTAGHMSYFLWEDDCPDPPALFSG Sbjct 96
RFGAIHVRCLLTPGHTAGHMSYFLWEDDCPDPPALFSG
133 Score = 82.0
bits (201), Expect(3) = 2e-50 Identities =
40/42 (95%), Positives = 40/42 (95%), Gaps = 0/42 (0%) Frame = +2 Query
863
DHARGNPELARLRPGLAVLGADERIFSLTRRLAHGEELRVSA 988
DHARGNPELARLRPGLAVLGADERIFSLTRRLAHGEELR A Sbjct 58 DHARGNPELARLRPGLAVLGADERIFSLTRRLAHGEELRFGA 99 Score = 74.3
bits (181), Expect(3) = 2e-50 Identities =
37/42 (88%), Positives = 37/42 (88%), Gaps = 4/42 (9%) Frame = +1 Query
1237
PPPP----GDALSVAGCGSCLEGSAQQMYQSLAELGTLPPET 1350 P
PP GDALSVAGCGSCLEGSAQQMYQSLAELGTLPPET Sbjct
125
PDPPALFSGDALSVAGCGSCLEGSAQQMYQSLAELGTLPPET 166 Score = 71.2
bits (173), Expect = 5e-11 Identities =
37/44 (84%), Positives = 39/44 (88%), Gaps = 0/44 (0%) Frame = +1 Query
256 MKVKVIPVLEDNYMYLVIEELTREAVAVDVAVPKRVRAGRGPQG 387
MKVKVIPVLEDNYMYLVIEELTREAVAVDVAVPKR+
G +G Sbjct 1
MKVKVIPVLEDNYMYLVIEELTREAVAVDVAVPKRLLEIVGREG 44 Score = 52.4
bits (124), Expect = 3e-05 Identities =
28/41 (68%), Positives = 29/41 (70%), Gaps = 2/41 (4%) Frame = +3 Query
702
PFQLLEIVGREGVSLTAVLTTHHHW*APAGRGEARGRRLVP 824 P
+LLEIVGREGVSLTAVLTTHHHW RG RL P Sbjct 33
PKRLLEIVGREGVSLTAVLTTHHHW--DHARGNPELARLRP 71 gi|109127081|ref|XP_001087099.1| Length=282 Score = 87.4
bits (215), Expect(3) = 4e-50 Identities = 36/38
(94%), Positives = 38/38 (100%), Gaps = 0/38 (0%) Frame = +3 Query
1059
QFGAIHVRCLLTPGHTAGHMSYFLWEDDCPDPPALFSG
1172
+FGAIHVRCLLTPGHT+GHMSYFLWEDDCPDPPALFSG Sbjct 96
RFGAIHVRCLLTPGHTSGHMSYFLWEDDCPDPPALFSG
133 Score = 82.0
bits (201), Expect(3) = 4e-50 Identities =
40/42 (95%), Positives = 40/42 (95%), Gaps = 0/42 (0%) Frame = +2 Query
863
DHARGNPELARLRPGLAVLGADERIFSLTRRLAHGEELRVSA 988
DHARGNPELARLRPGLAVLGADERIFSLTRRLAHGEELR A Sbjct 58 DHARGNPELARLRPGLAVLGADERIFSLTRRLAHGEELRFGA 99 Score = 74.3
bits (181), Expect(3) = 4e-50 Identities =
37/42 (88%), Positives = 37/42 (88%), Gaps = 4/42 (9%) Frame = +1 Query
1237
PPPP----GDALSVAGCGSCLEGSAQQMYQSLAELGTLPPET 1350 P
PP GDALSVAGCGSCLEGSAQQMYQSLAELGTLPPET Sbjct
125
PDPPALFSGDALSVAGCGSCLEGSAQQMYQSLAELGTLPPET 166 Score = 79.3
bits (194), Expect(2) = 5e-25 Identities =
35/36 (97%), Positives = 35/36 (97%), Gaps = 0/36 (0%) Frame = +1 Query
1540 KVFCGHEHTLSNLEFAQKVEPCNDHVRAKLSWAKAR 1647
KVFCGHEHTLSNLEFAQKVEPCNDHVRAKLSWAK R Sbjct
167
KVFCGHEHTLSNLEFAQKVEPCNDHVRAKLSWAKKR
202 Score = 59.3
bits (142), Expect(2) = 5e-25 Identities =
27/28 (96%), Positives = 28/28 (100%), Gaps = 0/28 (0%) Frame = +3 Query
1752
QKRDEDDVPTVPSTLGEERLYNPFLRVA
1835
+KRDEDDVPTVPSTLGEERLYNPFLRVA Sbjct
200
KKRDEDDVPTVPSTLGEERLYNPFLRVA
227 Score = 105 bits (261), Expect = 3e-21 Identities =
52/55 (94%), Positives = 53/55 (96%), Gaps = 0/55 (0%) Frame = +1 Query
2035
EEPVRKFTGKAVPADVLEALCKERARFEQAGEPRQPQARALLALQWGLLSAAPHD 2199
EEPVRKFTGKAVPADVLEAL KERARFEQAGEPRQPQARALLALQWGLLSAAP + Sbjct
228
EEPVRKFTGKAVPADVLEALYKERARFEQAGEPRQPQARALLALQWGLLSAAPQE 282 Score = 70.5
bits (171), Expect = 9e-11 Identities =
36/44 (81%), Positives = 39/44 (88%), Gaps = 0/44 (0%) Frame = +1 Query
256
MKVKVIPVLEDNYMYLVIEELTREAVAVDVAVPKRVRAGRGPQG 387
MKVKVIPVLEDNYMYLVIEE+TREAVAVDVAVPKR+
G +G Sbjct 1 MKVKVIPVLEDNYMYLVIEEITREAVAVDVAVPKRLLEIVGREG 44 Score = 52.4
bits (124), Expect = 3e-05 Identities =
28/41 (68%), Positives = 29/41 (70%), Gaps = 2/41 (4%) Frame = +3 Query
702
PFQLLEIVGREGVSLTAVLTTHHHW*APAGRGEARGRRLVP 824 P
+LLEIVGREGVSLTAVLTTHHHW RG RL P Sbjct 33
PKRLLEIVGREGVSLTAVLTTHHHW--DHARGNPELARLRP 71
Получается, что экзон из
человеческого генома включает в себя 3 экзона из генома обезьяны. {Мне странно, что такая большая
разница в конечных координатах (1911 и 2199). Ведь то маленькое пересечение 167-202
и 200- 227, а также то, что человеческий экзон заканчивается по белку на 290,
а макаки – на 282, все равно это все не объясняет разницу в конечных
координатах по ДНК….} Сравниваем с GeneScan
3).
Найдите Ваш ген в геноме человека, используя программу BLAT в Human Genome Browser. Выделите кодирующие и некодирующие
экзоны. База
Human Genome Browser содержит гены, белки, мРНК, EST и другие объекты,
картированные на геном человека (и не только человека). Браузер
позволяет просмотреть разнообразную информацию, относящуюся к заданному
фрагменту ДНК. Программа BLAT аналогично BLAST позволяет
искать последовательности в геноме с учетом возможной фрагментированности
генома. Пошел по ссылке "Blat". Поместил
последовательность ДНК в текстовое поле формы и нажмите кнопку Submit.
Получил список найденных фрагментов генома. Список
приведен ниже. Выбрал ту строку, которая имеет максимальное сходство с моей
последовательностью по SCORE и максимальную длину
выравнивания. Определил, что моя
последовательность находится на прямой цепи геномной ДНК, знак в колонке STRAND
«+», геномные
координаты моей последовательности (717256-719732 н.к.) и номер хромосомы =
16. Для
пересчета координат от геномных к координатам последовательности определим
число OFFSET: последовательность
находится на прямой цепи, значит, OFFSET=START(по хромосоме, после столбца
STRAND)–1, то есть OFFSET=717256-1=717255 BLAT
Search Results ACTIONS QUERY SCORE START END QSIZE IDENTITY CHRO STRAND START
END SPAN --------------------------------------------------------------------------------------------------- browser
details YourSeq 2477 1
2477 2477 100.0% 16
+ 717256 719732
2477 browser
details YourSeq 27 845
877 2477 78.6%
16 + 71639731
71639758 28 browser
details YourSeq 26 2307
2417 2477 48.3%
11 + 56021006
56021041 36 browser
details YourSeq 23 710
743 2477 96.0%
2 - 127156851 127156886 36 browser
details YourSeq 23 286
308 2477 100.0% 16
- 1812919 1812941
23 browser
details YourSeq 23 1438
1467 2477 90.0%
9 + 27635056
27635086 31 browser
details YourSeq 22 979
1008 2477 95.9%
1 - 199634988 199635020 33 browser
details YourSeq 22 905
926 2477 100.0% 10
+ 134290920 134290941 22 browser
details YourSeq 22 983
1007 2477 95.9%
10 + 725931
725964 34 browser
details YourSeq 21 1952
1972 2477 100.0% 3
+ 197742566 197742586 21 browser
details YourSeq 21 1666
1686 2477 100.0% 21
+ 34125066 34125086
21 browser
details YourSeq 21 1952
1972 2477 100.0% 20
+ 34516646 34516666
21 browser
details YourSeq 21
435 455 2477 100.0% 13
+ 22553911 22553931
21 browser
details YourSeq 19 1793
1814 2477 95.3%
4 + 44904372
44904396 25
Щёлкнул
по изображению мРНК или EST, появилось её подробное описание. На этой страничке
в разделе mRNA/Genomic Alignments перешел
по гиперссылке с выравниванием, а потом найдите раскрашенную
последовательность, заголовок которой начинается с "Genomic". На ней
синим выделены кодирующие участки, красным — некодирующие, но их не было
выделено, значит некодирующих участков нет. чёрным —
интроны, которые достаточно часто встречаются в Genomic chr16. Alignment of CR592523 and chr16:716937-719733 CCGGGCACGC GCACCGCCGG CGGGCCTTGT GGTCTCATCG
CGCGCAGGAT 50 CCGGGCCGGG CGGGGGCGGG GGCGGGGCGC
GTCCGCGGAG GGGGGCGGTT 100 GGCGGCTCCC GAGCCCAGCG
CCGCGCTCAG TCCGGACCCC GTGACCGGCG 150 GCCGAGGCCC CGCCTCCGTC
AGTCTGTCCT TCGGGTCCTC AGCACAGCCG 200 TGCCGCCCTT CCTAGGGTGT GGAGAGCGGG CCCCGCCCTG
AAGGGGCACC 250 GTGGGCTGGG GGGCCTGTTT
TGGAGCAGGC ACCGGTGGCC GAGCTCCGTG 300 ACCATGAAGG TCAAGGTCAT
CCCCGTGCTC GAGGACAACT ACATGTACCT 350 GGTCATCGAG GAGCTCACGC
GCGAGGCGGT GGCCGTGGAC GTGGCTGTGC 400 CCAAGAGGCT GCTGGAGATC GTGGGCCGGG AGGGGGTGTC
TCTGACCGCT 450 GTGCTGACCA CCCACCATCA CTGGGACCAC GCGCGGGGAA ACCCGGAGCT 500 GGCGCGGCTT CGTCCCGGGC
TGGCGGTGCT GGGCGCGGAC GAGCGCATCT 550 TCTCGCTGAC GCGCAGGCTG
GCGCACGGCG AGGAGCTGCG GTTCGGGGCC 600 ATCCACGTGC GTTGCCTCCT
GACGCCCGGC CACACCGCCG GCCACATGAG 650 CTACTTCCTG TGGGAGGACG
ATTGCCCGGA CCCACCCGCC CTGTTCTCGG 700 GTACCCGCAG CGCGGAGCGC
GCCCACCCCG CCTCCCGCCG GCCCCGCCCC 750 ATCTGCTCTG ACCCGCCCTC
CCCCGCCAGG CGACGCGCTG TCGGTGGCCG 800 GCTGCGGCTC GTGCCTGGAG
GGCAGCGCCC AGCAGATGTA CCAGAGCCTG 850 GCCGAGCTGG GTACCCTGCC
CCCCGAGACG AAGGTGTTCT GCGGCCACGA 900 GCACACGCTT AGCAACCTGG AGTTTGCCCA
GAAAGTGGAG CCCTGCAACG 950 ACCACGTGAG AGCCAAGCTG
TCCTGGGCTA AGGCACGGCC CCTTTCCCGC 1000 CGCGGCAAGA GGGTGGGGGG
GGAGGGAACA GGCTTCGGGG TGGGGGGGGC 1050 TCTCAGACAA GGCCTAATGG
TGACCGGGGC CTGTGGTCAC TCCAGAAGAG 1100 GGATGAGGAT GACGTGCCCA
CTGTGCCGTC GACTCTGGGC GAGGAGCGCC 1150 TCTACAACCC CTTCCTGCGG
GTGGCGTGAG TATGGCTGTT GTCCCGGGGC 1200 CTCCACCGTT ACGTGGACCC
TTAGGAAGGC ATCTGGGGAC TGCGTGTTGG 1250 GCTGAGTGAG CATCTCTGGC
TTGGGGGAGG CTGCTCATTA AGTGCCTGCC 1300 TGCCCGCCCA CCCCTCGGCG CCATGCTCCC
GCGTGGGCAG CGGGCCCTGC 1350 GCCTCACTGC ACCCCTCCCT
GCAGAGAGGA GCCGGTGCGC AAGTTCACGG 1400 GCAAGGCGGT CCCCGCCGAC
GTCCTGGAGG CGCTATGCAA GGAGCGGGCG 1450 CGCTTCGAAC AGGCGGGCGA
GCCGCGGCAG CCACAGGCGC GGGCCCTCCT 1500 TGCGCTGCAG TGGGGGCTCC
TGAGTGCAGC CCCACACGAC TGAGCCACCC 1550 AGACCCTCAC AGGGCTGGGG
CCTGCGTCCC TCCTCGTGAC CTCGGCCAGC 1600 TGGACCCACA TGAGGGCCAC
CTCTGGAACC TTCTTCGAGG CCCTGGCCAG 1650 CCATCTGCCC AGCCTCGGAG
GGTGGGCAAC CTGGTGCTTC CCGGGTGGAC 1700 ACACAGGACC ACTCAGTGGG
GCCTGTGTGG GCGCCGAGAC CTGGGTGTCT 1750 GGGAAGTGGG GCACACGGGG
CCTCCGAACT ATGAATAAAG CTTTGAAAGG 1800 CCGTTGTCAG TGTTGGCAGa aactcggaaa cgactggctc agccggggcg tcgggagggc
ttcctggagg 716886 aggtgccagc gccgggcgcg gcggggtcgg agcgtgcgcg
tggcgcccca 716936 CCGGGCACGC GCACCGCCGG
CGGGCCTTGT GGTCTCATCG CGCGCAGGAT
716986 CCGGGCCGGG CGGGGGCGGG
GGCGGGGCGC GTCCGCGGAG GGGGGCGGTT
717036 GGCGGCTCCC GAGCCCAGCG
CCGCGCTCAG TCCGGACCCC GTGACCGGCG
717086 GCCGAGGCCC CGCCTCCGTC
AGTCTGTCCT TCGGGTCCTC AGCACAGCCG
717136 TGCCGgtgag gcgggcggcg ggggaacgcg gctgtcccgg
gtcaggggtc 717186 ttgcggcggc agggcggggg gccgaggggc ggggcctggg
aggaaggcgt 717236 ggcctttggg gactggggct cggactgggg gcggagccgg
ggctggttgg 717286 ggaccggccg ggttccgctc ctgctggagc ccggtgcgtg
gaattccacg 717336 cgagtgccgg ggagttcctg gggagccggg cttctctttt
ggcccccagc 717386 gtgttgaccg agcccgcttc gcacagCCCT TCCTAGGGTG
TGGAGAGCGG 717436 GCCCCGCCCT GAAGGGGCAC
CGTGGGCTGG GGGGCCTGTT TTGGAGCAGG
717486 CACCGGTGGC CGAGCTCCGT
GACCATGAAG GTCAAGGTCA TCCCCGTGCT
717536 CGAGGACAAC TACATGTACC TGGTCATCGA
GGAGCTCACG CGCGAGGCGG 717586 TGGCCGTGGA CGTGGCTGTG
CCCAAGAGGg tgagggcagg ccgcgggccg 717636 cagggacccg gccgtgtccc ccgagagcct ccccgacccc
cctggtagga 717686 gcgagccccc acgtgctctg ctctccggaa gtcattggcg
gctggggttc 717736 cttgtttatc ttggggctcc ctgaagttac ggcacctctg
gcctccgccc 717786 tttcgctgct gcctggcggt ccctgcacgc gctgggcgca
gtcaccgccc 717836 gctgggtccc cgctccccgg cgctccccgg ggctctggcc
ggcctggggc 717886 agtgagcgcg gcggatcccg atatggaggg agtgggccac
cgggaccgtc 717936 tgtgttaccg tcactcccgt ccctttcagC TGCTGGAGAT
CGTGGGCCGG 717986 GAGGGGGTGT CTCTGACCGC
TGTGCTGACC ACCCACCATC ACTGgtgagc 718036 gccggcgggg cgcggggagg cacgaggacg ccgccttgtc
ccaacccgac 718086 ctaacccggc ccccgcccgc ccgcccgcag GGACCACGCG
CGGGGAAACC 718136 CGGAGCTGGC GCGGCTTCGT
CCCGGGCTGG CGGTGCTGGG CGCGGACGAG
718186 CGCATCTTCT CGCTGACGCG
CAGGCTGGCG CACGGCGAGG AGCTGCGGgt 718236 gagcgcgcgc tcccgggagg ggcggggagg gcgccccggg
tccacccgcc 718286 ctcacaggtc cgcctgctcc tccgccgcag TTCGGGGCCA
TCCACGTGCG 718336 TTGCCTCCTG ACGCCCGGCC
ACACCGCCGG CCACATGAGC TACTTCCTGT
718386 GGGAGGACGA TTGCCCGGAC
CCACCCGCCC TGTTCTCGGG TACCCGCAGC
718436 GCGGAGCGCG CCCACCCCGC
CTCCCGCCGG CCCCGCCCCA TCTGCTCTGA
718486 CCCGCCCTCC CCCGCCAGGC
GACGCGCTGT CGGTGGCCGG CTGCGGCTCG
718536 TGCCTGGAGG GCAGCGCCCA
GCAGATGTAC CAGAGCCTGG CCGAGCTGGG
718586 TACCCTGCCC CCCGAGACGg tgagcgggcc tgggccctcc cctcttctcc 718636 cgtgggcaca gcccccacgc tccgcaccct cactgtgcta
ggggtgcaga 718686 gtgaatgccc acctgagggc agaccgggca ggggaggcca
ggcccccggc 718736 gcaagcactt tccccgcttc ctggccgcgt gcgcgctcac cgagcgctct 718786 tcctccagAA GGTGTTCTGC
GGCCACGAGC ACACGCTTAG CAACCTGGAG
718836 TTTGCCCAGA AAGTGGAGCC
CTGCAACGAC CACGTGAGAG CCAAGCTGTC
718886 CTGGGCTAAG GCACGGCCCC
TTTCCCGCCG CGGCAAGAGG GTGGGGGGGG
718936 AGGGAACAGG CTTCGGGGTG
GGGGGGGCTC TCAGACAAGG CCTAATGGTG
718986 ACCGGGGCCT GTGGTCACTC
CAGAAGAGGG ATGAGGATGA CGTGCCCACT
719036 GTGCCGTCGA CTCTGGGCGA
GGAGCGCCTC TACAACCCCT TCCTGCGGGT
719086 GGCGTGAGTA TGGCTGTTGT
CCCGGGGCCT CCACCGTTAC GTGGACCCTT
719136 AGGAAGGCAT CTGGGGACTG
CGTGTTGGGC TGAGTGAGCA TCTCTGGCTT
719186 GGGGGAGGCT GCTCATTAAG
TGCCTGCCTG CCCGCCCACC CCTCGGCGCC
719236 ATGCTCCCGC GTGGGCAGCG
GGCCCTGCGC CTCACTGCAC CCCTCCCTGC
719286 AGAGAGGAGC CGGTGCGCAA
GTTCACGGGC AAGGCGGTCC CCGCCGACGT
719336 CCTGGAGGCG CTATGCAAGG
AGCGGGCGCG CTTCGAACAG GCGGGCGAGC 719386 CGCGGCAGCC ACAGGCGCGG
GCCCTCCTTG CGCTGCAGTG GGGGCTCCTG
719436 AGTGCAGCCC CACACGACTG
AGCCACCCAG ACCCTCACAG GGCTGGGGCC
719486 TGCGTCCCTC CTCGTGACCT
CGGCCAGCTG GACCCACATG AGGGCCACCT
719536 CTGGAACCTT CTTCGAGGCC
CTGGCCAGCC ATCTGCCCAG CCTCGGAGGG 719586 TGGGCAACCT GGTGCTTCCC
GGGTGGACAC ACAGGACCAC TCAGTGGGGC
719636 CTGTGTGGGC GCCGAGACCT
GGGTGTCTGG GAAGTGGGGC ACACGGGGCC
719686 TCCGAACTAT GAATAAAGCT
TTGAAAGGCC GTTGTCAGTG TTGGCAGatg 719736 tgccaggaga ggagctgttt tcgtaggcgt gttttaggag
gggtgcgttt 719786 attagacaaa
cgctgggaga caggcctggt ggggacctgg ctggggg
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
На главную страницу
третьего семестра
© Кузеванов Алексей,2005