Мембранные
белки.
|
1. Построение парного выравнивания исследуемого белка и заданного прототипа В БД Uniprot по AC P60844 была найдена последовательность
аквапорина (AQPZ_ECOLI). В БД PDB была найдена последовательность этого же
белка (PDB 1RC2). В
БД PDB были найдены две цепи 1RC2:A и 1RC2:B. Я построил выравнивание
этих цепей и увидел, что они одинаковы. Затем построил выравнивание
AQPZ_ECOLI и 1RC2:A. Получившееся
выравнивание имеет сходство 100%. Выравнивание приведено внизу страницы. В БД Uniprot получил последовательность изучаемого белка -
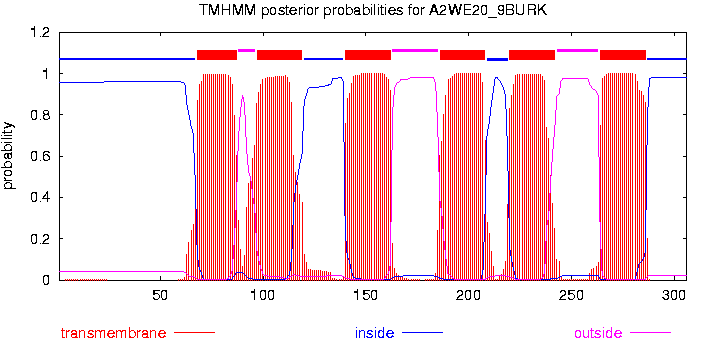
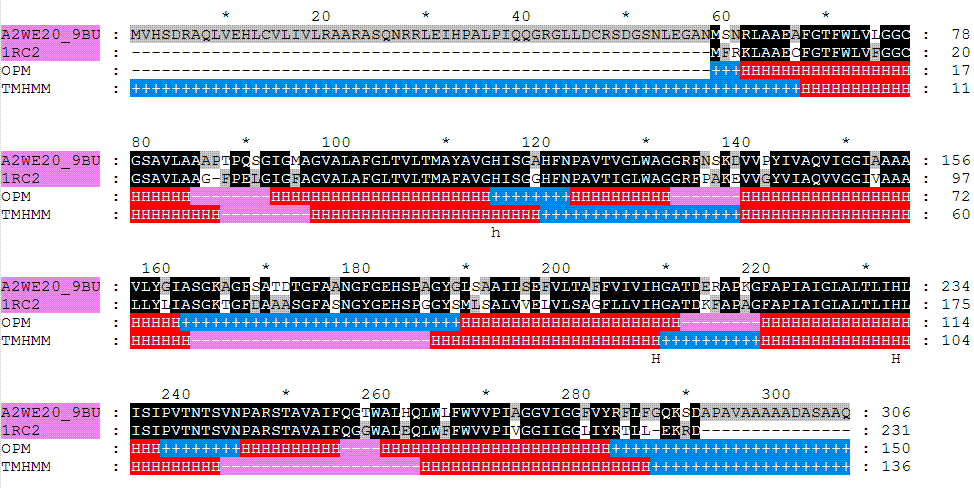
A2WE20_9BURK – Глицериновый канал. 2. Разметка мембранных сегментов на выравнивании. В БД OPM (Orientations of Proteins in Membranes database) по PDB –“1RC2”-белка-прототипа нашел описание ориентации белка в мембране. В файле mrking.msf под последовательностями исследуемого белка и белка-прототипа добавил последовательность с названием "OPM". Отметил позиции мембранных сегментов буквой "Н", позиции цитоплазматических петель знаком "+", остальные — знаком "-". 3. Предсказание топологии заданного белка с помощью наиболее популярной программы (TMHMM) Чтобы предсказать топологию, на вход программе была подана последовательность моего исследуемого белка в fasta-формате. Программа выдала следующий вариант локализации белка в мембране:
К последовательностям в файле mrking.msf добавил еще одну последовательность "TMHMM", которая отражает результаты данного предсказания.
В самом конце страницы приведен файл mrking.aln 4. Оценка качества предсказания Результаты предсказания топологии мембранного белка аквапорина
(AQPZ_ECOLI)
Результаты
анализа сравнения показывают, что программа TMHMM неплохо справилась с
поставленной задачей: значения для чувствительности и специфичности превышают
75.8% и 85,8% соответственно. А сверх- и недопредсказание составляют порядка
16-21%. Таким образом, программа довольно четко указала границы найденных
сегментов. Выравнивание AQPZ_ECOLI и 1RC2:A: #======================================= # # Aligned_sequences: 2 # 1: 1RC2 # 2: AQPZ_ECOLI # Matrix: EBLOSUM62 # Gap_penalty: 10.0 # Extend_penalty: 0.5 # # Length: 231 # Identity:
231/231 (100.0%) # Similarity:
231/231 (100.0%) # Gaps:
0/231 ( 0.0%) # Score: 1185.0 # # #======================================= 1RC2
1 MFRKLAAECFGTFWLVFGGCGSAVLAAGFPELGIGFAGVALAFGLTVLTM 50
|||||||||||||||||||||||||||||||||||||||||||||||||| AQPZ_ECOLI
1 MFRKLAAECFGTFWLVFGGCGSAVLAAGFPELGIGFAGVALAFGLTVLTM 50 1RC2
51 AFAVGHISGGHFNPAVTIGLWAGGRFPAKEVVGYVIAQVVGGIVAAALLY 100
|||||||||||||||||||||||||||||||||||||||||||||||||| AQPZ_ECOLI
51 AFAVGHISGGHFNPAVTIGLWAGGRFPAKEVVGYVIAQVVGGIVAAALLY 100 1RC2
101 LIASGKTGFDAAASGFASNGYGEHSPGGYSMLSALVVELVLSAGFLLVIH 150
|||||||||||||||||||||||||||||||||||||||||||||||||| AQPZ_ECOLI
101 LIASGKTGFDAAASGFASNGYGEHSPGGYSMLSALVVELVLSAGFLLVIH 150 1RC2
151 GATDKFAPAGFAPIAIGLALTLIHLISIPVTNTSVNPARSTAVAIFQGGW 200
|||||||||||||||||||||||||||||||||||||||||||||||||| AQPZ_ECOLI
151 GATDKFAPAGFAPIAIGLALTLIHLISIPVTNTSVNPARSTAVAIFQGGW 200 1RC2
201 ALEQLWFFWVVPIVGGIIGGLIYRTLLEKRD
231
||||||||||||||||||||||||||||||| AQPZ_ECOLI
201 ALEQLWFFWVVPIVGGIIGGLIYRTLLEKRD
231 #--------------------------------------- #---------------------------------------
CLUSTAL
W(1.60) multiple sequence alignment A2WE20_9BURK
MVHSDRAQLVEHLCVLIVLRAARASQNRRLEIHPALPIQQGRGLLDCRSDGSNLEGANMS 1RC2
----------------------------------------------------------MF OPM
----------------------------------------------------------++ TMHMM
++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++ A2WE20_9BURK
NRLAAEAFGTFWLVLGGCGSAVLAAAPTPQSGIGMAGVALAFGLTVLTMAYAVGHISGAH 1RC2 RKLAAECFGTFWLVFGGCGSAVLAAG-FPELGIGFAGVALAFGLTVLTMAFAVGHISGGH OPM
+HHHHHHHHHHHHHHHHHHHHHHH--------HHHHHHHHHHHHHHHHHHHHHH++++++ TMHMM
+++++++HHHHHHHHHHHHHHHHHHHH---------HHHHHHHHHHHHHHHHHHHHHHH+ A2WE20_9BURK FNPAVTVGLWAGGRFNSKDVVPYIVAQVIGGIAAAAVLYGIASGKAGFSATDTGFAANGF 1RC2
FNPAVTIGLWAGGRFPAKEVVGYVIAQVVGGIVAAALLYLIASGKTGFDAAASGFASNGY OPM
++HHHHHHHHHH-------HHHHHHHHHHHHHHHHHHHHHH+++++++++++++++++++ TMHMM
+++++++++++++++++++HHHHHHHHHHHHHHHHHHHHHHH------------------ A2WE20_9BURK
GEHSPAGYGLSAAILSEFVLTAFFVIVIHGATDERAPKGFAPIAIGLALTLIHLISIPVT 1RC2
GEHSPGGYSMLSALVVELVLSAGFLLVIHGATDKFAPAGFAPIAIGLALTLIHLISIPVT OPM
+++++++++HHHHHHHHHHHHHHHHHHHHHH--------HHHHHHHHHHHHHHHHHH+++ TMHMM
------HHHHHHHHHHHHHHHHHHHHHHH++++++++++HHHHHHHHHHHHHHHHHHHHH A2WE20_9BURK
NTSVNPARSTAVAIFQGTWALHQLWLFWVVPIAGGVIGGFVYRFLFGQKSDAPAVAAAAA 1RC2
NTSVNPARSTAVAIFQGGWALEQLWFFWVVPIVGGIIGGLIYRTLL-EKRD--------- OPM +++++HHHHHHHHHH----HHHHHHHHHHHHHHHHHHHHHHH++++++++++++++++++ TMHMM
HHH--------------------HHHHHHHHHHHHHHHHHHHHHHH++++++++++++++ A2WE20_9BURK DASAAQ 1RC2 ------ OPM ++++++ TMHMM ++++++ |
На главную страницу
четвертого семестра
© Кузеванов Алексей,2005