1.Создание изображения дерева, описанного
заданной мне формулой.
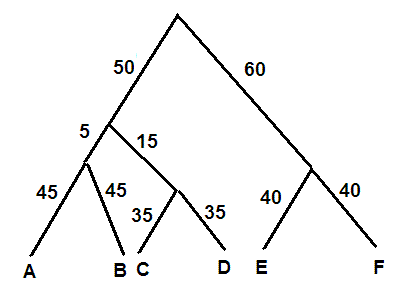
Скобочная формула моего дерева - (((А:45,В:45):5,(С:35,D:35):15):50,(Е:40,F:40):60);
Пользовался графическим редактором Paint.
2.Описание ветвей дерева как разбиений
множества листьев (считая дерево бескорневым).
A B C D E F * * . . * * * * * * . . . . * * * *
Приведено
правильное описание топологии приведённого дерева, состоящие всего из трёх
строк, отвечающих трём внутренним ветвям.
3.Получение
искусственных мутантных последовательности, соответствующих листьям и
узлам дерева, считая, что в корне находится
последовательность гена моего белка Glpk_ecoli.
Длина гена белка Glpk_ecoli
- 1593 нуклеотидов.
Формула для пересчёта расстояний в число мутаций в гене Glpk: (L/100)*1593, L - длина ветки.
Текст скрипта, которым получаются мутантные последовательности:
msbar glpk.fasta ef.fasta -point 4 -count 956 -auto msbar ef.fasta f.fasta -point 4 -count 1593 -auto msbar ef.fasta e.fasta -point 4 -count 1593 -auto msbar glpk.fasta abcd.fasta -point 4 -count 797 -auto msbar abcd.fasta ab.fasta -point 4 -count 80 -auto msbar ab.fasta a.fasta -point 4 -count 717 -auto msbar ab.fasta b.fasta -point 4 -count 717 -auto msbar abcd.fasta cd.fasta -point 4 -count 239 -auto msbar cd.fasta c.fasta -point 4 -count 558 -auto msbar cd.fasta d.fasta -point 4 -count 558 -auto cat a.fasta >> ali.fasta cat b.fasta >> ali.fasta cat c.fasta >> ali.fasta cat d.fasta >> ali.fasta cat e.fasta >> ali.fasta cat f.fasta >> ali.fasta
4.Реконструкция
дерева алгоритмами UPGMA, Neighbor-joining и максимального
правдоподобия.
Поместил последовательности, соответствующие листьям дерева, в один файл в
виде "выравнивания" в fasta-формате. Поменял имена
последовательностей (программа msbar сохраняет для мутантной
последовательности имя, которое имела исходная). Чтобы реконструировать
дерево алгоритмом максимального правдоподобия, запустил программу fdnaml:
fdnaml ali.fasta -ttratio 1 -auto
Чтобы
реконструировать дерево алгоритмами UPGMA или Neighbor-joining, сначала
посчитал попарные расстояния между последовательностями программой fdnadist:
fdnadist ali.fasta -ttratio 1 -auto
После
этого файл с расширением .fdnadist подал на вход программе fneighbor для
реконструкции алгоритмом Neighbor-joining:
fneighbor ali.fdnadist -auto
Переименовал
выходной файл первого запуска и запустил программу еще раз для реконструкции
алгоритмом UPGMA:
fneighbor
ali.fdnadist -treetype u -auto
Дерево,
реконструированное алгоритмом максимального правдоподобия:
+-------------B
|
|
+-------------F
| +---------------4
| | +-----------E
1--3
| | +----------D
| +-----2
| +----------C
|
+------------A
Дерево,
реконструированное алгоритмом Neighbor-joining:
+-------------B
!
! +----------C
!
+-----2
!
! +----------D
3-4
!
! +------------E
!
+-----------------1
!
+------------F
!
+------------A
Дерево,
реконструированное алгоритмом UPGMA:
+-------------------------A
+-----3
! +-------------------------B
+-------------4
! ! +--------------------C
! +----------1
--5
+--------------------D
!
!
+------------------------E
+--------------------2
+------------------------F
Для
сравнение предложено было сделать таблицу, в левой части которой приведены (в
виде точек и звёздочек) все ветви, встреченные во всех деревьях (исходном и
трёх реконструкциях), а в правой добавлены четыре столбца, соостветствующие
четырём деревьям. Знаком + отмечено, в каких деревьях встретилась каждая из
ветвей.
|
A
|
B
|
C
|
D
|
E
|
F
|
правильное дерево
|
1ое
|
2ое
|
3е
|
|
*
|
*
|
.
|
.
|
*
|
*
|
+
|
+
|
+
|
+
|
|
*
|
*
|
*
|
*
|
.
|
.
|
+
|
+
|
+
|
+
|
|
.
|
.
|
*
|
*
|
*
|
*
|
+
|
+
|
+
|
+
|
Дерево,
реконструированное алгоритмом максимального правдоподобия, оказалось таким же
как и дерево, реконструированное алгоритмом Neighbor-joining. Дерево,
реконструированное алгоритмом UPGMA, оказалось одинаковым с исходным деревом.
|