.png)
.png)
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных последовательностях. C помощью нее была совершена попытка найти возможные комплементарные участки в последовательности исследуемой тРНК. Наиболее адекватные результаты получились при параметрах 1)gap penalty 10 2)minimum score threshold 30 3)match score 5 4)mismatch -4. Однако они очень сильно отличались от реальных, полученных с помощью find_pair. Из всех стеблей программа обнаружила только акцепторный, и то со сдвигом на одну пару.
Еще одна программа для предсказания: RNAfold из пакета Viena Rna Package реализует алгоритм Зукера. Т.к. несмотря на то, был указан путь к ней (export PATH=${RNAfold}:/home/preps/golovin/progs/bin), программа не работала, был использован web вариант.
В результате работы этой программы были получены minimum free energy (MFE) structure, картинка с изображение этой потенциальной структуры и график возможных связей между основаниями (см. ниже).
.png) | .png) |
| Таблица №1 | |||
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера (RNAfold) |
| Акцепторный стебель | 5'-902-907-3' 5'-966-971-3' Всего 6 пар | 5'-901-907-3' 5'-965-971-3' Всего 7 пар (со сдвигом на 1 нуклеоитид) | 5'-901-907-3' 5'-966-972-3' Всего 7 пар (1 лишняя) |
| D-стебель | 5'-910-912-3' 5'-923-925-3' Всего 3 пары | 0 пар | 5'-910-912-3' 5'-923-925-3' Всего 3 пары (совпадают) |
| T-стебель | 5'-949-953-3' 5'-961-965-3' Всего 5 пар | 0 пар | 5'-949-953-3' 5'-961-965-3' Всего 5 пар (совпадают) |
| Антикодоновый стебель | 5'-937-944-3' 5'-926-933-3' Всего 8 пар | 0 пар | 5'-940-944-3' 5'-926-930-3' Всего 5 пар (3 пары пропущены) |
| Общее число канонических пар нуклеотидов | 22 пары | 7 пар | 20 пар |
Ниже представлен апплет Jmol, в котором с помощью скрипта_1 задаются и показываются атомы ДНК из следующих множеств: set1 - множество атомов кислорода 2'-дезоксирибозы; set2 - множество атомов кислорода в остатке фосфорной кислоты; set3 - множество атомов азота в азотистых основаниях.
Далее скрипт_2 были определены полярные и неполярные контакты между заданными ДНК и белком. При том считалось, что полярные - атомы кислорода и азота, неполярные - атомы углерода, фосфора и серы, а полярный контакт - полярный атом белка и полярный атом ДНК на расстоянии меньше 3.5Å, аналогично, неполярным контакт - пара неполярных атомов на расстоянии меньше 4.5Å.
В результате были получены данные, представленные в таблице №2. Интересно заметить, что полярные контакты найдены только для фосфатных остатков, тогда как неполярные встречаются со всеми группами атомов. При этом, что естественно было ожидать, в основном контакты образованы со внешней стороной ДНК, тогда как контактов со азотистыми основаниями, обращеннем внутрь двойной спирали, весьма мало.
| Таблица №2 | |||
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 0 | 90 | 90 |
| остатками фосфорной кислоты | 67 | 28 | 95 |
| остатками азотистых оснований со стороны большой бороздки | 0 | 29 | 29 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 2 | 2 |
C помощью программы nucplot была получена схема ДНК-белковых контактов, которая затем была переведена в формат изображений и представлена ниже.
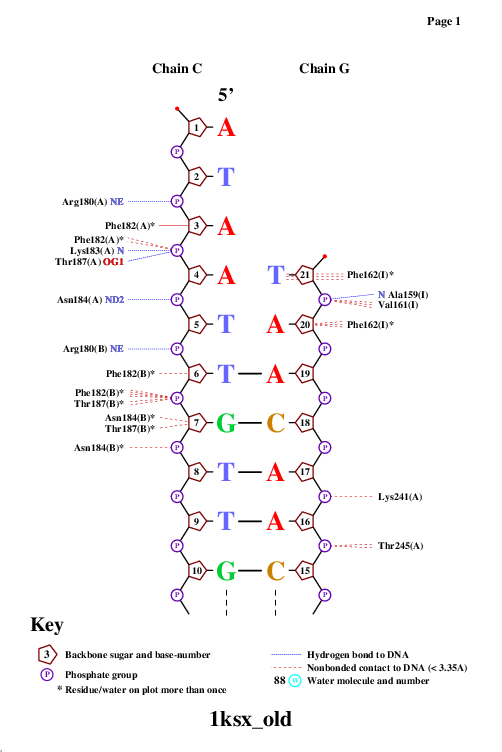 | 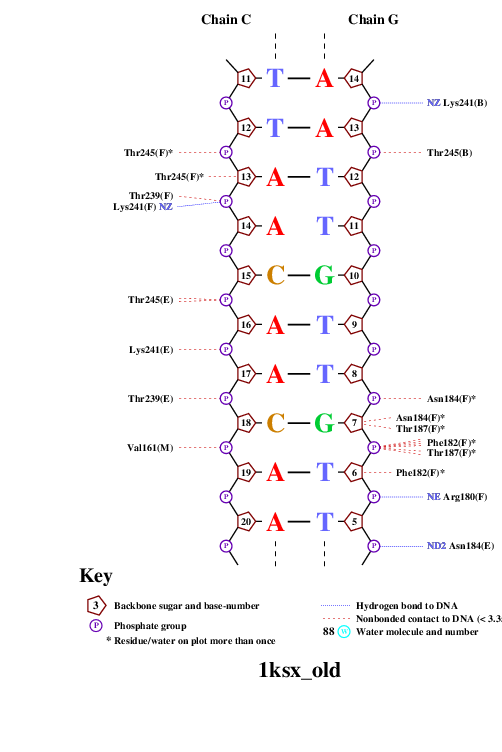 | 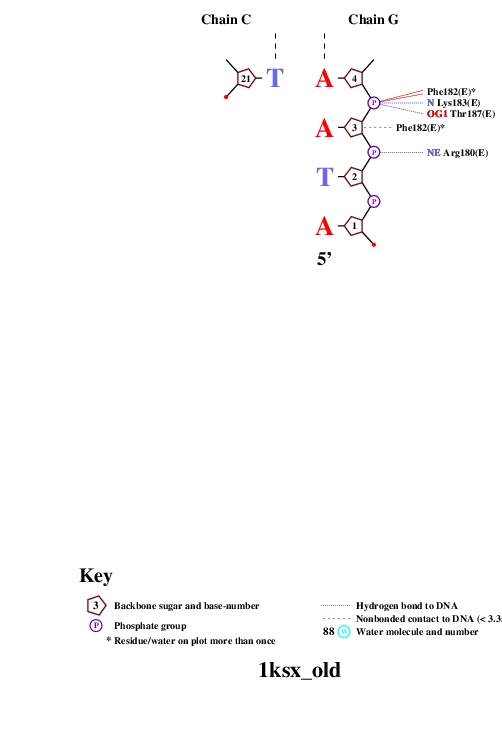 |
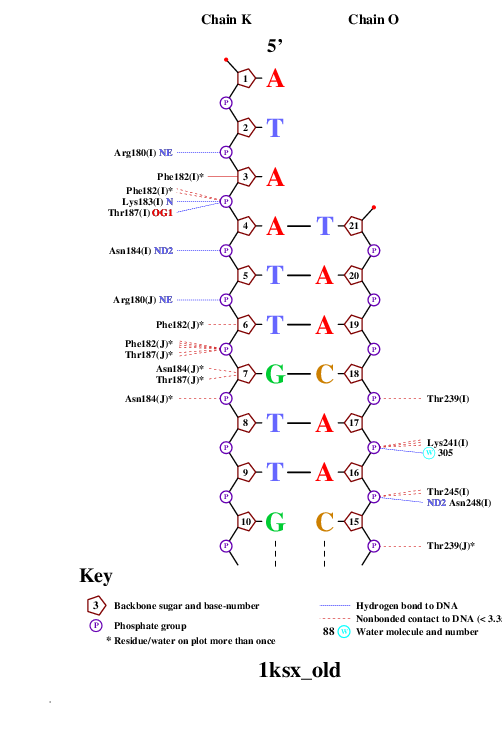 | 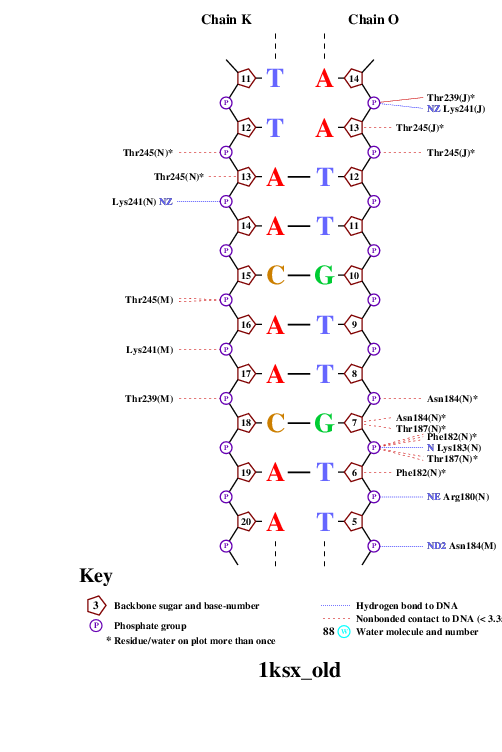 | 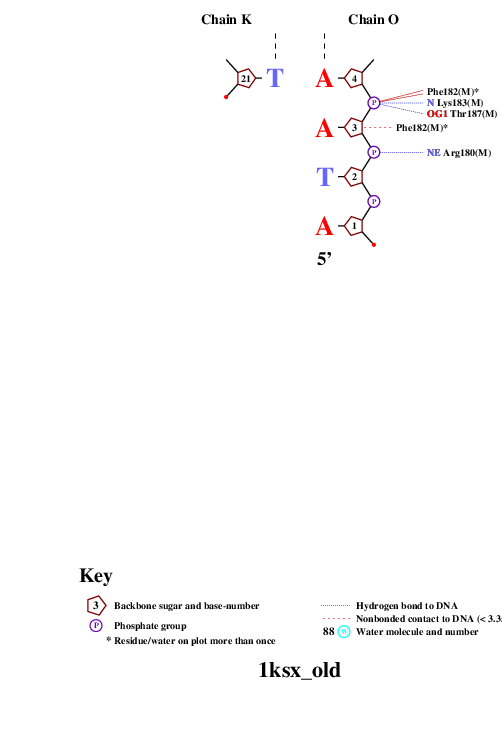 |
Исходя из данной схемы контактов можно предположить, что аминокислотный остаток, наиболее важный для распознавания последовательности ДНК, это Phe162(I), потому что он образует 2 контакта непосредственно с азотистыми основаниями (А и Т) с растоянием в районе 3Å, (как длина водородной связи). Очень много остатоков, образующих по два контакта с ДНК, при том часто встречаются одинаковые остатки, принадлежащие разным цепям, но ни один остаток не образует единолично более двух связей.
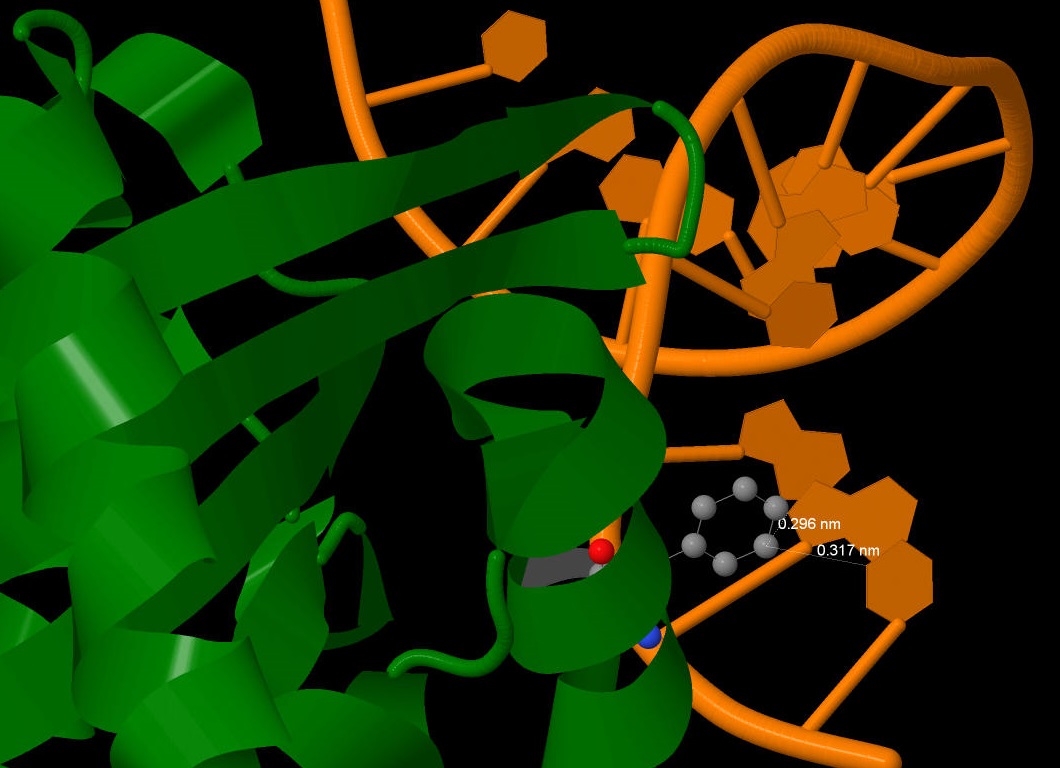 | 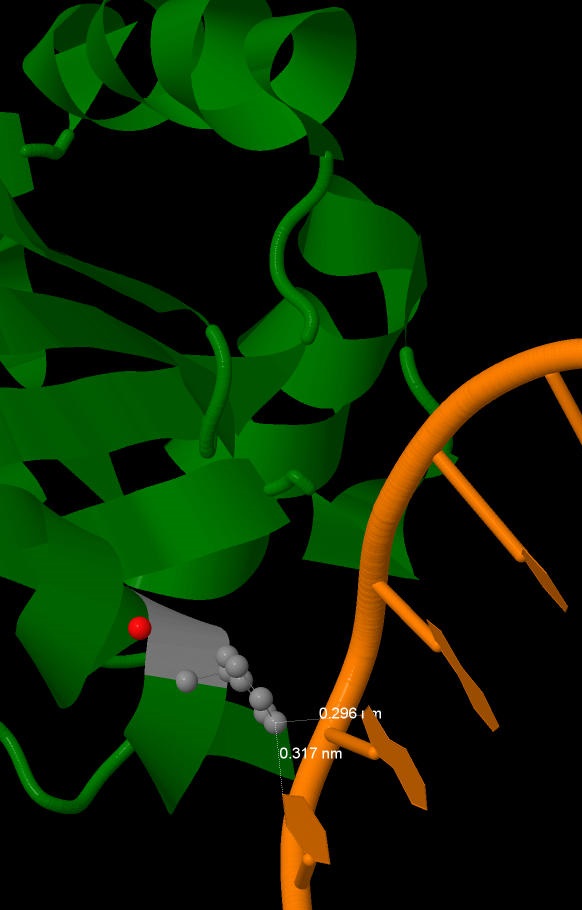 |
Изображение, показывающее контакт Phe162(I) с ДНК (G).