В данном практикуме с помощью программы Chromas (Lite) анализировались две последовательности (прямой и обратной цепочки) ДНК, выданные капиллярным секвенатором по Сангеру. Исходные файлы F.ab1, R.ab1 прямая и обратная цепь соответственно. Обратная цепь ДНК была перевернута и переведена в комплементарную (reverse+comlement).
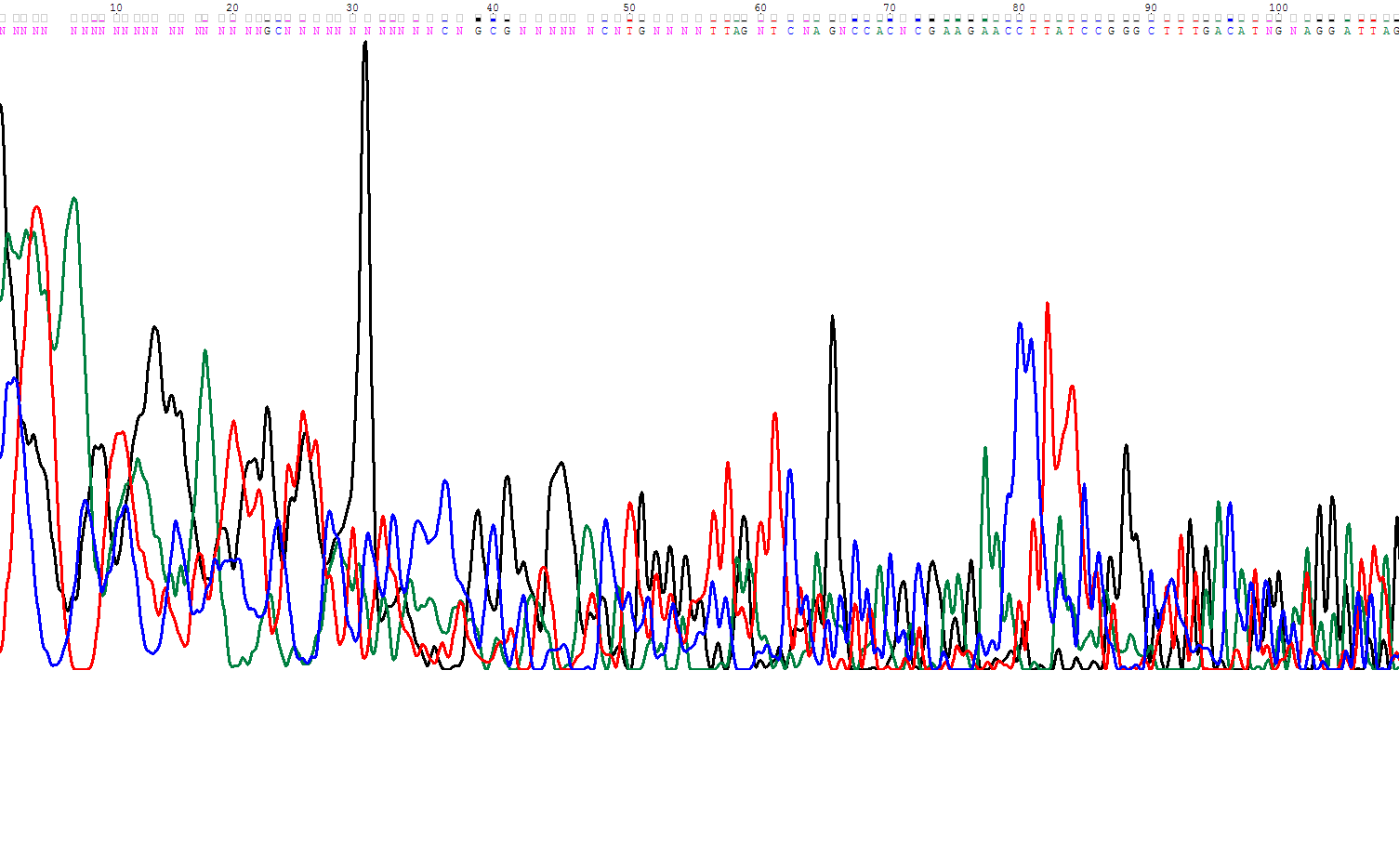
На прямой цепочке длина начального нечитаемого участка (5' конец) составляет 51 нуклеотид, тогда как в обратной цепочке 138 нуклеотидов (для прямой последовательности по 87 нуклеотид). А конечный нечитаемый участок (3' конец) в прямой цепи начинается с 773 нуклеотида (что соответвует 779 нуклеотиду на обратной цепи) и по конец хромотограммы 936 нуклеотид, а для обратной цепи нечитаемый участок с 905 по 950 нуклеотид (картированию по прямой последовательности не поддается).
Хроматограмма прямой цепи
Хроматограмма обратной цепи
После были удалены нечитаемые концы обеих цепей (Options -- Continuous edit; Edit -- Delete Trimmed Sequences). Нумерация нуклеотидов изменилась. Теперь первый нуклеотид обратной последовательности соответвует восемьдесят третьему в прямой.
В результате проверки и исправления данных последовательностей ДНК (в основном основанием для исправлений служил соответствующий участок на другой цепи) были получены окончательные последовательности в формате .fasta Прямая цепь, Обратная цепь
Ниже представлено выравнивание итоговых последовательностей в JalView

Ссылка на проект выравнивания в Jalview
Далее представлены примеры исправлений для четырех проблемных нуклеотидов.
Первый нуклеотид в прямой цепи с №96.
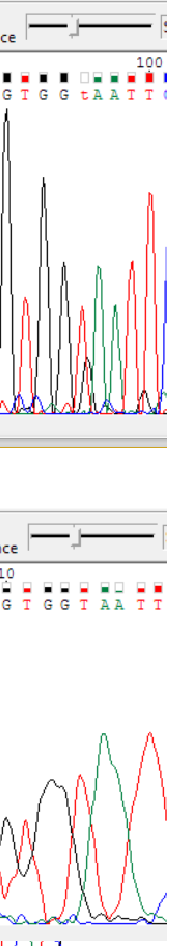
Вопрос возникает в связи с частичным перекрыванием красного пика тимина и черного пика гуанина, который выше уровня шума, но ниже уровня сигнала. Однако, пик гуанина раза в два ниже уровня сигнала и пика тимина, который соответствует среднему уровню сигнала, и он именно втиснулся между пиками тимина и аденина, расстояние между ними и гуанином не соответствует основному среднему расстоянию между пиками. И окончательным фактором, решающим, что пик гуанина не имеет никакого отношения к реальной последовательности, явилось его отсутвие в обратной цепи.
Второй нуклеотид в обратной цепи с №25 (по прямой цепи №117).
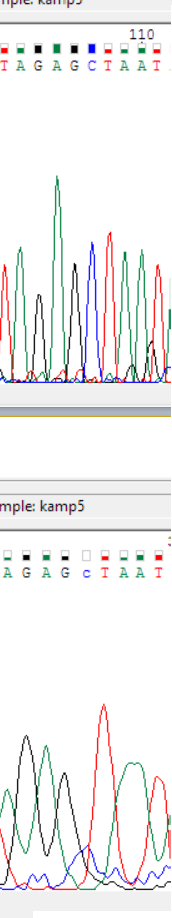
В данном случае между пиками гаунина и тимина видно синее поднятие сигнала цитозина не сильно выше шума, но, однако, на соответствующем расстоянии от соседних пиков, которые будут находится на слишком большом расстоянии, если его не учесть. И если обратиться к прямой последовательности, то на соответствующем месте виден несомненный пик цитозина. Так что, решение было принято в его пользу.
Третий нуклеотид в прямой цепи с №400
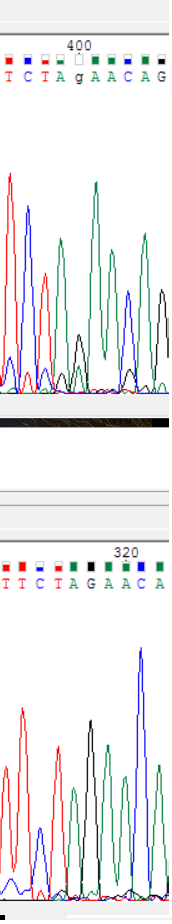
Здесь пик гуанина сильно ниже уровня сигнала, всего раза в два выше пика аденина, находящегося ровно под ним, который по силе скорее соответствует шуму. В принципе можно было бы заподозрить полиморфизм, т.к. в этом случае суммарный сигнал больше бы соответствовал общему уроню. Но на соответствующем месте в обратной цепи виден весьма сильный пик гуанина и полное отсутствие аденина. В связи с чем был выбран гуанин.
Четвертый нуклеотид в обратной цепи с №107 (по прямой цепи №189).
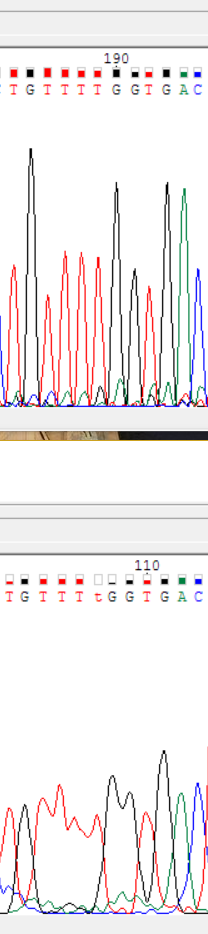
Последний случай связан с тем, что вместо отдельных пиков, четыре пика тимина обьединились в один, и последний пик для программы оказался под вопросом. Но на прямой цепи видны четыре четко различимых пика.
В результате редактирования последовательности остался один не определенный нуклеотид в обратной цепи ДНК №729 (соответствующего участка на прямой цепи нет).
Ниже представлен фрагмент из плохой по качеству хромотограммы Последовательность не была определена программой и действительно является не читаемой, потому что сила сигнала и шума одинакова, их не различить, пики не четкие: они размазанные (широкие), находят друг на друга, расстояние между вершинами пиков постоянно меняется, нельзя сказать что сила сигнала равномерна на каких-либо значительных участках хромотограммы.