
Требовалось определить с помощью BLAST таксономию и функцию нуклеотидной последовательности, прочтенной в практикуме №6. Для начала стоит обратить внимание, что из практикума №6 мы имеем две последовательности: с прямой цепи и с обратной цепи. Было решено объединить в единную последовательность, для того, чтобы не терять значительную и довольно не плохо прочитанную часть последовательности. После, при анализе полученных находок, можно будет учесть, что концы менее достоверны, чем средняя часть.
Данная последовательность была брошена в нуклеотидный BLAST; по базе данных Nucleotide collection (nr/nt); с длиной слова 16. Выдача blast (лучшие находки) представлена ниже.
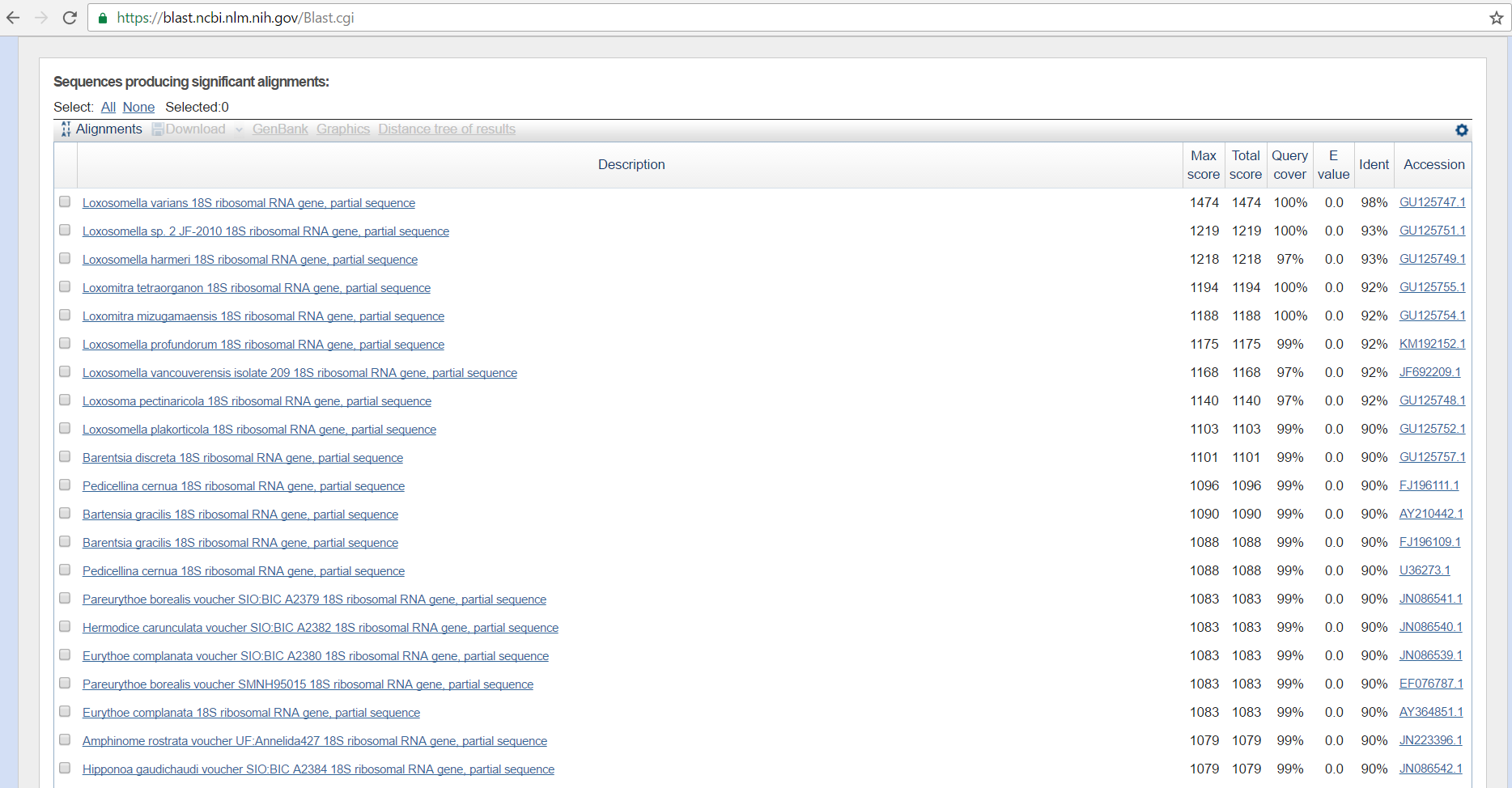
Судя по всему, данная последовательность не является белоккодирующей, а последовательностью 18S рРНК малой (40S) субъединицы рибосомы эукариот. Последовательность гена рРНК явялется одной из наиболее консервативных (что и видно по выдаче blast, в которой разнообразные организмы имеют довольно высокое сходство).
Чтобы определить таксономию данной последовательности было построено выравнивание ее и 9 лучших находок (с identity от 98% до 92%) из семейства Loxosomatidae (рис.1), и из них 5 лучших находок, которые относятся к роду Loxosomella (рис.2).
Рисунок 1

Рисунок 2
.png)
Видно, что исходная последовательность (первая в выравнивании) различается примерно так же, как и остальные последовательности разных видов внутри этого рода (при том некоторые виды даже больше отличаются). Добавление последовательностей из других родов еще ухудшает ситуацию, но не сильно. Значит можно утверждать, что данная последовательность относится к роду Loxosomella.
Полная таксономия: Eukaryota; Metazoa; Lophotrochozoa; Entoprocta; Loxosomatidae; Loxosomella
Сравнивались (для последовательности из первого задания) три алгоритма работы blast: 1) megablast (Optimize for Highly similar sequences) 2)discontiguous megablast (More dissimilar sequences) 3)blastn (Somewhat similar sequences). Все параметры кроме длины слова и количества находок были дефолтными.
Таблица 1. Параметры запуска.
| Algoritm | Database | Organism | Max target sequences | Expect threshold | Word size | Max matches in a query range | Match/Mismatch Scores | Gap Costs |
| megablast | Nucleotide collection (nr/nt) | Entoprocta (taxid:43120) | 1000 | 10 | 16 | 0 | 1,-2 | Linear |
| discontiguous megablast | Nucleotide collection (nr/nt) | Entoprocta (taxid:43120) | 1000 | 10 | 11 | 0 | 2,-3 | Existence:5 Extention: 2 |
| blastn | Nucleotide collection (nr/nt) | Entoprocta (taxid:43120) | 1000 | 10 | 7 | 0 | 2,-3 | Existence:5 Extention: 2 |
В результате были получены 3 выдачи. Их сравнение представлено в таблице 2.
Таблица 2.
| Алгоритм | Количество находок | Находка 1 | Находка 2 | Количество находок только при данном алгоритме | Пример | Количество находок других алгоритмов, не найденных данным | Конмментарии |
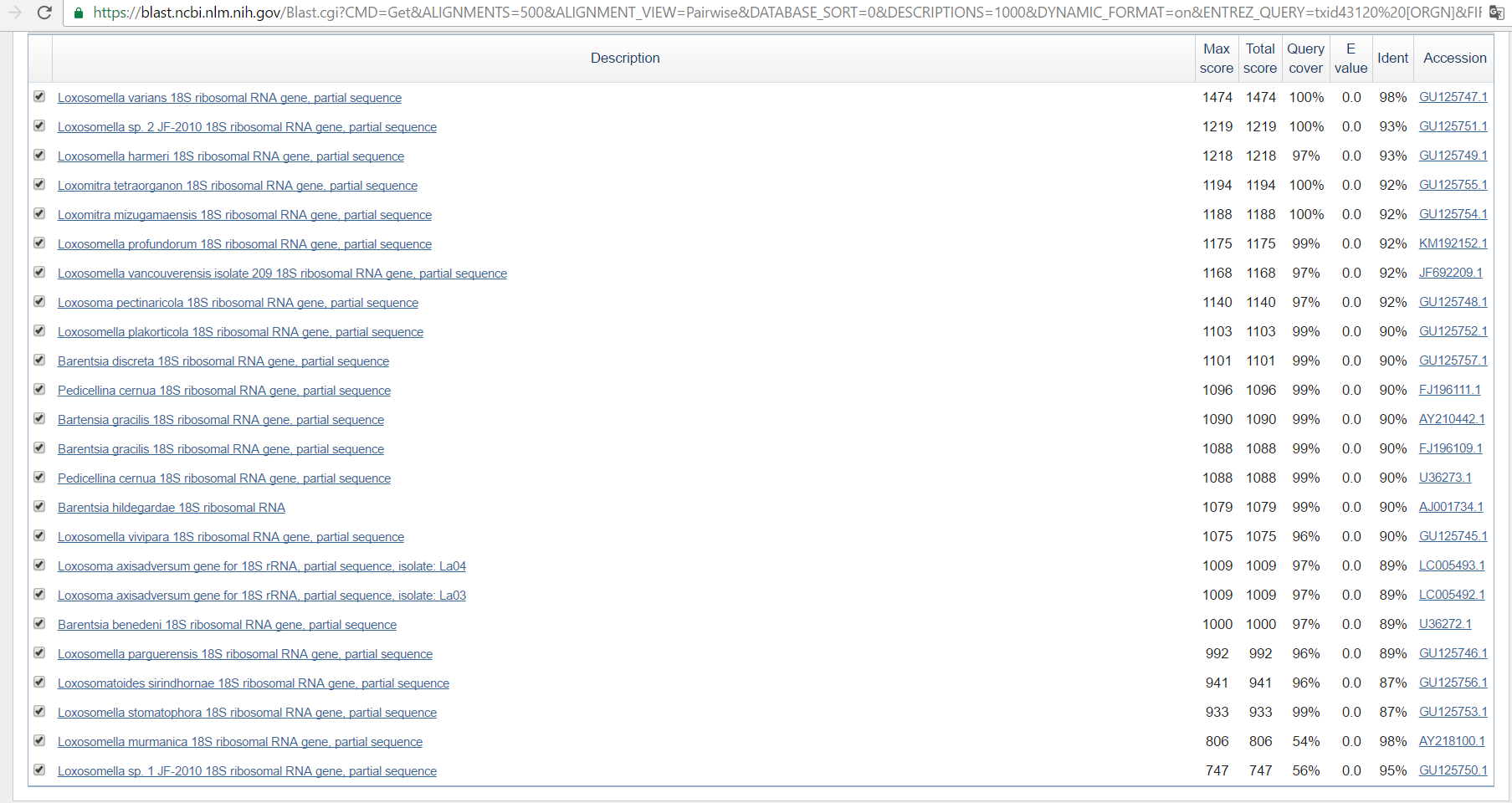
| megablast | 24 | Loxosomella varians 1474(Max score) 1474(Total score) 100%(Query cover) 0.0(E-value) 98%(Ident) | Loxosomella sp. 2 JF-2010 1219(Max score) 1219(Total score) 100%(Query cover) 0.0(E-value) 93%(Ident) | 0 | - | 7 | Набор находок совпадает с алгоритмом discontiguous megablast |
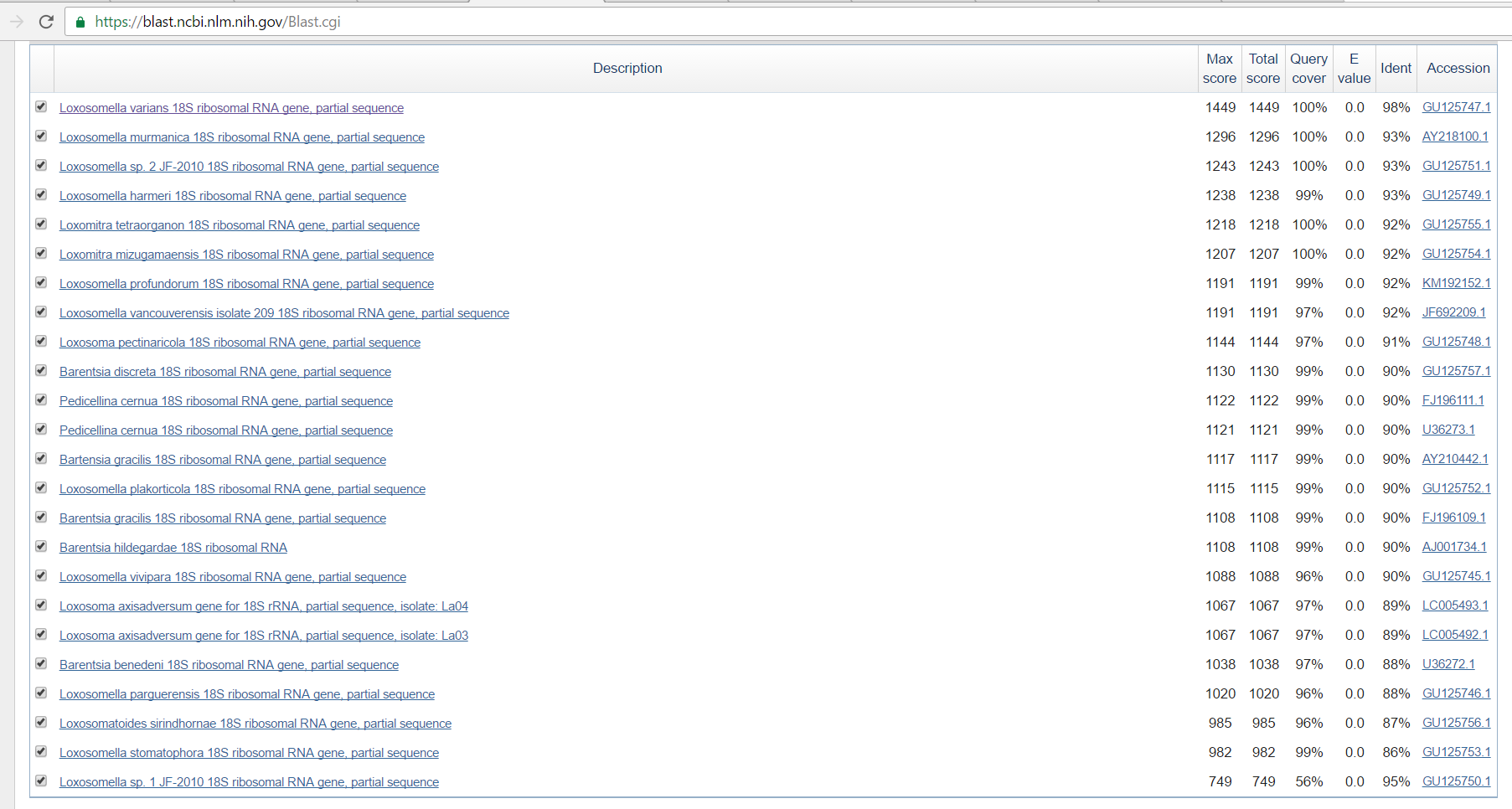
| discontiguous megablast | 24 | Loxosomella varians 1474(Max score) 1474(Total score) 100%(Query cover) 0.0(E-value) 98%(Ident) | Loxosomella murmanica 1296(Max score) 1296(Total score) 100%(Query cover) 0.0(E-value) 93%(Ident) | 0 | - | 7 | Однако очень отличается посчтанный Score: к примеру, находка 2 в алгоритме megablast находится на предпоследнем месте |
| blastn | 31 | Loxosomella varians 1474(Max score) 1474(Total score) 100%(Query cover) 0.0(E-value) 98%(Ident) | Loxosomella murmanica 1296(Max score) 1296(Total score) 100%(Query cover) 0.0(E-value) 93%(Ident) | 7 | Loxosomella murmanica cytochrome c oxidase subunit I gene, partial cds; mitochondrial 28.3(Max score) 28.3(Total score) 5%(Query cover) 0.32(E-value) 75%(Ident) | 0 | Эти дополнительные 7 находок находятся в самом конце и с абсолютно неправдоподобными E-value |
Выдача megablast
Выдача discontiguous megablast
Выдача blastn
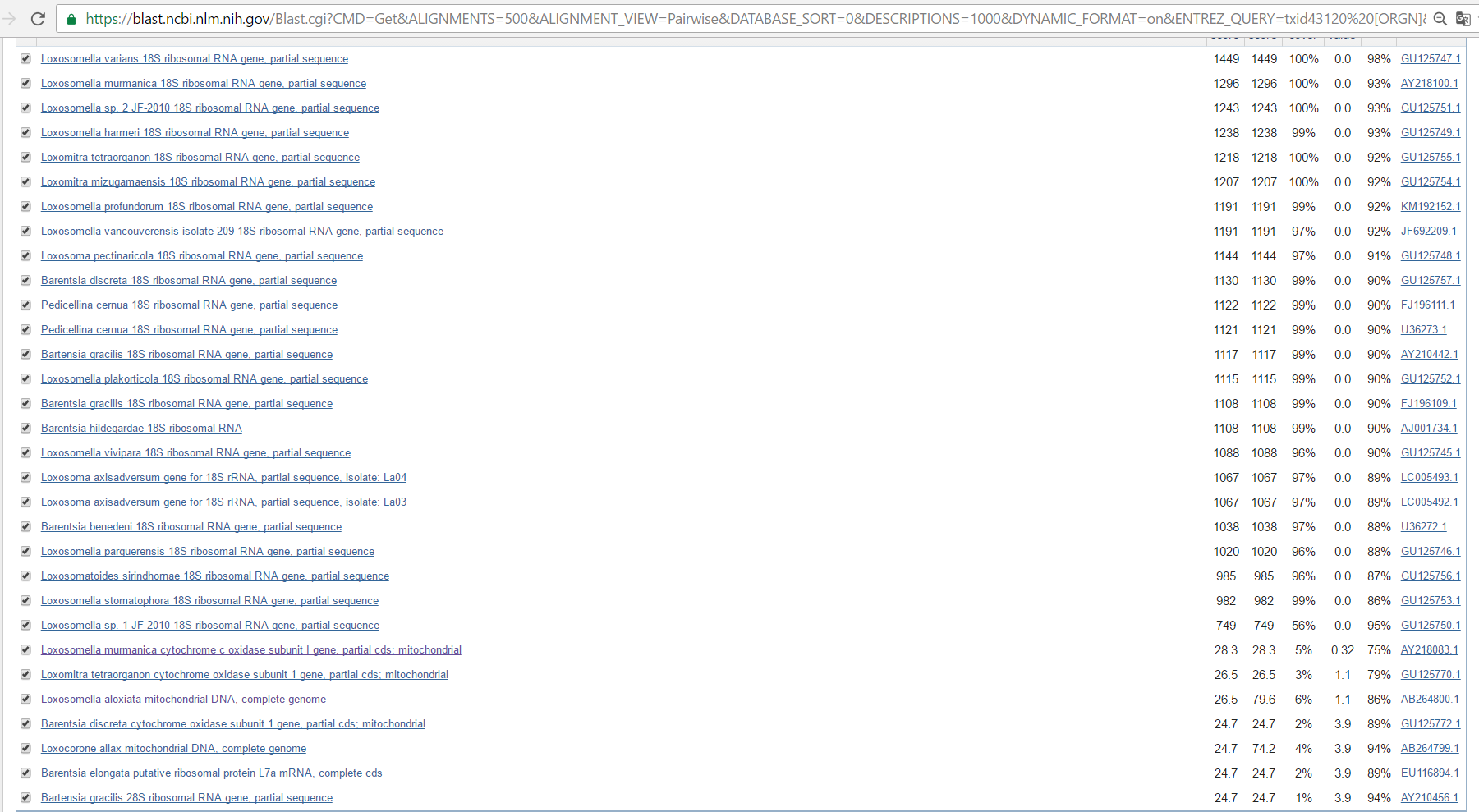
Сравнение этих алгоритмов демострирует, что blastn выдает большее количество находок чем остальные, однако требует от них как и сильно меньшей схожести (minIdent 24.7 против ~750), так и сильно меньшего качества (minE-value 3.9 против 0.0). А алгоритмы megablast и discontiguous megablast не столь сильно различаются между собой.
Надо было проверить наличие гомологов трех белков (были выбраны HSP7C_HUMAN, CISY_HUMAN, PABP2_HUMAN) в геноме Amoboaphelidium protococarum.
В начале была создан банк данных по геному Amoboaphelidium с помощью команды makeblastdb -in X5.fasta -dbtype nucl. Далее для проверки наличия гомологов надо было запустить локальный бласт нуклеотидной последовательности против нуклеотдного банка данных, но проверять на сходство белковые последовательности. Это делает алгоритм tblastn. Команда: tblastn -query input.fasta -db X5.fasta > output.out
HSP7C_HUMAN
Консервативный шаперон HSP70, белок теплового шока (белок стресса), с молекулярной массой 70кДа. Белки теплового шока действуют как внутриклеточные шапероны в отношении других белков: принимают участие в сворачивании и разворачивании белков, обеспечивают клетке нечувствительность к нагреванию. Предотвращает сворачивание белков в ходе посттрансляционного транспорта в митохондрии и хлоропласты. Компонент комплекса PRP19-CDC5, формирующего неотъемлемую часть сплайсингсомы и обязательного для активации pre-mRNA сплайсинга. Играет роль в сердечно-сосудистой системе. Участвует в связывании и презентации антигенов. Связывает бактериальные липополисахариды (LPS), медиирующие LPS-индуцированный воспалительный ответ, включающий выработку TNF моноцитами.
Результаты работы локального бласт представлены в файле HSP7C_HUMAN.OUT
Было получено 16 находок, все с разумным E-value (< 0,001) и первые со значительным Score.
Параметры лучшей находки:
scaffold-199
Length=1112851
Score = 917 bits (2369), Expect = 0.0, Identities = 474/607 (78%), Positives = 538/607 (89%), Gaps = 0/607 (0%) Frame = -2
На основании этого можно предположить, что данный участок гомологичен hps70 и выполняет подобную функцию.
CISY_HUMAN
Митохондриальная цитратсинтаза - фермент, катализирующий реакцию конденсации ацетата (ацетил-CoA) и оксалоацетата, в результате чего образуется цитрат. Катализируемая реакция является лимитирующей на первом этапе цикла трикарбоновых кислот. Синтетаза цитрата обнаружена практически во всех клетках аэробных организмов.
Результаты работы локального бласт представлены в файле CISY_HUMAN.OUT
Было получено 16 находок, все с разумным E-value (< 0,001) и первые два имеют не такой маленький Score, но все-таки значительно меньше, чем для hps70.
Параметры лучшей находки:
scaffold-693
Length=1268102
Score = 565 bits (1457), Expect = 2e-180, Identities = 262/377 (69%), Positives = 315/377 (84%), Gaps = 3/377 (1%) Frame = +1
На основании этого можно предположить условно положительный ответ: данный участок является гомологом, но функции могут быть видоизменены, т.к. сходство значительно ниже, чем в первом случае. При том нельзя утверждать наличие каких-то гомологичных доменов, потому что несоответствия распределены равномерно вдоль всей последовательности.
PABP2_HUMAN
Белок, связывающий поли(А) хвост (200-250нк) матричной РНК. Стимулирует поли(А)полимеразу. Может защищать поли(А) хвост от разрушения.
Результаты работы локального бласт представлены в файле PABP2_HUMAN.OUT
Было получено 16 находок, из который 6 с с разумным E-value (< 0,001), но даже у первых находок Score весьма низкий.
Параметры лучшей находки:
scaffold-100
Length=762135
Score = 117 bits (292), Expect = 2e-28, Identities = 54/86 (63%), Positives = 64/86 (74%), Gaps = 0/86 (0%) Frame = +1
На основании этого можно дать отрицательный ответ, потому что хоть E-value и достаточный, но вес выравнивания слишком мал, чтобы утверждать гомологию.
Надо найти один ген белка, закодированный в одном скэффолде ''Amoboaphelidium''
Чтобы выбрать скеффолд подходящей длины была использована команда infpseq из пакета (EMBOSS) infoseq X5.fasta -only -name -length. В результате был выбран контиг unplaced-1071 с длиной 86429. Он довольно длинный, и в нем вполне может уместиться ген.
Для извлечения этого контига была использована команда: seqret X5.fasta: unplaced-1071 -out unplaced1071.fasta. Полученный файл
Результаты выдачи blastn c дефолтными параметрами по таксону fungi (taxid:4751)
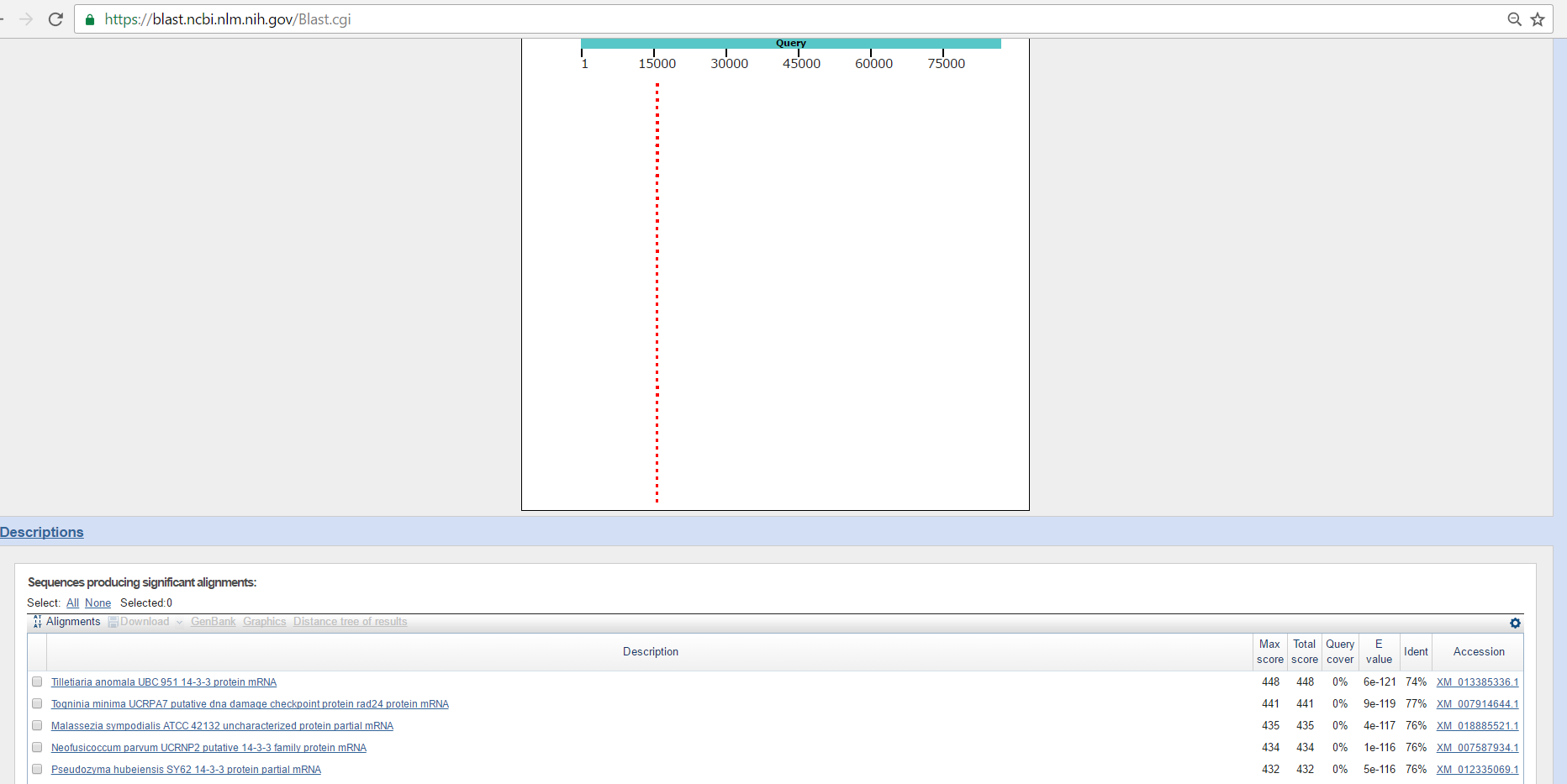
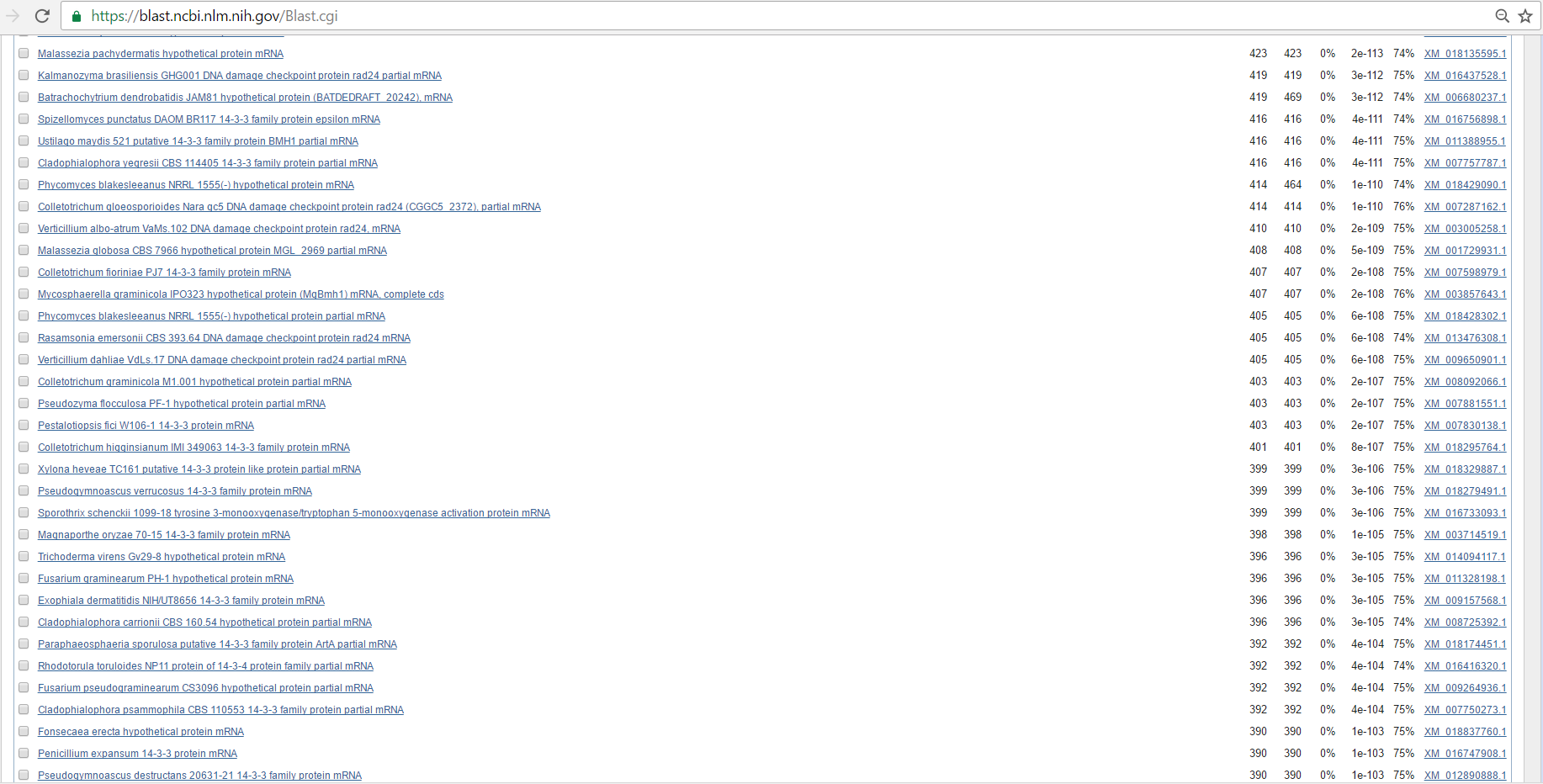
У находок хорошее E-value и неплохое Identity, так что можно утверждать, что в данном скеффолде закодирована mRNA белка 14-3-3