Упражнения на некторое команды EMBOSS
| 1.(seqret) Несколько файлов в формате fasta собрать в единый файл | ||
| Команда: seqret @list_seq.txt 1_seqret.fasta | Имена исходных последовательностей | Результат |
| 2.seqretsplit) Один файл в формате fasta с несколькими последовательностями разделить на отдельные fasta файлы | ||
| Команда: seqretsplit 1_seqret.fasta (можно еще -auto) | Исходный файл | Результат: x13141.fasta x13142.fasta x13143.fasta x13144.fasta x13145.fasta x13146.fasta |
| 3.(seqret) Из файла с хромосомой в формате .gb вырезать три кодирующих последовательности по указанным координатам "от", "до", "ориентация" и сохранить в одном fasta файле | ||
| Команда: seqret @cod_seq_3.txt 3_seqret.fasta | Файл с последовательностью хромосомы Burkholderia cenocepacia Файл с указанием координат коодирующих участков | Результат |
| 4.(transeq) Транслировать кодирующие последовательности, лежащие в одном fasta файле, в аминокислотные, используя указанную таблицу генетического кода. Результат - в одном fasta файле. | ||
| Команда: transeq x13141.fasta x13141.pep -table 0 | Исходный файл | Результат |
| 5.(transeq) Транслировать данную нуклеотидную последовательность в шести рамках. | ||
| Команда: transeq x13141.fasta x13141_5.pep -table 0 -frame 6 | Исходный файл | Результат |
| 6.(seqret) Перевести выравнивание и из fasta формате в формат .msf | ||
| Команда: seqret fasta::vyr_water_good.fasta msf::vyr_water_good.msf | Исходный файл | Результат |
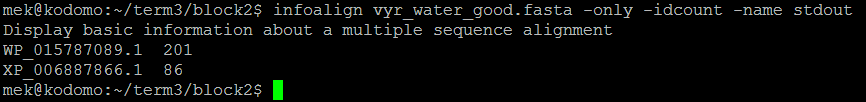
| 7.(infoalign) Выдать в выходной поток число совпадающих букв между второй последовательностью выравнивания и всеми остальными (на выходе только имя последовательности и число) | ||
| Команда: infoalign vyr_water_good.fasta -only -idcount -name stdout | Исходный файл |  |
| 8.(featcopy) Перевести аннотации особенностей в записи формата .gb в табличный формат .gff | ||
| Команда: featcopy sequence.gb sequence.gff > sequence.gff | Исходный файл | Результат |
| 9.(extractfeat) Из данного файла с хромосомой в формате .gb получить fasta файл с кодирующими последовательностями; (*) добавить в описание каждой последовательности функцию белка (из поля product) | ||
| Команда: extractfeat sequence.gb info.fasta -type CDS -describe product | Исходный файл | Результат |
| 10.(shuffle) Перемешать буквы в данной нуклеотидной последовательности; (*) проверить с помощью blastn сколько "достоверных" находок (с E-value < 0.1) найдется в нуклеотидном банке данных (запустите с порогом E = 10 - по умолчанию) | ||
| Команда: shuffle -o x13142_shuffled.fasta x13142.fasta | Исходный файл | Результат |
| 11.(cusp)Найдите частоты кодонов в данных кодирующих последовательностях | ||
| Команда: cusp info.fasta Codon_usage.cusp | Исходный файл | Результат |
| 12.(compseq) Найдите частоты динуклеотидов в данной нуклеотидной последовательности и сравните их с ожидаемыми | ||
| Команда: compseq x13143.fasta x13143.compseq -word 2 -calcfreq | Исходный файл | Результат |
| 13.(tranalign) Выровняйте кодирующие последовательности соответственно выравниванию белков - их продуктов | ||
| Команда: tranalign x1314_1-6.fasta vyr_1314_1-6.mfa vyr_x1314_1-6.fasta | Невыровненые нуклеотидные последовательности Выровненные белковые последовательности (получены с помощью transeq и Jalview) | Результат Однако возможно из-за того, что последовательности брались случайным образом, он записывает в выдачу пустой файл |
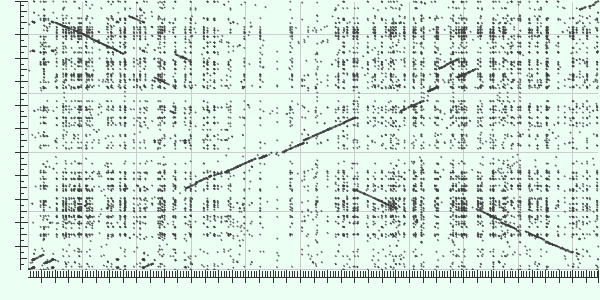
С помощью blast2seq (выравние двух или более последовательностей) с алгоритмом blastn надо было получить карту локального сходства последовательностей двух геномов родственных бактерий.
Были выбраны следующие бактерии: Streptococcus suis SC84 (последовательность) и Streptococcus suis strain NSUI060 (последовательность).
У поолученного выравнивания (прямых цепей plus strand) оказались следующие параметры:
Ниже представлена получившаяся карта локального сходства (dot matrix view)
| Синим выделены инверсии |  |
В центре и справа зеленым выделены вставки в Query(Streptococcus suis SC84) = делеции в Subject(Streptococcus suis strain NSUI060). Сверху зеленым веделены наоборот вставки в Subject = делеции в Query. | 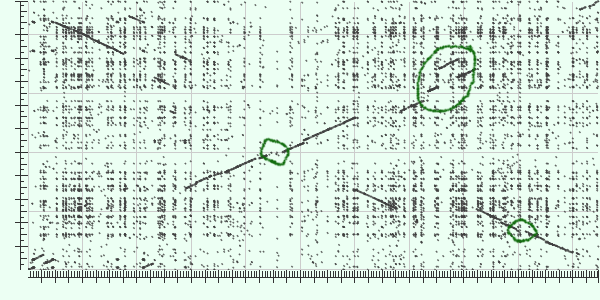 |
| Желтым выделена дуплекация в Subject (Streptococcus suis strain NSUI060) | 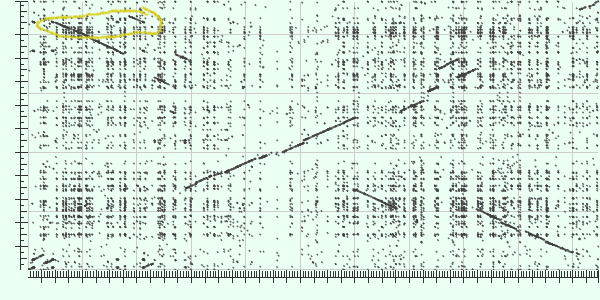 |