Для данного практикума был взять белок Potassium channel subfamily K member 10, closed state c идентификатором 4xdl (цепь D) в базе данных PDB. С помощью базы данных ORM (Orientation of Proteins in the Membrane) было получено его расположение в мембране.
Это трансмембранный белок, относящийся к альфа-спиральному политопическому классу, суперсемейству ионных каналов (VIC), семейству духпоровых каналов. Он представлен в цитоплазматической мембране человека.
Таблица 1. Характеристики мембранного расположения белка 4xdl
| Толщина гидрофобной части мембраны | 32.2 ± 0.7 Å |
| Координаты трансмембранных спиралей | Субьединица А: 1( 75- 95), 2( 185- 219), 3( 234- 260), 4( 300- 328) Субьединица В: 1( 75- 95), 2( 187- 215), 3( 238- 263), 4( 299- 327) |
| Среднее количество остатков в одной трансмембранной спирали белка | 27 a.o. |
| В какой мембране находится белок | Цитоплазматическая мембрана человека |

Рис1. Изображение структуры белка 4xdl. (р-сторона красная, n-сторона синяя)
Далее был выбран белок с трансмебранной частью в виде β-листов. Outer membrane protein A (OMPA), disordered loops (1qjp идентификатор PDB). Относится к суперсемейству OmpA-OmpF porin family (n=8,S=10), семйству OmpA. Находится во внешней мембране грамм-отрицательной бактерии Escherichia coli.
Таблица 2. Характеристики мембранного расположения белка 1qjp
| Толщина гидрофобной части мембраны | 25.4 ± 1.5 Å |
| Координаты трансмембранных спиралей | Субъединица А: 1(7-15), 2(35-45), 3(49-58), 4(76-85), 5(91-101), 6(120-131), 7(135-144), 8(160-169) |
| Среднее количество остатков в одной трансмембранной спирали белка | 9.375 а.о. |
| В какой мембране находится белок | Внешняя мембрана Escherichia coli |
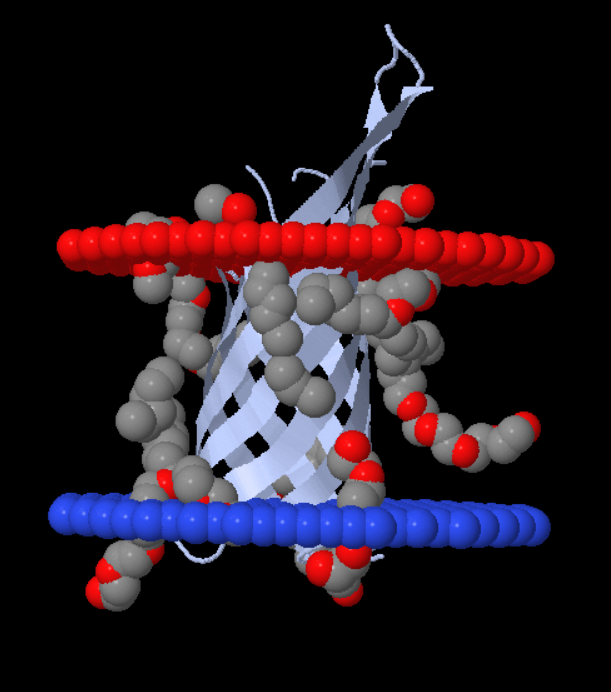
Рис2. Изображение структуры белка 1qjp. (р-сторона красная, n-сторона синяя)
Далее с помощью сервисов TMHMM и Phobius на основе 4xdl.fasta последовательности было получено предсказание мембранного расположения белка 4xdl цепи D. Полученные результаты были сравнены с расположением этого белка по базе данных ОРМ.
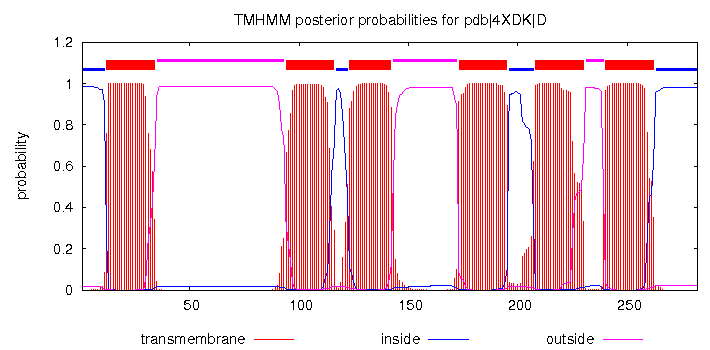
Рис3. Выдача сервиса TMHMM. По горизонтальной оси координаты а.о. в последовательности, по вертикальной оси вероятность предсказания для данного а.о. Красный цвет - трансмембранный участки, синий - участки внтури клетки, малиновый - участки снаружи клетки.
# pdb|4XDK|D Length: 282 # pdb|4XDK|D Number of predicted TMHs: 6 # pdb|4XDK|D Exp number of AAs in TMHs: 129.78283 # pdb|4XDK|D Exp number, first 60 AAs: 21.20494 # pdb|4XDK|D Total prob of N-in: 0.98489 # pdb|4XDK|D POSSIBLE N-term signal sequence pdb|4XDK|D TMHMM2.0 inside 1 11 pdb|4XDK|D TMHMM2.0 TMhelix 12 34 86 107 pdb|4XDK|D TMHMM2.0 outside 35 93 pdb|4XDK|D TMHMM2.0 TMhelix 94 116 167 189 pdb|4XDK|D TMHMM2.0 inside 117 122 pdb|4XDK|D TMHMM2.0 TMhelix 123 142 196 215 pdb|4XDK|D TMHMM2.0 outside 143 172 pdb|4XDK|D TMHMM2.0 TMhelix 173 195 246 268 pdb|4XDK|D TMHMM2.0 inside 196 207 pdb|4XDK|D TMHMM2.0 TMhelix 208 230 281 303 pdb|4XDK|D TMHMM2.0 outside 231 239 pdb|4XDK|D TMHMM2.0 TMhelix 240 262 313 335 pdb|4XDK|D TMHMM2.0 inside 263 282
Сравнивать полученное предсказание надо с координатами четырех альфа спиралей для В-субъединицы. Как мы можем видеть TMHMM нашел две лишних спирали. А для того, чтобы сравнивать координаты надо учесть, что в последовательности, которую мы подали TMHMM, основанной на pdb файле 1 аминокислотный остаток на самом деле 73. А значит ко всем координатам надо прибавить 73 (что сделано в конце табличной выдачи). Однако все равно, координаты трансмембранных участков не совпадают с аннотировавнными.
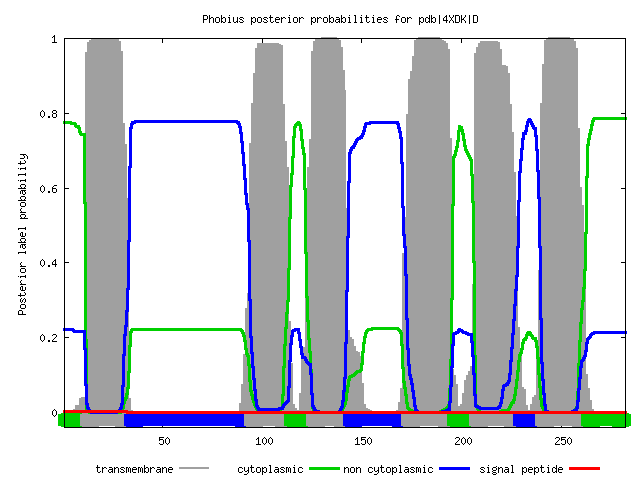
Выдача сервиса Phobius. По горизонтальной оси координаты а.о. в последовательности, по вертикальной оси вероятность предсказания для данного а.о. Серый цвет - трансмембранный участки, зеленый - участки внтури клетки, синий - участки снаружи клетки
ID pdb|4XDK|D FT TOPO_DOM 1 11 CYTOPLASMIC. FT TRANSMEM 12 33 86 106 FT TOPO_DOM 34 93 NON CYTOPLASMIC. FT TRANSMEM 94 113 167 186 FT TOPO_DOM 114 124 CYTOPLASMIC. FT TRANSMEM 125 143 198 216 FT TOPO_DOM 144 172 NON CYTOPLASMIC. FT TRANSMEM 173 195 246 268 FT TOPO_DOM 196 206 CYTOPLASMIC. FT TRANSMEM 207 228 280 301 FT TOPO_DOM 229 239 NON CYTOPLASMIC. FT TRANSMEM 240 262 313 335 FT TOPO_DOM 263 282 CYTOPLASMIC. //
Как моно видеть, предсказание Phobius польностью совпало с TMHMM по типу и количеству разных участков расположения и почти совпало по координатам. Т.е. он тоже нашел две трансмембранных спирали аннотированный в базе данныз ОРМ, и те, которые нашел, все равно оказались не те, судя по координатам..
Это можно объяснить, тем что исследуемый белок является кальциевым каналом, а для транспортировки кальция там встречаются заряженные аминокислоты, и это сильно путается и TMHMM и Phobius.
В базе данных транспортеров TCBD (Transport Classification DataBase) белок 4xdl не представлен, но представлен белок 1qjp: 1.B.6.1.1, где 1.В.6. означает принадлежность к семейству The OmpA-OmpF Porin (OOP) (октаметилпирофосфоамид октаметилпирофосфофторид порин)