Для работы был выбран фермент Acyl-CoA оксидаза с PBD ID 1w07. Разрешение структуры - 2Å.
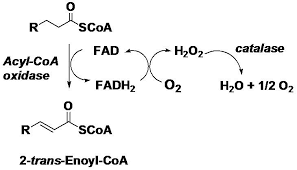
Acyl-CoA оксидаза каталиизирует окисление Acyl-CoA, имеет FAD в качестве кофактора и участвует в трех метаболических путях: метаболизм жирных кислот, синтез полиненасыщенных жирных кислот и в PPAR (pецепторы, активируемые пероксисомным пролифератором) сигнальном пути.

Рис 1. Реакция, катализируемая Acyl-CoA оксидазой
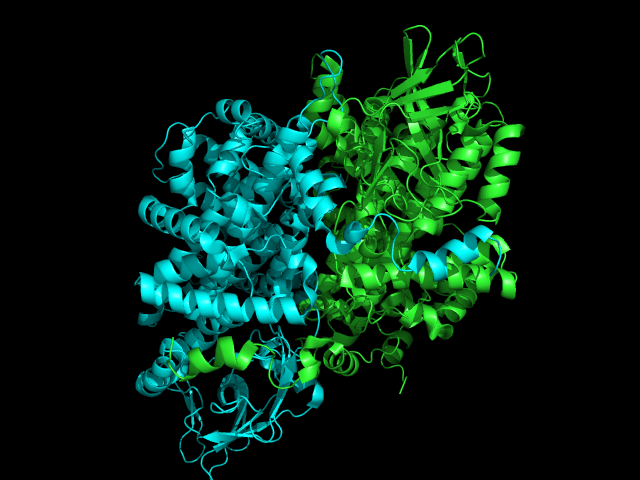
Рис 2. Структура Acyl-CoA оксидазы, окрашенная по цепям (для дальнейшей работы была взята цепь А)
Для визуализации использовались два файла: PDB file pdb1w07.ent и файл с электроной плотностью EDS map 1w07.ccp4
Для выделения основной цепи использовалась команда:
| select backbone |
Для визуализации электронной плотности использовалась команда:
| isomesh name, 1w07_map, 0.5, selected, carve=2 |
Рис 3. Электронная плотность Acyl-CoA оксидазы. σ=0.5 |
Рис 4. Электронная плотность Acyl-CoA оксидазы. σ=1.5 |
|
Рис 5. Электронная плотность Acyl-CoA оксидазы. σ=2.5 |
Рис 6. Электронная плотность Acyl-CoA оксидазы. σ=3.5 |
Для более подробного рассмотрения были выбраны три подряд идущих аминокислотных остатка: отрицательно заряженный глутамат, ароматический триптофан и алифатический валин.
|
Рис 7. Электронная плотность GLu, Trp, Val. σ=1.5 |
Рис 8. Электронная плотность GLu, Trp, Val. σ=2.0 | <
Рис 9. Электронная плотность GLu, Trp, Val. σ=2.5 |
Как можно видеть по представленным изображениям, если не делать σ очень большим, то карта электронной плотности довольно хорошо отражает структуру белка и сгущения электронной плотности соответсвуют координатам атомов.