Обзор генома бактерии Citrobacter tructae
Автор: Сучков Михаил
Аннотация
Данная работа является анализом генома бактерии Citrobacter tructae, с использованием методов программирования на языке Python и функционала электронных таблиц.
Введение
Citrobacter — это грамотрицательные палочковидные бактерии из типа Proteobacteria, семейства Enterobacteriaceae. Клетки длинные, палочковидные, обычно 1–5 мкм в длину, с жгутиками для передвижения. Citrobacter tructae была выделена из почек больной радужной форели (Oncorhynchus mykiss) [1].
В настоящем мини-обзоре рассматриваются и анализируются базовые особенности генома и протеома бактерии с помощью биоинформатических методов. Исследуются кодирующие белок последовательности, пересечения кодирующих участков генов, а также нуклеотидные последовательности псевдогенов.
Методы
Информация о геноме бактерии была взята с данного сайта: NCBI Genomes.
Для анализа данных использовались программы, написанные на языке программирования Python, и также применялись электронные таблицы Google Sheets. В них были использованы функции: ЕСЛИ, СУММ, =СЧЁТЕСЛИМН и фильтр. Ссылки на программы Python можно найти в разделе "Сопроводительные материалы".
Результаты
Описание длин белков бактерии
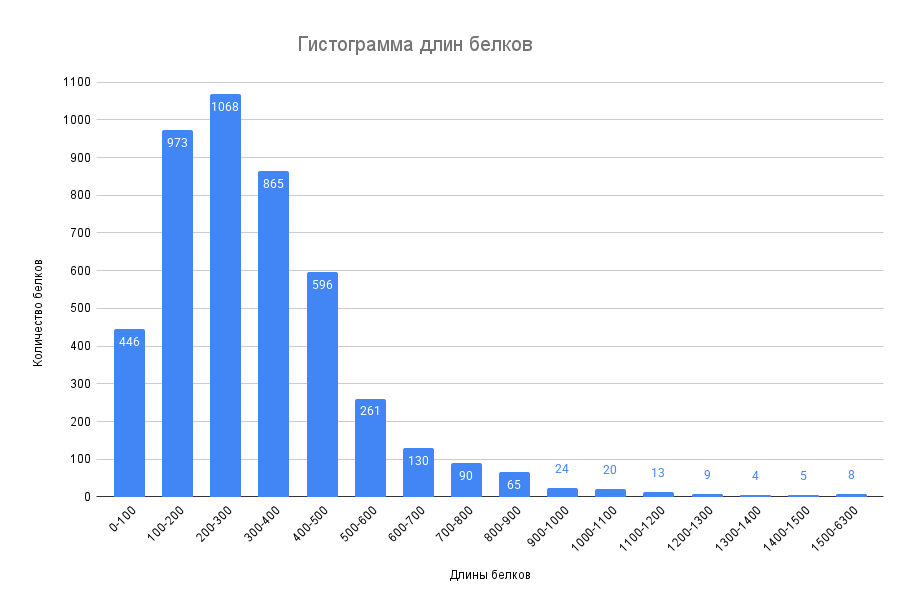
Из таблицы cds_from_genomic были получены длины белков. С помощью Google Sheets эти данные были представлены в виде гистограммы (рисунок 1).
По рисунку 1 видно, что самая частая длина белка у бактерии Citrobacter tructae имеет длину 200–300 аминокислот, причем более 90% белков имеют длину от 0 до 600 аминокислот. А всего белков 4577.
Описание пересечений генов бактерии
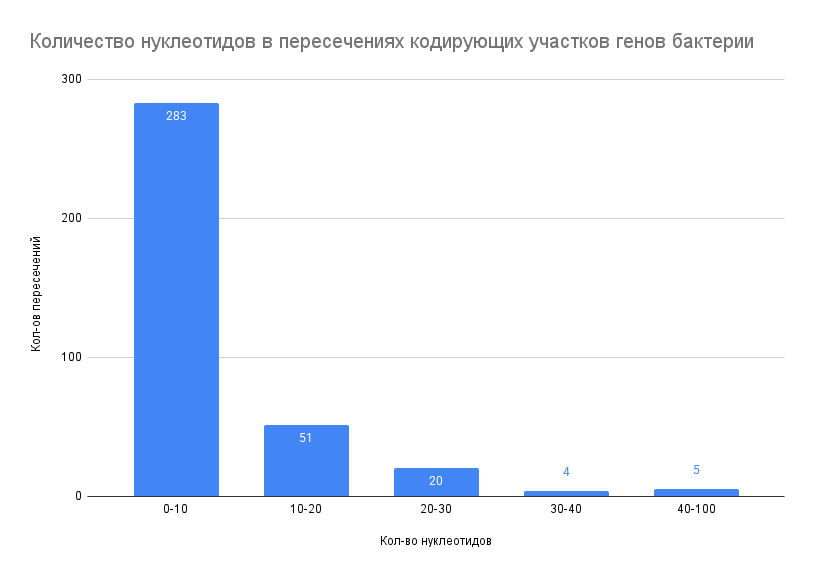
Из таблицы feature table были получены номера старт-кодонов и стоп-кодонов. При помощи Google Sheets было посчитано количество нуклеотидов в пересечениях кодирующих участков генов на положительной цепи ДНК бактерии Citrobacter tructae (рисунок 2).
По рисунку 2 видно, что самое частое количество нуклеотидов при пересечении находится в диапазоне 0–10, и чем больше нуклеотидов в пересечении, тем реже такое пересечение будет встречаться.
Анализ стоп-кодонов псевдогенов
Из таблицы cds_from_genomic с помощью программы на языке Python были получены псевдогены и проведен анализ их стоп-кодонов. Полученные данные находятся в файле stop_codons.txt и представлены в виде таблицы и гистограмм. Всего было найдено 90 псевдогенов.
По таблице 1 видно, что 40% псевдогенов утратили стоп-кодон в конце гена, а количество преждевременных стоп-кодонов больше, чем стоп-кодонов в конце гена.
| В конце гена | В остальном участке гена | |
|---|---|---|
| Стоп-кодоны | 51 | 180 |
| Не стоп-кодоны | 39 | - |
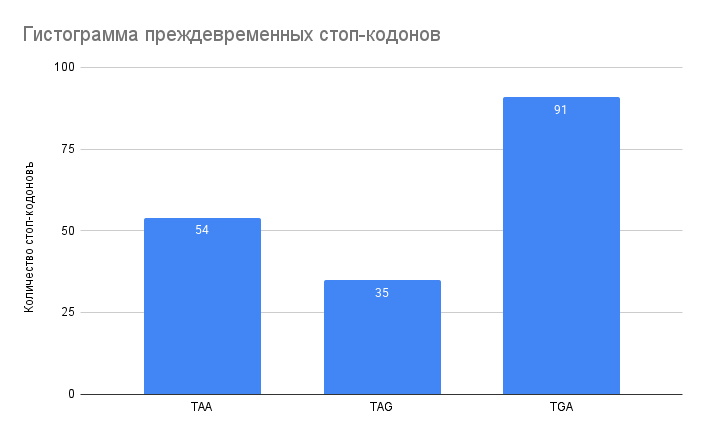
По рисунку 3 видно, что самым часто встречающимся преждевременным стоп-кодоном является TGA, а TAA и TAG встречаются намного реже.
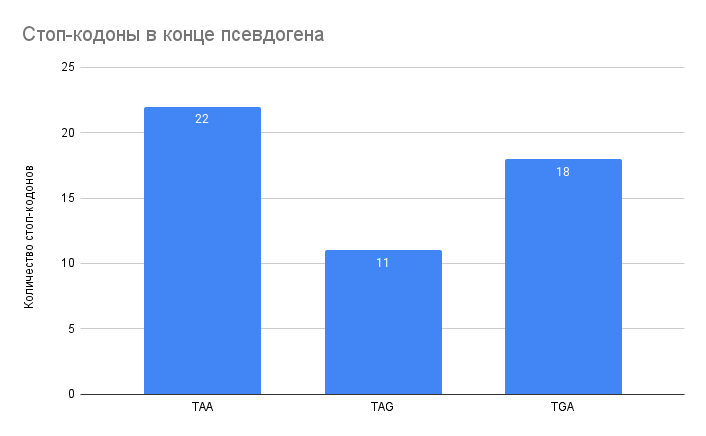
По рисунку 4 видно, что самым часто встречающимся стоп-кодоном в конце псевдогена является TAA, TGA встречается чуть реже. Это существенно отличается от показателей преждевременных стоп-кодонов.
Сопроводительные материалы
Литература
- Jung W. J. et al. Citrobacter tructae sp. nov. isolated from kidney of diseased rainbow trout (Oncorhynchus mykiss) //Microorganisms. – 2021. – Т. 9. – №. 2. – С. 275.
- Tutar Y. Pseudogenes //International Journal of Genomics. – 2012. – Т. 2012. – №. 1. – С. 424526.
- Wang X. et al. Efficient gene silencing by adenine base editor-mediated start codon mutation //Molecular Therapy. – 2020. – Т. 28. – №. 2. – С. 431-440.
Благодарности
- Сучкову Б.А. за ежедневную моральную поддержку.
- Спирину С.А. за исправления и корректировки.
- Куликову Антону за исправления и корректировки.
- Гришину Вадиму за исправления и корректировки.
- Петрушко Ивану за корректировки.
- Бритвину А.А. за исправление ошибки в коде.