Нуклеотидный BLAST
1. Поиск гена δ-субъединицы АТФ-синтазы в геноме дрожжей
1.1 Поиск и сохранение белковой последовательности
Белок был найден в файле белковых последовательностей с помощью команды:
Был получен NP_010280.1
1.2 Поиск идентификатора нуклеотидной записи
Был произведен поиск в GBFF файле с помощью команды, а далее выполнен конвейер для получения идентификатора:
Результат: LOCUS NC_001136 1531933 bp DNA linear
Идентификатор нуклеотидной записи: NC_001136.10
1.3 Получение последовательности ДНК гена
CDS последовательность гена ATP16 получена при помощи сайта NCBI Nucleotide:
Координаты CDS: 708,891-709,394
1.4 Изображение окрестности гена в геномном браузере
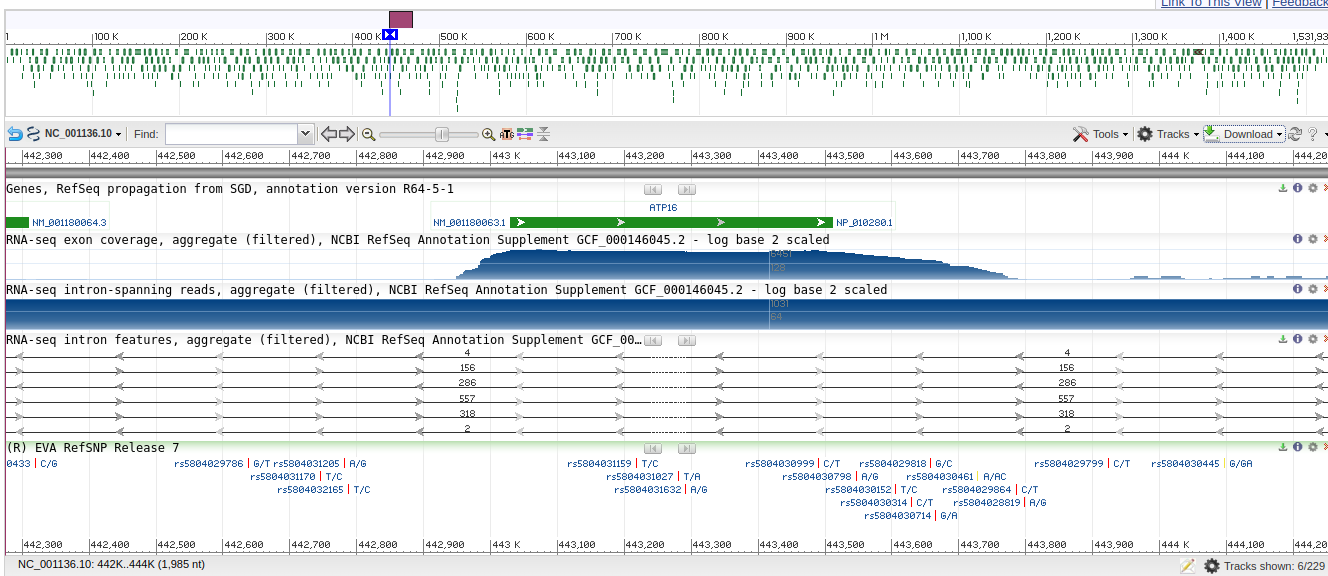
Рисунок 1. Окрестность гена ATP16 в геноме Saccharomyces cerevisiae
Идентификатор белка: NP_010280.1
Идентификатор нуклеотидной записи: NC_001136.10
Координаты CDS: 708,891-709,394
Файл с последовательностью: atp16_cds.fasta
2.Сравнение вариантов поиска гомологов BLAST
2.1 Выбор семейства
Выбрано семейство: Кошачьи (Felidae)
2.2 Параметры поиска BLAST
База данных: refseq_genomes (15 сборок геномов кошачьих)
| Метод | Параметры | Число находок | Лучший E-value |
|---|---|---|---|
| BLASTN/Megablast | word_size=16, evalue=0.05 | 4 | 0.027 |
| TBLASTN | word_size=3, evalue=0.05 | 15 | 4e-04 |
2.3 Графические результаты BLAST

Рисунок 2: Результаты BLASTN - найдено 4 коротких высокогомологичных участка
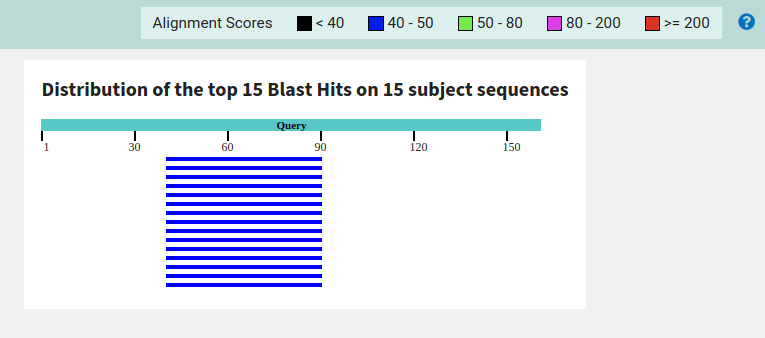
Рисунок 3: Результаты TBLASTN поиска - найдено 15 гомологов с лучшим покрытием
2.4 Анализ результатов
Ожидаемое число находок: 15 (по числу сборок геномов кошачьих в RefSeq)
Фактические результаты:
- BLASTN: 4 находки (27% от ожидаемого)
- TBLASTN: 15 находок (100% от ожидаемого)
Вывод: TBLASTN значительно эффективнее для поиска гомологов у эволюционно далеких организмов, так как использует сравнение на уровне аминокислотных последовательностей, которые являются более консервативны.
3. Поиск генов рибосомальных РНК по гомологу E. coli
3.1 Индексация генома для локального BLAST
3.2 Поиск рРНК
Для проведения поиска были использованы следующие команды:
3.3 Результаты поиска
16S рРНК: 12 находок (результат)
23S рРНК: 26 находок (результат)
3.4 Схема расположения находок в геноме
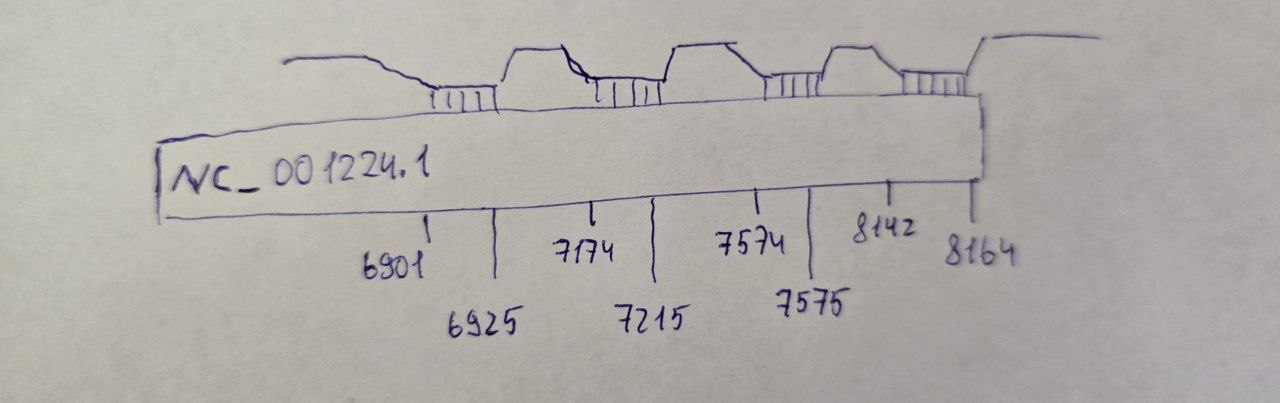
Рисунок 4. Схематичное изображение расположения гомологов прокариотических рРНК в геноме S. cerevisiae
4. Карты локального сходства геномов
4.1 Выбранные последовательности
Для анализа были выбраны плазмиды двух бактерий рода Pseudomonas
Sequence 1: NZ_CP120882.1 - плазмида бактерии
Sequence 2: NZ_CP199807.1 - плазмида бактерии
4.2 Карты локального сходства
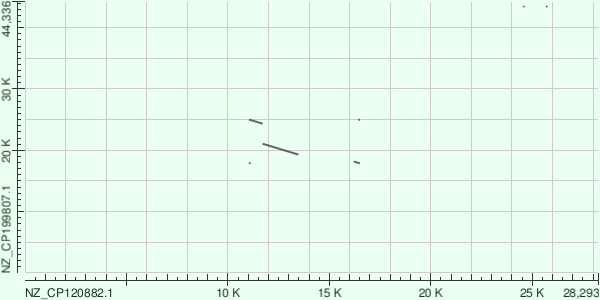
Рисунок 5.Megablast
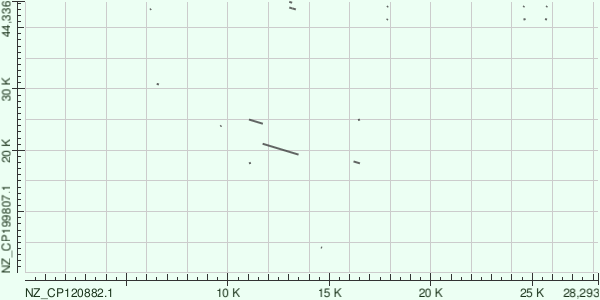
Рисунок 6.BLASTN (word_size=7, match/mismatch=1/-1)
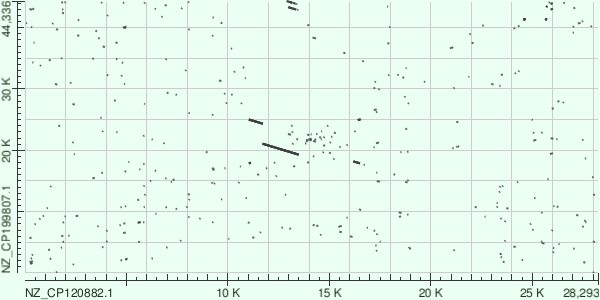
Рисунок 7.TBLASTX
4.3 Вывод
Различия между методами:
- Megablast показал только участки с высокой идентичностью
- BLASTN с чувствительными параметрами показал больше более мелких участков сходства.
- TBLASTX обнаружил те же, участки, что и предыдущие, однако показал еще больше мелких участков сходства.